Практикум 10. Сравнение геномов
Задание 1.Постройте карту сходства хромосом двух родственных бактерий
В качестве первого организма я выбрала Mycobacterium tuberculosis strain 96121 (AC NZ_CP009427.1). В качестве второго - Mycobacterium smegmatis strain INHR2 (AC NZ_CP009496.1).
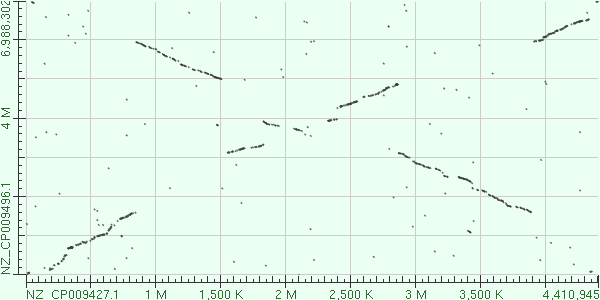
Рис.1 Карта локального сходства геномов данных бактерий
Размеры геномов:
Mycobacterium smegmatis strain INHR2 - 6988302 bp
Mycobacterium tuberculosis strain 96121 - 4410945 bp
Видно, что размеры геномов этих бактерий отличаются в полтора раза. Ожидаемо, что у M. smegmatis будет видно множество вставок.
Проанализируем карту локального сходства. Во-первых, видно, что в данных геномах присутсвует несколько крупных выравненных фрагментов. Можно утверждать, что они гомологичны, и в них сходна не только нуклеотидная последовательность генов и некодирующих участков, но и взаимное расположение элементов. Также присутствует множество более мелких находок, которые могут соответствовать эволюционным изменениям небольших участков генома. несколько крупных эволюционных событий. Их мы рассматривать не будем, а разберемся с крупными эволюционными событиями.
1. Инверсия в районе 2 Mb генома M.tuberculosis (NZ_CP009427.1)
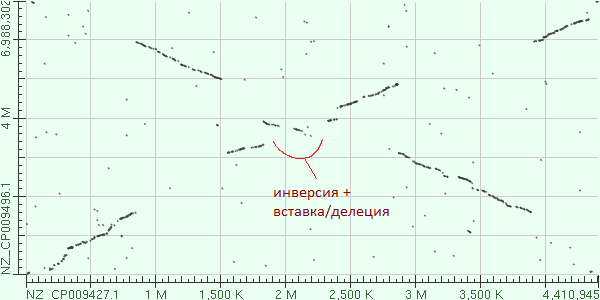
Рис.2 Инверсия + вставка (у NZ_CP009427.1) или делеция (у NZ_CP009496.1)
Выделенный фрагмент генома M.tuberculosis (NZ_CP009427.1) инвертирован у M.smegmatis (NZ_CP009496.1). Кроме того, у M.smegmatis не хватает кусочка в середине данного инвертированного фрагмента; либо у M.smegmatis произошла делеция, либо у M.tuberculosis - вставка. (Необходимо оговориться, что без дополнительных исследований, - например, построения филогенетического дерева этих и группы родственных организмов, - невозможно определить, является конкретное эволюционное событие вставкой или делецией. Что касается инверсии, тоже невозможно определить без дополнительной информации, какое состояние - как у M.tuberculosis или как у M.smegmatis - было предковым).
2. Инверсия + транслокация в районе 1-1.5 Mb и 3-4 Mb генома M.tuberculosis (NZ_CP009427.1)
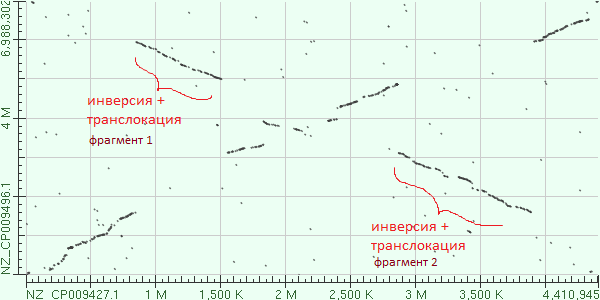
Рис.3 Два случая инверсии, сопряженной с транслокацией, в данных геномах
Два очень крупных фрагмента претерпели инверсию и транслокацию. При этом из карты видно, что фрагменты 1 и 2 поменялись местами в геноме.
3. Пример вставки/делеции - в районе 2.4 Mb генома M.tuberculosis (NZ_CP009427.1)
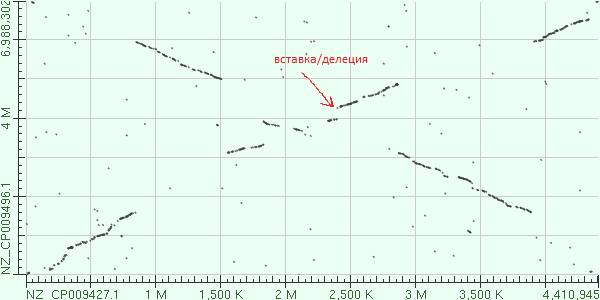
Рис.4 Вставка/делеция в районе 2.4 Mb генома M.tuberculosis
В районе 2.4 Mb генома M.tuberculosis отсутствует фрагмент длиной около 0.4 Mb, имеющийся в геноме M. smegmatis.
Подобных вставок/делеций в этих геномах довольно много, именно им соответствуют изломы и небольшие разрывы сплошных линий.
Задание 2. Опишите сходство и различие геномов близкородственных бактерий
Метод: построение нуклеотидного пангенома (NPG) с помощью пакета NPG-explorer Нуклеотидныq пангеном - этоспециальный формат для множественного выравнивания геномов, ориентированный на геномы близкородственных бактерий или архей. Материал: геномы 3-4х разных штаммов одного вида.
В качестве организмов, чьи геномы анализировали, были выбраны три бактерии: Acinetobacter baumannii strain AB031 (AC CP009256.1), Acinetobacter baumannii strain AB030 (AC CP009257.1) и Acinetobacter baumannii strain AbH12O-A2 (AC CP009534.1). Геномы выбранных бактерий я проверила на наличие достаточного количества гомологичных участков с помощью blast. Карты выравниваний свидетельствовали о хорошей гомологии.
Далее я создала файл genomes.tsv. Затем следовала последовательно инструкциям по работе с NPG-explorer.
Краткая справка по терминологии:s-блоки - стабильные (коровые) блоки, по одному фрагменту из каждого генома
h-блоки - "полустабильные" блоки - по одному фрагменту из части геномов
u-блоки - и не блоки вовсе, а уникальные последовательности из одного генома, у них нет гомологов среди всех геномов, кроме самой себя
r- блоки - блоки с повторами, по крайней мере, в одном геноме
m-блоки - минорные блоки - короткие (
Идентификатор блока r34x1201 устроен так: r - тип блока (от repeat); 34 фрагмента в блоке; 1201 позиций в выравнивании блока; иногда приходится добавлять "n1", "n2" и т.п. на конце чтобы сохранить уникальность имен pangenome/pangenome.bi содержит информацию по каждому блоку, включая информацию фрагменты каких геномов входят в блок; удобен для поиска крупных делеций/вставок (h-блоки и u-блоки) анализа блоков с повторами.
Результаты:
Excel-таблица с результатами1) Описание синтеничных участков - g-блоков - и их перестановок в геномах:
Число g-блоков (согласно данным из визуализатора) = 30.
Их порядок в каждой хромосоме для всех геномов (выравнивание) приведен в прикрепленной таблице на листе g-blocks. Выравнивание вышло слишком длинным, чтобы вставлять его картинку на сайт. Привожу фрагмент для иллюстрации того, как оно выглядит в визуализаторе:
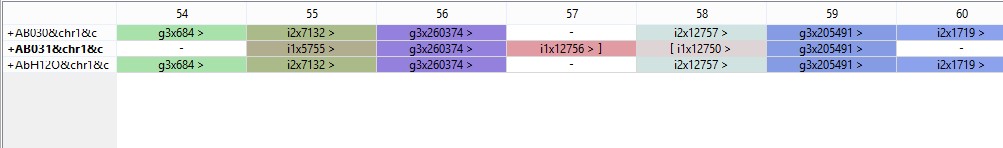
Рис.2.1 Фрагмент выравнивания g-блоков.
Несмотря на то, что я предварительно прогоняла геномы данных бактерий через blast2seq и карты выравниваний выглядели вполне приемлемо, бактерии были выбраны явно неудачно для учебных целей. Картина вышла довольно сложная.
Поскольку g-блоки представляют из себя наборы последовательно идущих стабильных блоков, их перемещения соответствуют перемещениям крупных фрагментов генома. Эти перемещения и иллюстрирует полученное выравнивание. По результатам работы NPGE видно, что в процессе видообразования геномы этих трех бактерий претерпевали многочисленные так называемые крупные эволюционные события: инверсии, делеции, вставки и др.
Хочу дополнительно привести карты локального сходства blast2seq, чтобы проиллюстрировать это.
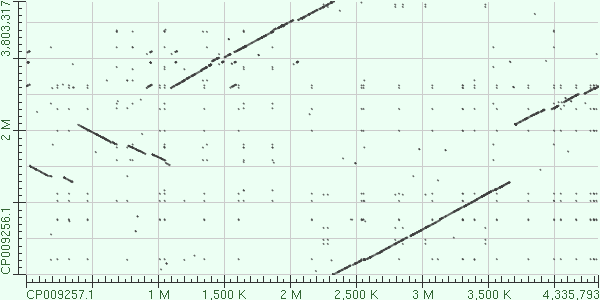
Рис.2.2 Карта локального сходства геномов Acinetobacter baumannii strain AB031 (AC CP009256.1)и A. baumannii strain AB030 (AC CP009257.1)
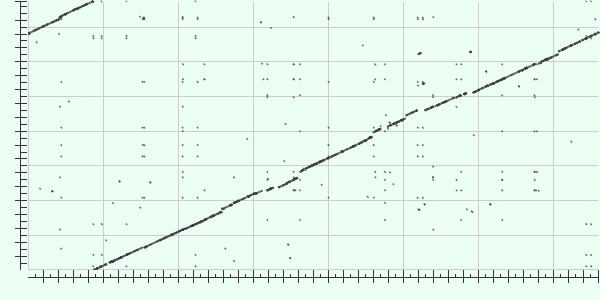
Рис.2.3 Карта локального сходства геномов A. baumannii strain AB031 (AC CP009256.1) и A. baumannii strain AbH12O-A2 (AC CP009534.1)
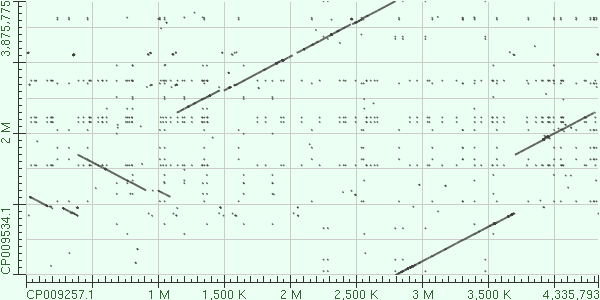
Рис.2.4 Карта локального сходства геномов A. baumannii strain AB030 (AC CP009257.1) и A. baumannii strain AbH12O-A2 (AC CP009534.1)
Обсуждение расположения g-блоков по результатам работы NPGE.
Всего 9 из 30 g-блоков занимают одинаковое положение во всех трех геномах (графа conservative в файле global-blocks/blocks.blocks). Остальные расположены по-разному. Например, некоторые блоки расположены в разных участках генома; или же конкретный g-блок занимает одно и то же место в геномах, но у одной из бактерий он инвертирован. Такие случаи видны из данных файла global-blocks/blocks.blocks.
Например:
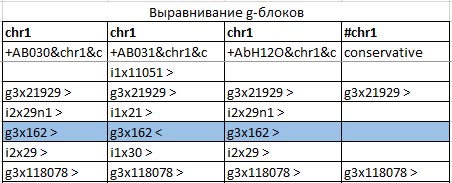
Рис.2.5 Инвертированное расположение g-блока (выделен синим) у одной из бактерий - A. baumannii strain AbH12O-A2. На это указывает галочка, смотрящая в другую сторону.
2) Описание ядра геномов - s-блоков:
(Данные взяты из файла pangenome.bi и pangenome.info:)
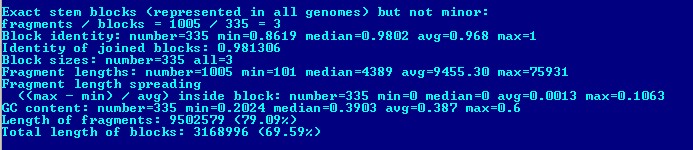
Рис.2.6 Фрагмент файла pangenome.info, посвященный s-блокам
Число s-блоков = 335
Их суммарная длина = 3168996 (69.59%)
Процент от длины генома в среднем - в файле pangenome.info не нашла, посчитала вручную: относительная суммарная длина s-блоков / кол-во s-блоков = 69.59%/335 = 0.2%.
Сходство геномов (процент консервативных позиций в объединенном выравнивании s-блоков) = 0.981306
3) Описание повторов - на 1-2 примерах r-блоков:
Для начала приведу несколько примеров r-блоков. Синтаксис таков: в идентификаторе блока r34x1201 r - тип блока (от repeat), в блоке 34 фрагмента, 1201 позиций в выравнивании блока.
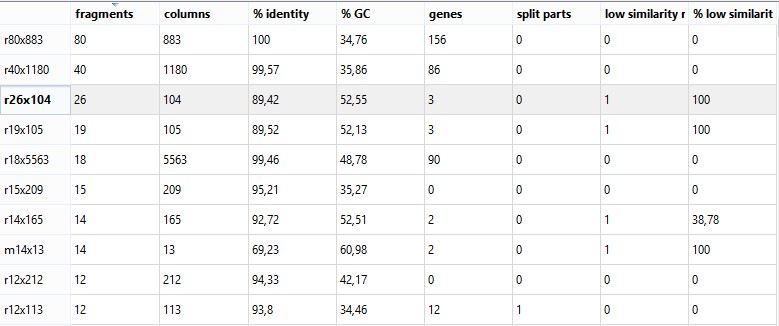
Рис.2.7 Некоторые r-блоки, найденные программой NPGE. Визуализация с помощью qnpge.
Опишу подробнее блок r26x104. Он выделен серой полосой на рис.2.7. Этот блок содержит в совокупности фрагменты 3 генов (по одному на каждую бактерию) - это CDS натрий-протонового антипортера (IX88_16655_IX88_16655 sodium:proton antiporter (AB031), 5706 bp <). При этом части этого гена встречаются также в других повторах (например, в r19x105, рис. 2.7).
Визуализатор предоставляет возможность посмотреть на выравнивание последовательностей, составляющих данный блок:
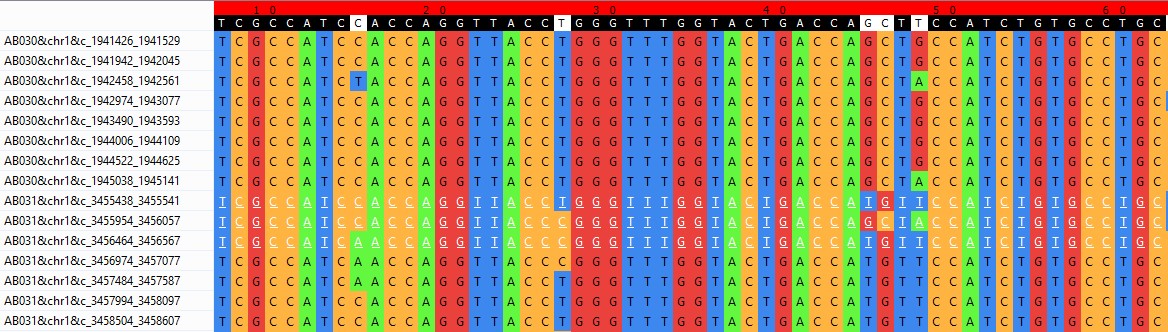
Рис.2.8 Выравнивание последовательностей блока r26x104.
Опишу также самый распространенный в этих геномах повтор - блок r80x833.

Рис.2.9 Характеристики блока r80x833.
Этот блок встречается в совокупности 80 раз и имеет большую протяженность последовательностей - около 830 п.н. При этом, что интересно, подавляющая часть повторов блока, а именно 76 из 80, принадлежат A. baumannii strain AB030, только 4 повтора - A. baumannii strain AbH12O-A2 (у A.baumannii strain AB031 нет ни одной такой последовательности). Большая часть последовательностей этого блока - кодирующие, и гены самые разные. У A. baumannii strain AB030 в повторах чаще всего встречается ген транспозазы - фермента вирусного происхождения, связанного с жизненным циклом некоторых вирусов, и, как следствие, со следами жизнедеятельности вирусов в геномах клеточных организмов. Такими следами являются некоторые повторы и мобильные элементы. Транспозаза способна связывать фрагменты ДНК и встраивать их в геном. Также у AB030 присутствуют другие гены, например, multidrug ABC transporter ATP-binding protein, homocysteine methyltransferase, несколько разных генов ионных каналов и других мембранных и регуляторных белков. Думаю, тот факт, что у A. baumannii strain AB030 ген транспозазы является частью данного повтора (r80x833), свидетельствует о вирусном происхождении этих последовательностей. Другие гены, возможно, были захвачены в процессе вырезания и перемещения мобильного элемента, которым, похоже, является этот повтор. У AbH12O-A2 одна последовательность некодирующая, три другие представляют из себя гены tRNA-Val, universal stress protein и гипотетического белка. Транспозазы нет.
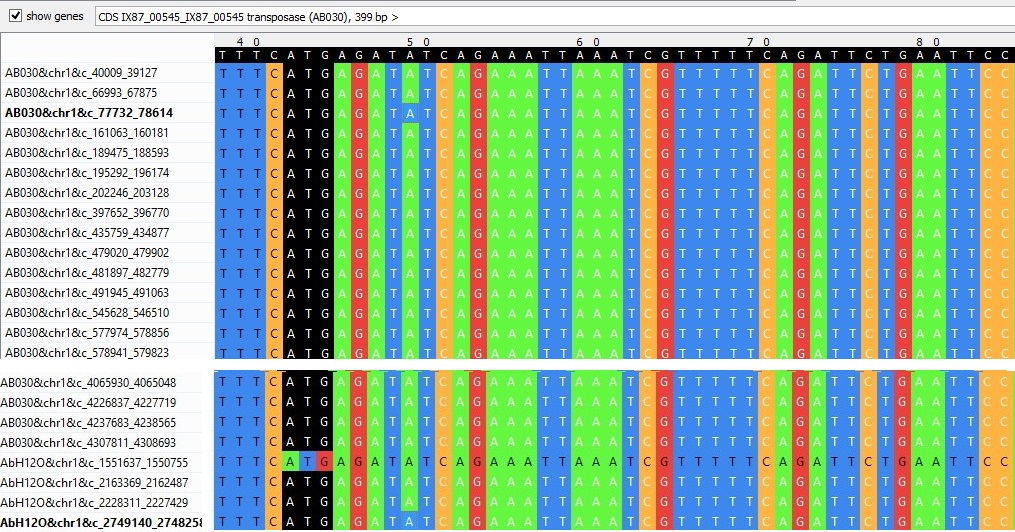
Рис.2.10 Выравнивание последовательностей блока r80x833.
4) Описание крупных делеций на примерах h-блоков
H-блоки - это "полустабильные" блоки, включающие по одному фрагменту из части геномов. То есть у какого-то из организмов данная последовательность отсутствует.
Ниже представлены три самых длинных h-блока:
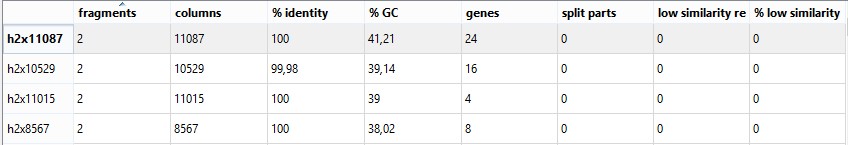
Рис.2.11 Три самых длинных h-блока
Они соответствуют трем крупным делециям. При этом все три данных блока отсутствуют у штамма AB031.
Приведу для примера иллюстрации структуры блока h2x11087:
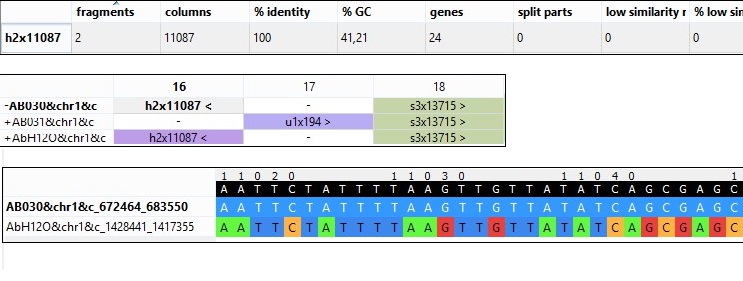
Рис.2.12 Описание блока h2x11087
Описание уникальных последовательностей (u-блоки)
Требовалось аннотировать несколько уникальных последовательностей с помощью blastn и определить, являются ли они результатом горизонтального переноса из бактерий другого вида.
В выдаче NPGE присутствует более десятка крупных уникальных фрагментов (длиной около 10 kb), а также большое количество более мелких.
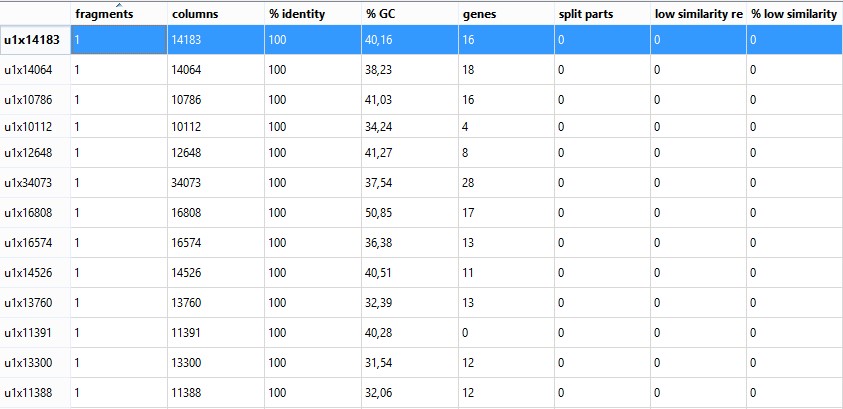
Рис.2.13 Некоторые u-блоки
Например, я нашла у штамма AB030 блок u1x697, содержащий 1 ген, а именно, phage tail protein. Ген фагового белка в геноме бактерии явно является результатом переноса генов (из этого самого фага, вероятно).
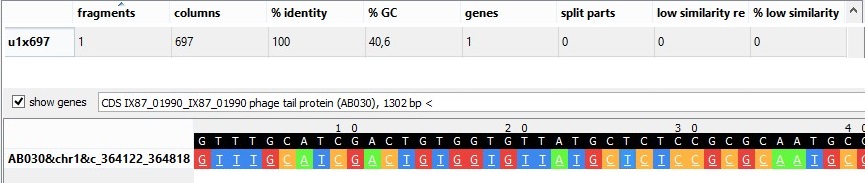
Рис.2.14 Блок u1x697
Выдача nblast содержит только 3 достоверные находки, все - в штаммах бактерий того же вида. Возможно, эти три штамма (см. рис. 2.15) являются монофилетической группой, и их общий предок приобрел данную последовательность после отхождения от общего ствола, но до разделения штаммов. Довольно низкое покрытие последовательности в находках может означать потерю части гена у двух штаммов. А может, предки этих штаммов просто болели одним и тем же фагом, уже после расхождения от общего предка.
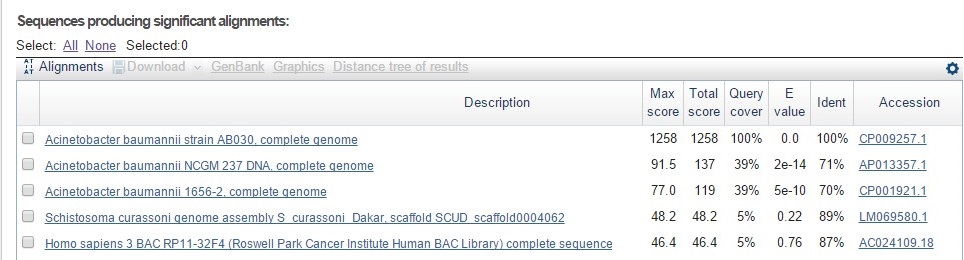
Рис.2.15 Выдача blast по запросу последовательности из блока u1x697
Еще один интересный u-блок - u1x1189.
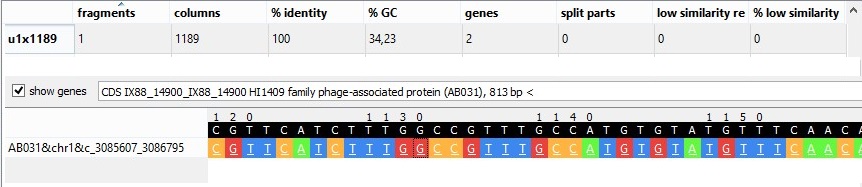
Рис.2.16 Блок u1x1189
Он содержит сразу два гена, оба вирусные: HI1409 family phage-associated protein и phage F-like protein. Выдача blast показала примерно такие же результаты, как и с предыдущим блоком: часть последовательности есть у нескольких других штаммов того же вида (покрытие небольшое, около 17%, см. рис. 2.17), а также у холерного вибриона. Логично предположить, что эти два гена были приобретены путем горизонтального переноса от фага.
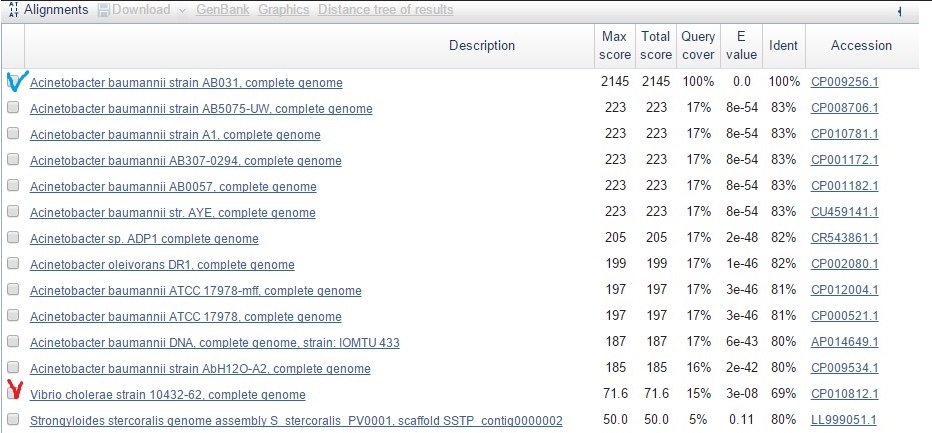
Рис.2.17 Выдача blast по блоку u1x1189
Примеры расхождений между аннотациями генов из одного блока
1. В блоке s3x23809 различаются названия одного комплекта выравненных генов.
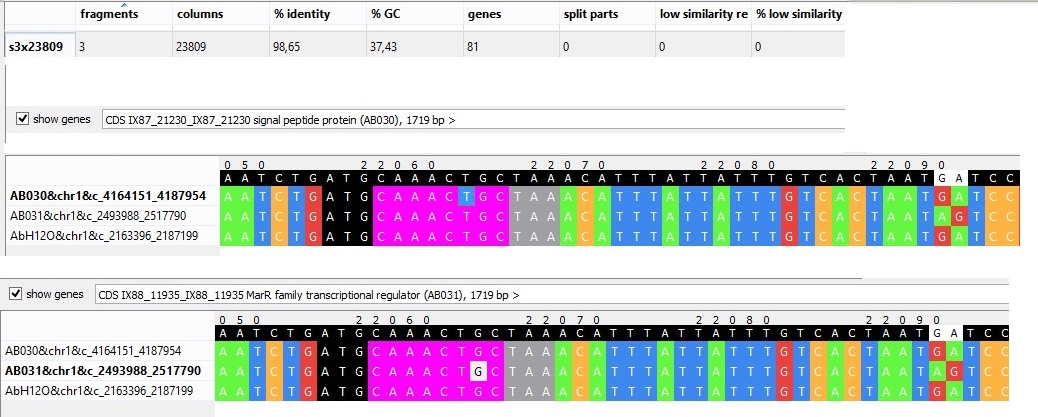
Рис.2.18 Гомологичные гены с разными названиями из блока s3x23809
У двух штаммов, AB030 и AbH12O-A2, данная последовательность аннотирована как ген, кодирующий signal peptide protein. У третьего штамма, AB031, аннотирован MarR family transcriptional regulator. Судя по выравниванию, данные гены очевидно гомологичны, но ортологичны ли - непонятно. В записях геномов штаммов AB030 и AB031 из GenBank в аннотациях рассматриваемых генов стоит пометка, что они аннотированы автоматически на основании гомологии (см. рис. 2.19). Можно ли верить в данном случае автоматической аннотации и утверждать, что продукт экспрессии гена в трех штаммах бактерий выполняет разные функции (то есть гены не ортологичны и имеют право называться по-разному)? Вообще говоря, нет. До тех пор, пока наличие и функционирование этого продукта в клетке не подтверждено экспериментально, сомнительно даже его существование.
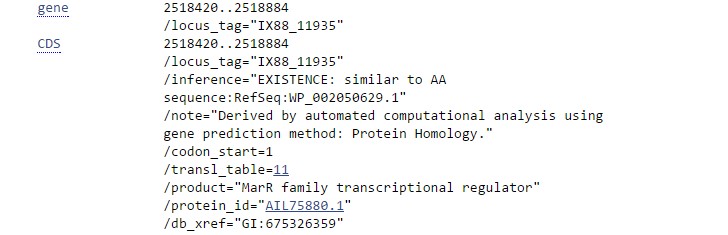
Рис.2.18 Аннотация рассматриваемого гена из записи генома A. baumannii штамма AB031 из GenBank
Длина генов у трех штаммов одинакова и равна приблизительно 1710 п.н., при этом я насчитала 29 нуклеотидных замен в MarR family transcriptional regulator относительно signal peptide protein, то есть идентичность = 98,3% - весьма высокая. В целом, думаю, нельзя с уверенностью сказать, ортологичны ли рассматриваемые гены.
2. Еще один случай расхождения аннотации генов - в блоке s3x50610.
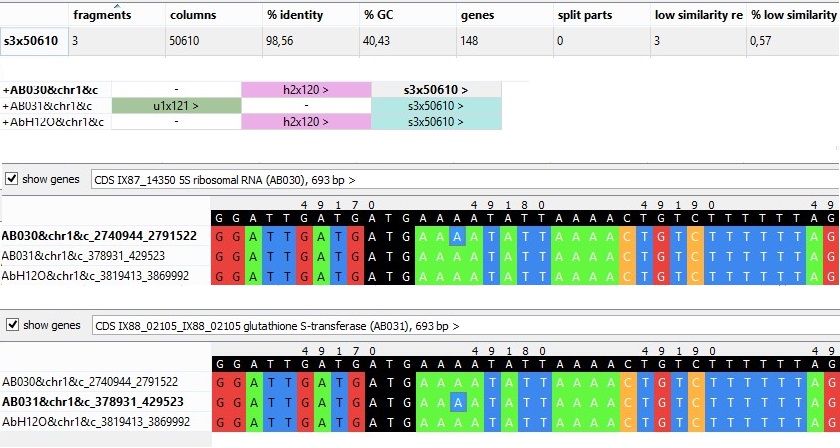
Рис.2.19 Выровненные гены с разными названиями из блока s3x50610
В большом блоке s3x50610, содержащем в общей сложности 1148 генов, я наткнулась на выровненные гены с разными аннотациями. В штамме AB031 это глутатион-S-трансфераза, а в штаммах AB030 и AbH12O-A2 - 5S ribosomal RNA. Что само по себе странно. В выравнивании 7 однонуклеотидных замен на 693 п.н. длины, то есть последовательности явно гомологичны. Я попробовала выяснить, что же это все-таки за ген и как он был аннотирован в данных штаммах.
У штамма AB030 ген был аннотирован автоматически c помощью алгоритма cmsearch, который используется для поиска РНК (см. рис. 2.20). То есть полностью доверять такой аннотации нельзя.
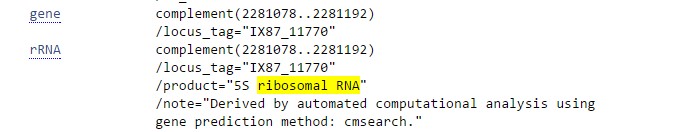
Рис.2.20 Аннотация рассматриваемого гена из записи генома A. baumannii штамма AB030 из GenBank
Далее я попробовала выяснить, действительно ли данная последовательность из AB030 и AbH12O-A2 похожа на 5S-рРНК. Вообще рРНК отличаются высокой консервативностью у разных организмов, по ним часто строят филогенетические деревья. Поэтому можно было ожидать, что если исследуемая последовательность и впрямь рРНК, то легко можно будет это доказать с помощью blast (должно быть много находок хотя бы из протеобактерий,группы бактерий, к которым относится A. baumannii) или путем сравнения с 5S-рРНК какой-нибудь, опять же, протеобактерии.
Поиск данной последовательности из AB030 в blast выдал около 30 достоверных находок, все из A. baumannii. Это указывает на то, что найденная последовательность не рРНК - маловероятно, чтобы рРНК наших бактерий имела так мало общего с рРНК других организмов. Когда я специально нашла в Gene NCBI последовательность рРНК другого вида того же рода, Acinetobacter baylyi, и запустила blast. Было найдено множество результатов из разных бактерий, что подтверждает выдвинутые предположения. Потом, длина нашей последовательности - 693 п.н., а 5S-рРНК - обычно около 120 п.н. То есть все указывает на то, что наша последовательность не 5S-рРНК и аннотирована неверно. На всякий случай я получила выравнивание данной последовательности из A. baumannii AB030 с 5S-рРНК из Acinetobacter baylyi. В последовательностях есть схожие участки, разделенные длиными гэпами в 5S-рРНК из Acinetobacter baylyi. Насколько мне известно, сплайсинг у эубактерий встречается крайне редко, так что не думаю, что исследуемая последовательность имеет прямое отношение к 5S-рРНК. Скорее всего, с аннотацией произошла какая-то ошибка.
Теперь разберемся с последовательностью из AB031, аннотированной как глутатион-S-трансфераза.
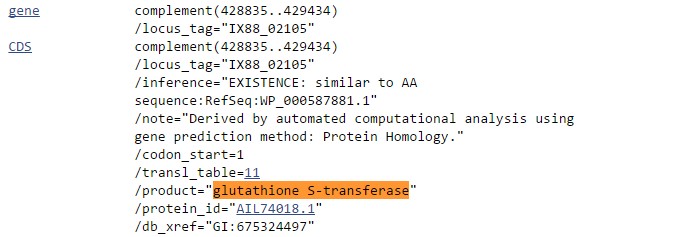
Рис.2.20 Аннотация рассматриваемого гена из записи генома A. baumannii штамма AB031 из GenBank
Оказывается, существование белкового продукта этого гена подтверждено - " аминокислотная последовательность похожа на RefSeq:WP_000587881.1", и даже есть protein_id = AIL74018.1.
Поскольку последовательности из AB031 и AB030 очень похожи (identity = 0.99), а из AB030 и AbH12O-A2 полностью идентичны, можно заключить, что с высокой вероятностью при аннотировании данных последовательностей из AB030 и AbH12O-A2 произошла ошибка, и на самом деле это гены глутатион-S-трансферазы.