Практикум 11. Чтение последовательностей по Сэнгеру
Задание 1. Аннотируйте последовательность и сравните с аннотацией генов в записи GenBank
Я работала с плазмидой pABIR бактерии Acinetobacter baumannii, предположительно отвечающей за патогенность и множественную лекарственную резистентность этого организма. Размер плазмиды составляет 29823 п.н.
Таксономия бактерии: cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales; Moraxellaceae; Acinetobacter; Acinetobacter calcoaceticus/baumannii complex; Acinetobacter baumannii
Идентификаторы данной плазмиды в БД:
NCBI Reference Sequence: NC_010481.1
GenBank: EU294228.1
Я работала с Acinetobacter baumannii в предыдущем практикуме, и она показалась мне удобным объектом исследования. Дело в том, что эта бактерия важна в медицинском плане, поскольку является распространенным возбудителем внутрибольничных инфекций и часто имеет полирезистентность к лекарствам. Поэтому отсеквенировано множество геномов штаммов этого вида, и наличествует большая информационная база для разного рода биоинформатических изысканий.
Ход работы:
Следуя инструкциям, с помощью RAST я получила таблицу аннотаций генов. Исходная таблица аннотаций из RAST
Также я скачала по ссылке со страницы плазмиды в GenBank уже существующую таблицу аннотаций генов ( из NCBI Gene). Исходная таблица аннотаций из NCBI Gene
По каким-то не вполне ясным причинам в этой таблице присутствуют три случая повторного аннотирования одной и той же последовательности (кординаты их старт- и стоп-кодонов совпадают, различаются aliases).

Рис.1 Два случая повторного аннотирования одной и той же последовательности в таблице из NCBI Gene. Повторы выделены красным.
Далее я сравнила полученные аннотации. Результаты представлены в следующей таблице .
Результаты сравнения таблиц:
Таблица 1. Результаты сравнения аннотаций RAST и GenBank
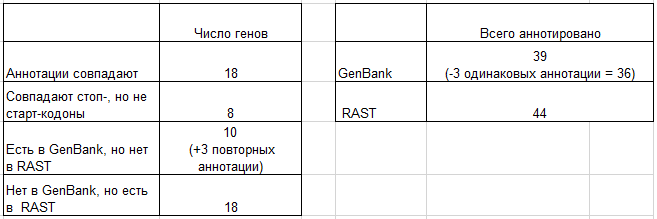
RAST не нашел 10 генов, аннотированых в GenBank, в том числе несколько генов, для которых известны функции продуктов: два гена септиколизина(septicolysin), гены macrolide 2'-phosphotransferase и macrolide efflux protein. Также в таблице из GB прис утствуют 5 гипотетических белков, не найденные RAST. 18 находок RAST, отсутствующие в GB, в основном аннотированы как гипотетические белки (в одном случае аннотирован мобильный элемент).
Некоторые случаи несовпадения аннотаций:
1. Ген бета-лактамазы: несовпадение старт-кодонов в аннотациях RAST и GB

Рис.1 Аннотации данного гена в RAST и GB
При ближайшем рассмотрении записи плазмиды в GB оказалось, что приведенные аминокислотные последовательности белкового продукта совпадают, а старт-кодоны различаются за счет того, что в GB в составе гена указана 5'-некодирующая область (промотор и др.).
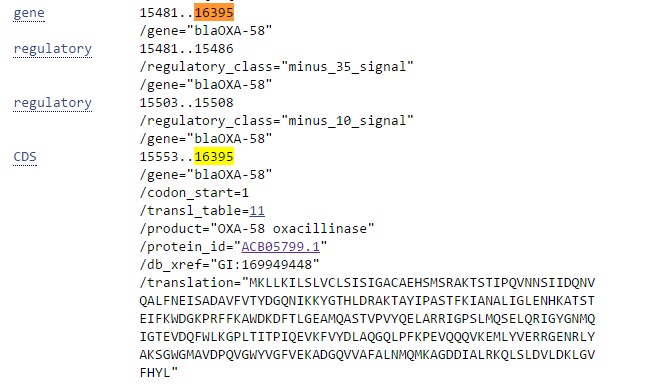
Рис.2 Аннотации данного гена в записи плазмиды в GB
Интересно, что когда я попробовала поискать данную аминокислотную последовательность в BLASTp, среди лучших находок у того же вида были такие, которые начинались с более раннего старт-кодона, хотя RAST и GB солидарны насчет старт-кодона этого гена в данной плазмиде.
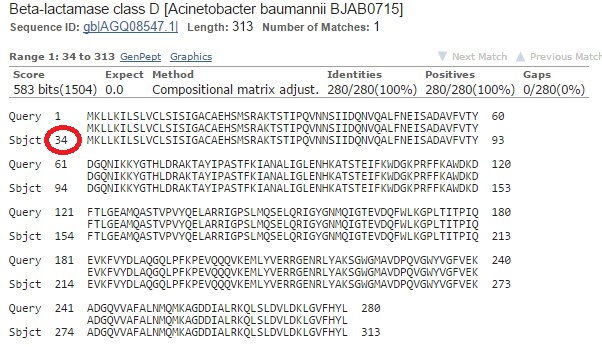
Рис.3 Находки BLASTp по запросу данной аминокислотной последовательности гена бета-лактамазы
Вообще с использованием blast для нахождения более вероятного старт-кодона возникли проблемы. Оказалось, что большая часть находок по генам данной плазмиды аннотирована автоматически, и нет причин доверять им больше, чем аннотациям RAST и GenBank. В случае расхождения аннотаций по старт-кодонам я пробовала использовать blastp, выполняя поиск по аминокислотным последовательностям. Обычно blastp выдавал множество находок у того же вида бактерии, при этом среди этих находок были соответствующие обоим вариантам старт-кодонов. И все они были аннотированы автоматически.
Потенциальным выходом из положения является использование поиска blastp против БД SwissProt и сравнение последовательностей белков с теми их гомологами, существование которых подтверждено экспериментально на уровне белка или хотя бы транскрипта. К сожалению, для следующих двух изучаемых генов в SwP не нашлось достаточно похожих гомологов, по которым можно было бы судить о старт-кодонах белков-предметов исследования.
2. Ген транспортера lysE

Рис.4 Аннотации данного гена в RAST и GB
По аннотации RAST белок длиннее на 4 аминокислотных остатка и последовательность гена начинается с другого старт-кодона (отличного по последовательности).

Рис.5 Последовательности белкового продукта данного гена в RAST и GB
Вначале я запустила blastp по аминокислотной последовательности из RAST. Результаты:
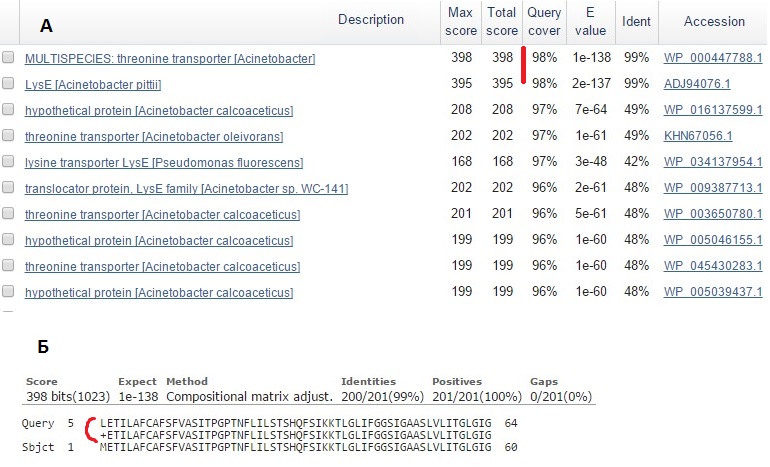
Рис.6 Результаты работы blastp по запросу аминокислотной последовательности из RAST продукта данного гена. А. Первые несколько находок. Сортировка по query cover. Б.Часть лучшей находки.
Ни одна из находок не имела значения query cover = 100% (рис. 6А) и не начиналась аналогично последовательности из RAST. Все хорошие находки начинались так же, как последовательность из записи GB (рис. 6Б).
Затем я запустила blastp против базы SwissProt. К сожалению, ни одной достоверной находки не обнаружилось.
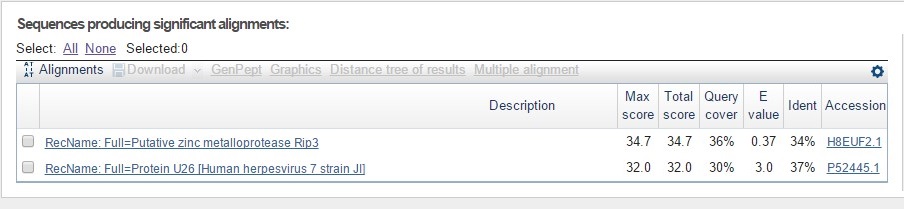
Рис.6.1 Результаты работы blastp против SwissProt
Остается судить только по результатам blast по всем БД. Можно, пожалуй, заключить, что аннотация GenBank правильна с большей вероятностью, раз уж все хорошие находки начинаются так же, как и аннотация из GB.
3. Расхождение аннотаций некоего предполагаемого гена белка (с координатами в аннотации GenBank 18914..19243)
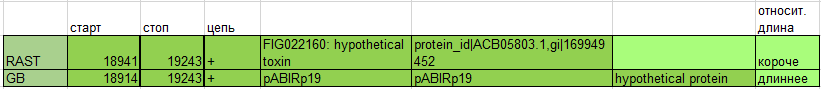
Рис.8 Аннотации выбранного гена в RAST и GB
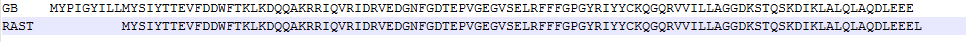
Рис.9 Последовательности белкового продукта данного гена в RAST и GB
Последовательность из GB длиннее, при этом кодирующая последовательность начинается с первого нуклеотида аннотированной.
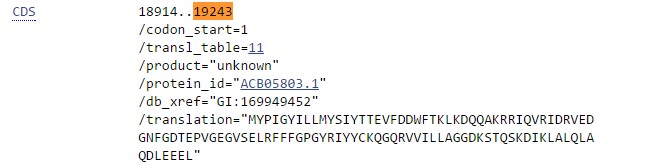
Рис.10 Запись данного предполагаемого гена в GenBank
Я запустила blastp по более длинной последовательности - из GenBank. Только одна находка соответствовала полной последовательности, подавляющее же большинство начиналось так же, как и последовательность из RAST.
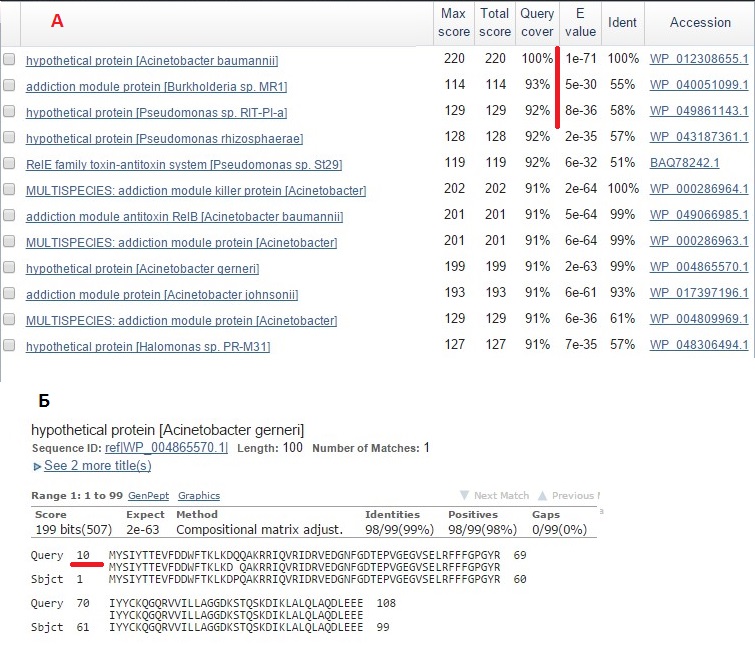
Рис.11 Результаты работы blastp по запросу аминокислотной последовательности из GenBank данного предполагаемого гена. А. Первые несколько находок. Сортировка по query cover. Б.Часть типичной хорошей находки.
Потом я запустила blastp против SwissProt по той же самой более длинной последовательности из GenBank. Результат:
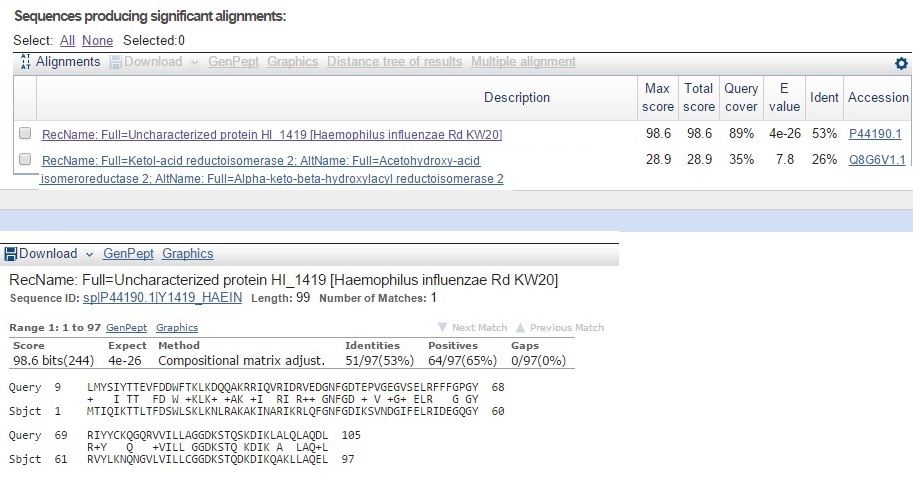
Рис.11.1 Результаты работы blastp против SwissProt
Видно, что было найдено всего две последовательности.Лучшая из них содержит участок интереса, но, во-первых, аннотирована не экспериментально, и, во-вторых,явно имеет слишком малое сходство с искомой последовательностью в искомом участке. Она начинается отличным образом и от аннотации RAST (на 1 а.о. раньше), и от аннотации GB (на 8 а.о. позже), однако, очевидно, сходство с последовательностью из RAST существенно выше.
Учитывая находки blastp по всей области поиска, можно заключить, что аннотация RAST правильна с большей вероятностью.