Практикум 13. Задача: найти и описать полиморфизмы у пациента
Дано:
1. Чтения экзома, картирующиеся на участок хромосомы человека - chr9_2.fastq
Ход работы:
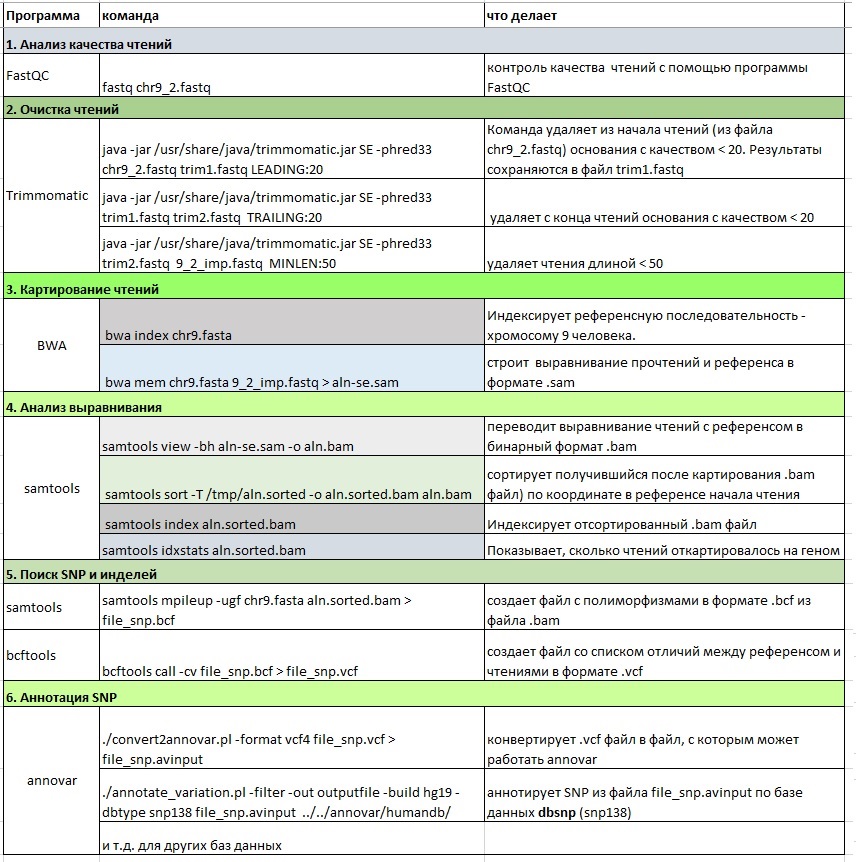
Рис.1 Программы и команды, использовавшиеся в каждом шаге.
Отчет по использованным командам в виде таблицы продублирован в прикрепленном файле
1. Анализ качества чтений с помощью программы FastQC.
пользовалась программой, установленной на kodomo. Запрос: fastq chr9_2.fastq . Результат - zip-архив, в котором содержался помимо всего прочего html-файл с отчетом.
2. Очистка чтений с помощью программы Trimmomatic.
Требовалось отрезать с конца каждого чтения нуклеотиды с качеством ниже 20 и оставить только чтения длиной не меньше 50 нуклеотидов. В чтении, с которым я работала, адаптеры уже были удалены.
Что сделано:

Рис.2 Команды для Trimmomatic
Итоговый файл trim3.fastq я переименовала в 9_2_imp.fastq .В нем содержатся чтения с качеством оснований > 20 и длиной > 50.
Для проверки результатов я посмотрела, как изменилась оценка качества чтений с помощью программы FastQC.
Результаты:
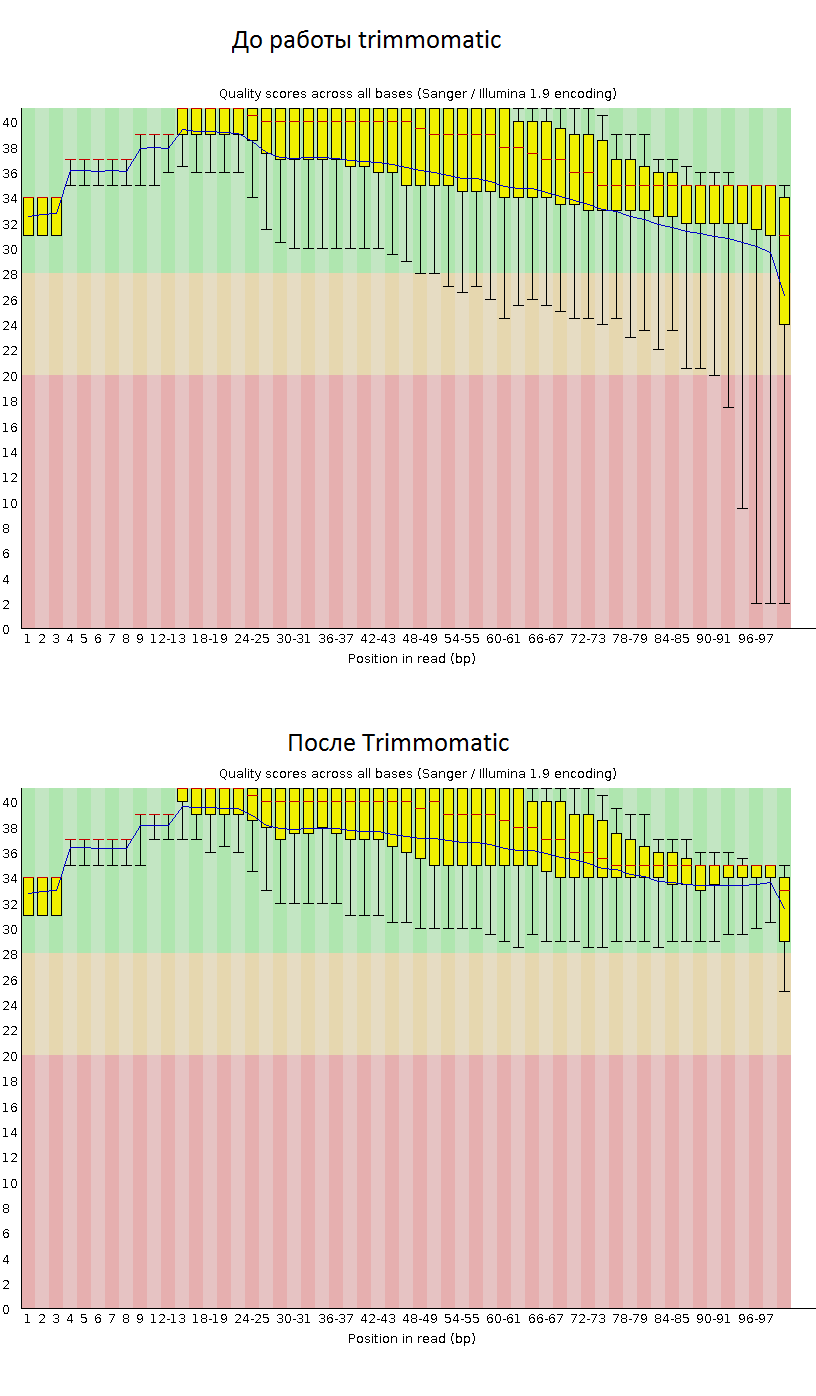
Рис.3 Изображение FastQC "Per base sequence quality" до и после чистки программой Trimmomatic
Число чтений до чистки: 2206
Число чтений после чистки: 2141
Программа работала таким образом, что отрезались "некачественные" основания с концов, а затем удалялись те риды, которые в результате такого обрезания становились слишком короткими ( <50 п.н.). Судя по отчету FastQC и выдаче программы Trimmomatic, удалилось, во-первых, небольшое число очень плохих по всей длине ридов, во-вторых, те риды, у которых были слишком длинные "плохие" концы. В целом, качество чтения нуклеотидов улучшилось по всей длине. На заднем конце улучшение было наиболее значительным, что ожидаемо, поскольку качество чтения падает к концу рида в связи с особенностями работы секвенатора.
Далее было проведено картирование чтений, анализ выравнивания, поиск SNP и инделей. Использованные команды приведены выше.
Поиск SNP и инделей
Следуя руководству, я создала файл со списком отличий между референсом и чтениями в формате .vcf.
Затем требовалось найти и описать в отчете три полиморфизма из .vcf файла. Легенда к основной части .vcf файла - таблице - приведена в шапке, оттуда я брала необходимую информацию.
Аннотация SNP
Требовалось с помощью программы annovar проаннотировать только полученные snp (индели не нужно), пользуясь базами данных: refgene, dbsnp, 1000 genomes, GWAS, Clinvar.
Последовательность действий:
1) Получить из .vcf файла другой файл, с которым умеет работать annovar, с помощью скрипта convert2annovar.pl. Скрипт лежит на kodomo: /nfs/srv/databases/annovar . Для дальнейшей работы я переместила его в свою рабочую директорию и поменяла права (chmod agu+xwr convert2annovar.pl).
Аннотировать по базам данных с помощью команды annotate_variation.pl
Примечание: поскольку просили аннотировать только SNP, но не индели, из всех таблиц с аннотациями по базам данных индели длиной больше 1 удалены.
Результаты
Результаты в форме таблиц, в том числе описание полиморфизмов из .vcf файла и сводная таблица по аннотациям по базам данных, приведены в прикрепленном xslx-файле (приведен выше).
Отчет о полученных snp:
1) Сколько snp и сколько инделей получили:
В .vcf файле полиморфизмов: 5 инделей, в т.ч. 2 однонуклеотидных; с ними 61 SNP, без них 59
По базам данных: 61 SNP
2) На какие категории делит snp база данных refseq в annovar? Сколько snp попало в каждую группу?
Категории: exonic, splicing, ncRNA, UTR5, UTR3, intronic, upstream, downstream, intergenic

Рис.4 Категории SNP в бд refseq
В наших чтений к каждому классу принадлежат:
intronic: 42
exonic: 17
UTR3: 2
downstream: 2
3) rs имееет 56 полиморфизмов, в том числе 54 SNP и 1 индель
4)SNP попали в 3 гена:
TNFSF15 - tumor necrosis factor receptor superfamily member 25. Белок, который кодирует данных ген, принадлежит к суперсемейству TNF-рецепторов. Данный рецептор экспрессируется преимущественно в тканях, богатых лимфоцитами, и, возможно, участвует в регуляции лимфоцитарного гомеостаза.
NDUFA8 - NADH:ubiquinone oxidoreductase subunit A8. Белок, который кодирует данный ген, является компонентом комплекса I 19 kDA subunit family, который в свою очередь является частью электрон-транспортной цепи митохондрий.
ABO - ABO blood group (transferase A, alpha 1-3-N-acetylgalactosaminyltransferase; transferase B, alpha 1-3-galactosyltransferase). Этот ген кодирует белки, связанные с первой открытой системой определения группы крови - ABO. Группа крови у индивида определяется тем, какие аллели данного гена у него присутствуют. Группа 'O' получается в результате делеции гуанина-285 возле N-конца белка, из-за чего происходит сдвиг рамки считывания и трансляция, фактически, другого белка (неактивного). В результате у человека отсутствуют антигены A и B. У людей с A, B и AB-аллелями экспрессируются активные гликозидтрансферазы, превращающие предшествующий антиген H в антиген A или B. Также существуют другие, более редкие аллели этого гена.
5) Хорошее ли покрытие и качество у найденных полиморфизмов?
Информация для ответа на этот вопрос приведена в таблице с аннотациями по RefGene (и по другим бд тоже, но в RefGene аннотаций больше всего, и они включают в себя все найденные в других бд полиморфизмы). Итак, 20 из 61 SNP имеют покрытие > 10, 29 - больше 5, то есть довольно хорошее. Вторая половина имеет покрытие <= 4, причем 19 из них имеют покрытие 1 (соответствующее качество чтений для них тоже низко, часто менее 10, поэтому я не уверена, что это действительно полиморфизмы, а не ошибки секвенирования). SNP с хорошим покрытием также имеют высокое качество чтений.
6) К каким нуклеотидным и аминокислотным заменам привели snp?
Необходимые данные приведены в файле exonic_variant_function, полученном при аннотации по refseq (скопирован в прикрепленную таблицу). В нем содержится информация о заменах в экзонах генов и их функциональности. Для моих чтений данные известны только для одного SNP (rs3810936), при этом произошедная замена синонимична (валин заменился на валин).
7) Частота найденных snp
Данные о частоте аллели я брала из аннотаций SNP из базы данных 1000genomes. Информация имеется для большей части SNP. Среди аннотированных полиморфизмов большая часть весьма распространенные, с частотами от 0.7 до 0.3. Есть только один редкий SNP - rs78979172 в гене TNFSF15, с частотой встречаемости 0,0341454. Функция его неизвестна.
8) Что можно сказать о клинической аннотации snp?
Как я поняла, клинические аннотации приводят базы данных GWAS (аннотировались 9) и Clinvar (2).
SNP в ABO связаны с характеристиками крови и кровотока, что неудивительно. Например, присутствуют полиморфизмы, которые связаны со временем свертывания крови, концентрациями факторов свертываемости и риском венозной тромбоэмболии. SNP, найденные в гене в TNFSF15, вызывают риск заболеваний кишечника: болезни Крона и язвенного колита. Единственный аннотированный полиморфизм в гене NDUFA8 связан с риском ожирения, что понятно, поскольку продукт гена является компонетом дыхательной цепи митохондрий и, таким образом, связан с энергетическим балансом клетки.