Практикум 2.
A- и В- формы ДНК. Структура РНК.
Задание 1. Требовалось построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
С помощью программы fiber пакета 3DNA были построены A-, B- и Z-формы дуплекса ДНК, последовательность одной из нитей которого представляла собой 5 раз повторенную последовательность "gatc" (для a- и b-форм; Z-форму программа fiber по умолчанию делает состоящей из пар G-C).
Результаты представлены на рис. 1-3:
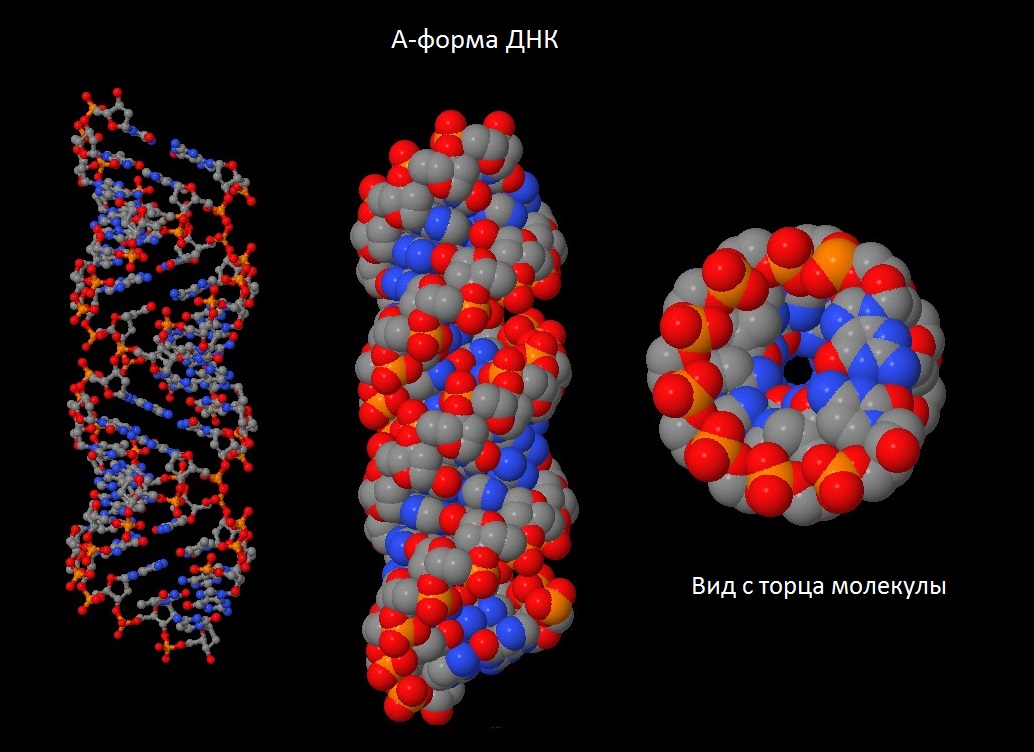
Рис.1 A-форма ДНК
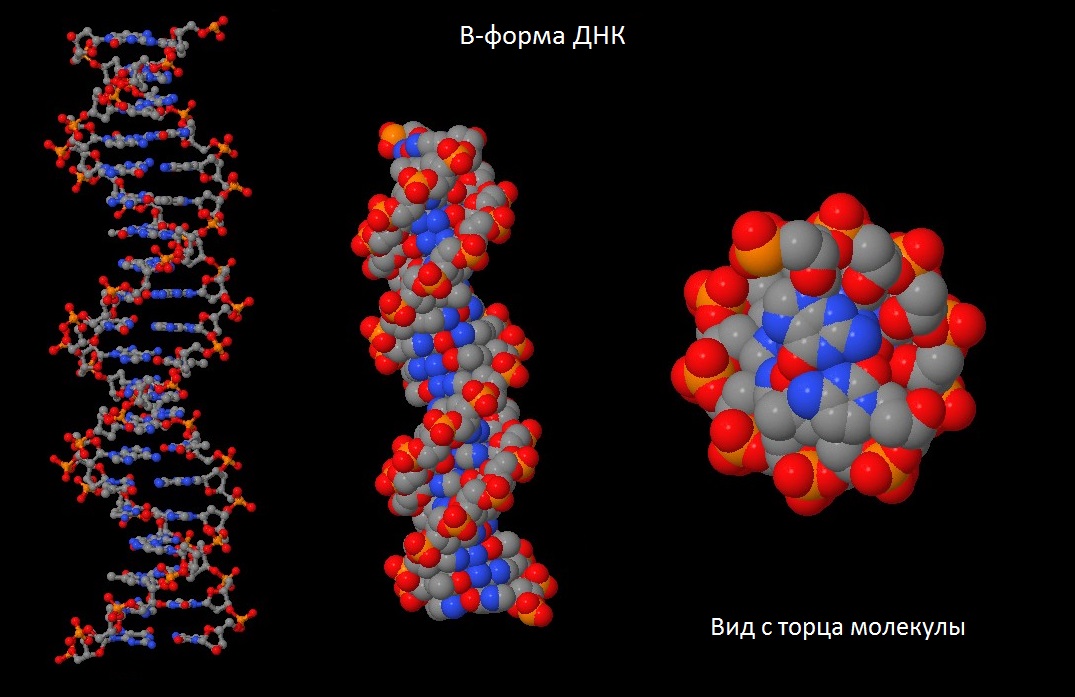
Рис.2 B-форма ДНК
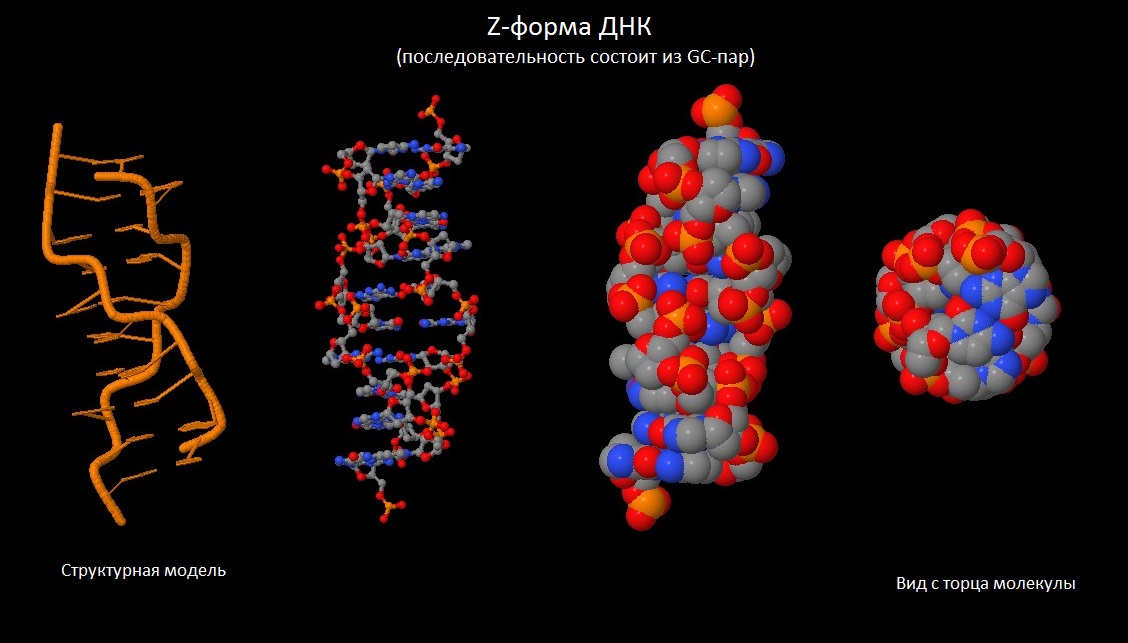
Рис.3 Z-форма ДНК
Задание 2.
Требовалось Научиться выделять разные атомы и химические группировки, используя предопределенные множества JMol на примере А-формы ДНК.
Выделены:
1. Сахарофосфатный остов ДНК
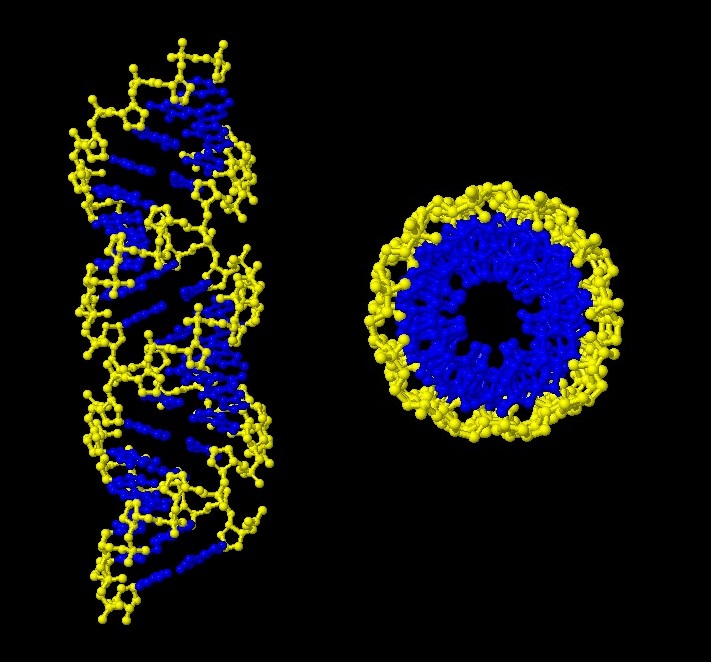
Рис.4 Сахарофосфатный остов ДНК покрашен в желтый, азостистые основания - в синий.
2. Все нуклеотиды
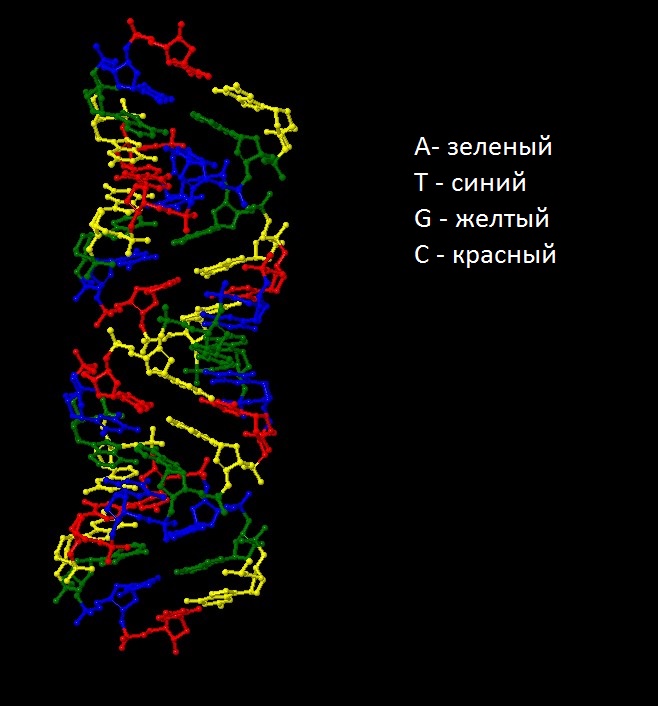
Рис.5 Нуклеотиды ДНК.
3. Все нуклеотиды, содержащие основание аденин
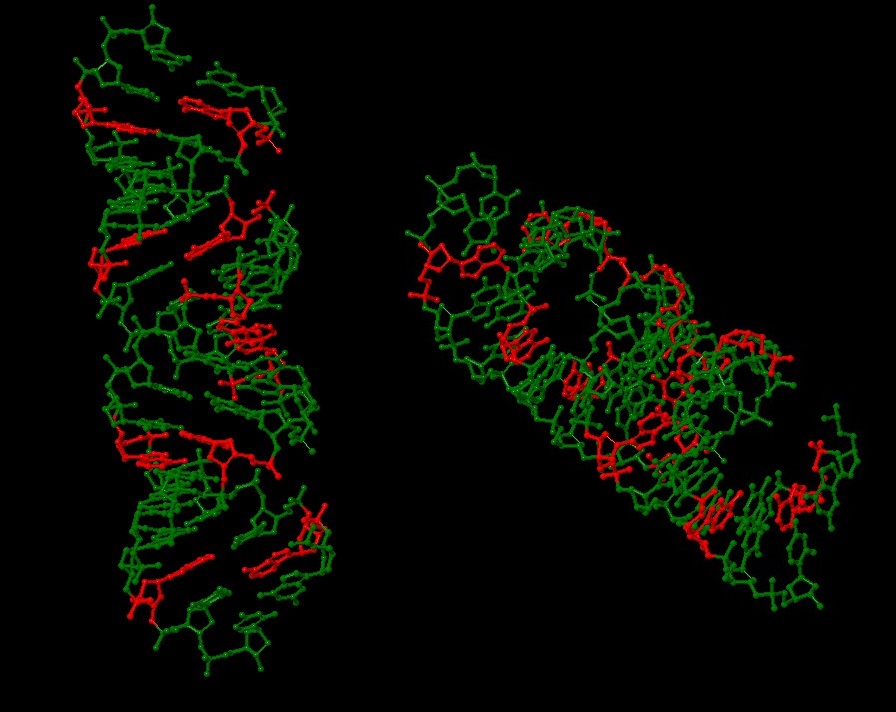
Рис.6 Все нуклеотиды, содержащие основание аденин, покрашены в желтый.
4. Атом N7 во всех гуанинах и/или только в первом по последовательности
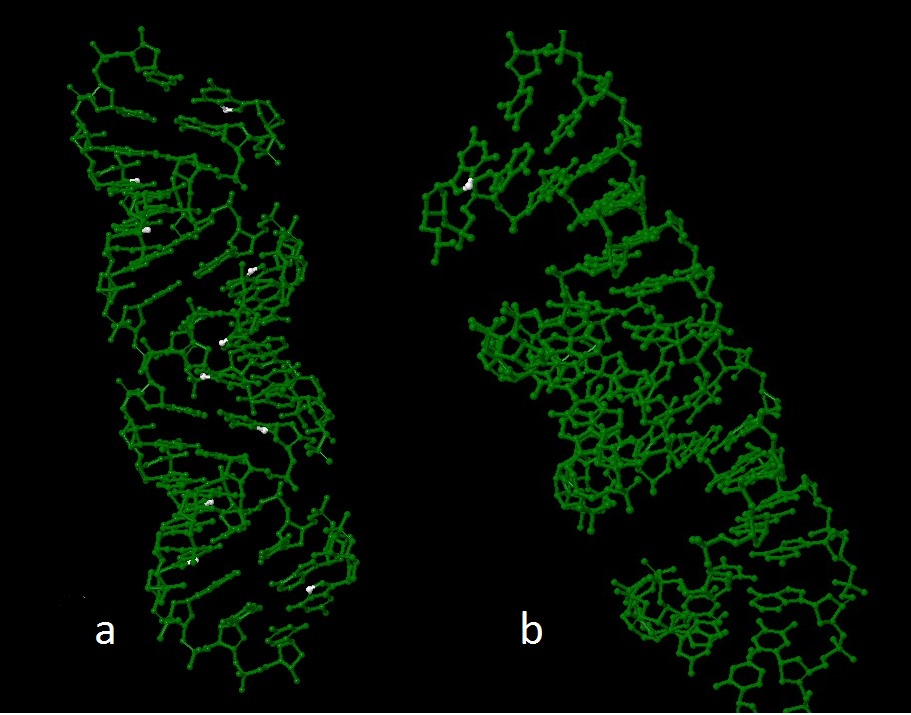
Рис.7 a) атом N7 во всех гуанинах, b) атом N7 только в первом по последовательности гуанине. Искомые атомы выделены белым цветом.
Задание 2.
Требовалось найти и скачать заданные файлы из базы данных PDB. Затем нужно было получить изображение только нуклеиновой кислоты, входящей в найденные структуры, и проверить эту ДНК или РНК на наличие разрывов.
Структура 1. Аргинил-тРНК синтетаза в комплексе с тРНК и L-аргинином. PDB ID - 1F7U.
С помощью средств программы JMol получено изображение только тРНК (рис.8). Была использована команда restrict rna. Разрывов в структуре не обнаружено.
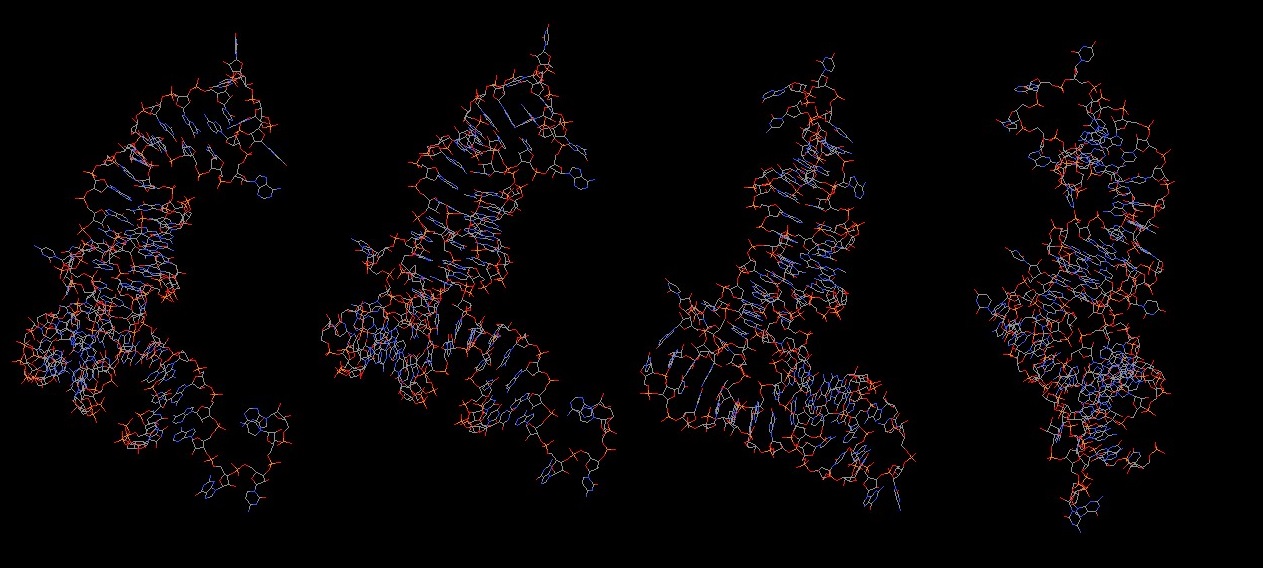
Рис.8 Изображение тРНК в проволочной модели с разных ракурсов, полученное из PDB-файла 1F7U.
Структура 2. Комплекс репресор/оператор триптофанового оперона E.coli. PDB ID - 1TRO.
С помощью средств программы JMol получено изображение только ДНК. Была использована команда restrict dna. В структуре обнаружены разрывы в обеих цепях ДНК. После применения окраски по цепям ДНК (color chain) выяснилось, что структура действительно содержит несколько фрагментов, а именно 2 дуплекса ДНК. Результаты представлены на рис.9.
Рис.9 Изображение дуплекса ДНК, полученное из PDB-файла 1TRO.
a) Изображение всей ДНК из файла PDB в проволочной модели; b) двуцепочечный разрыв в структуре; c) раскраска по цепям ДНК: видно, что в структуре присутствуют 4 разных цепи.
Задание 3.
Сравнение структур 3-х форм ДНК с помощью средств JMol.
Упражнение 1.
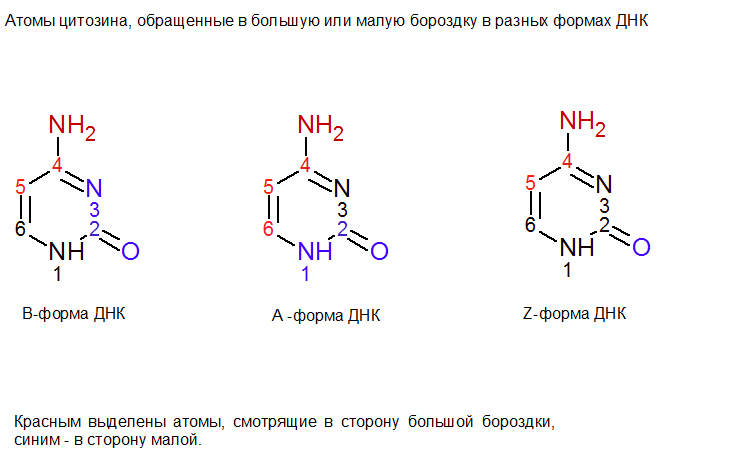
Требовалось научиться находить большие и малые бороздки. В файлах, полученных в первом задании исодержащих дуплексы ДНК в A, B- или Z-форме, нужно было выбрать заданное азотистое основание(в моем случае - цитозин) и определить, какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой.
Результаты:
B-форма ДНК:
В сторону большой бороздки обращены атомы C4, C5, N4.
В сторону малой бороздки обращены атомы C2, O2, N3. 5
Остальные атомы основания (N1, C6) не обращены явно в какую-либо из бороздок и находятся в плоскости витка спирали.
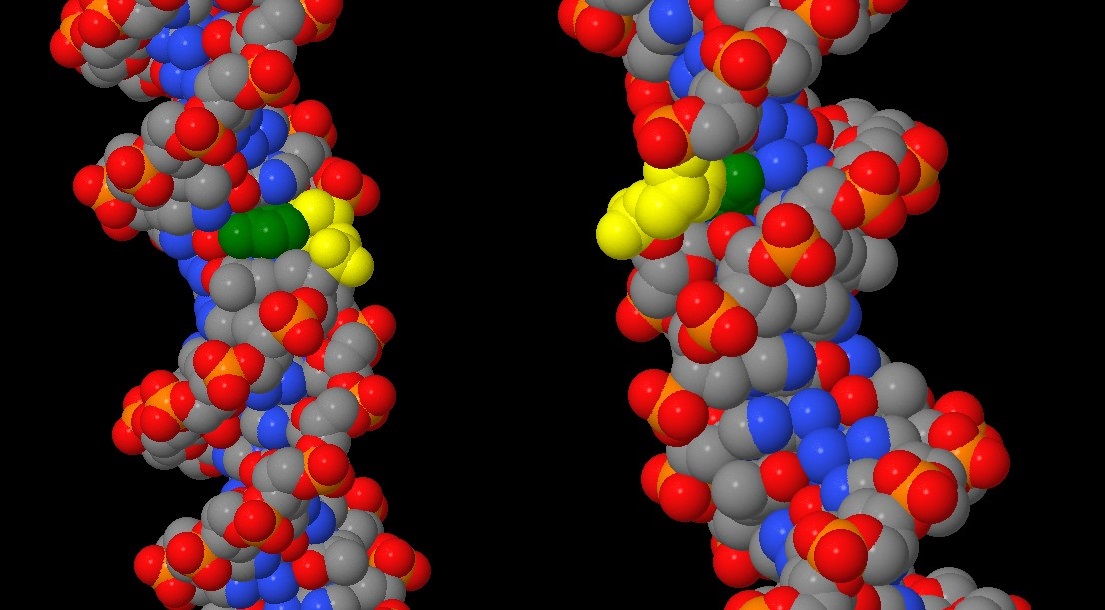
Рис.11
A-форма ДНК:
В сторону большой бороздки обращены атомы (глубокой) C4, C5, C6, N4.
В сторону малой бороздки обращены атомы O2,C2,N1.
Оставшийся атом основания - N3, - не обращен явно в какую-либо из бороздок и находится в плоскости витка спирали.
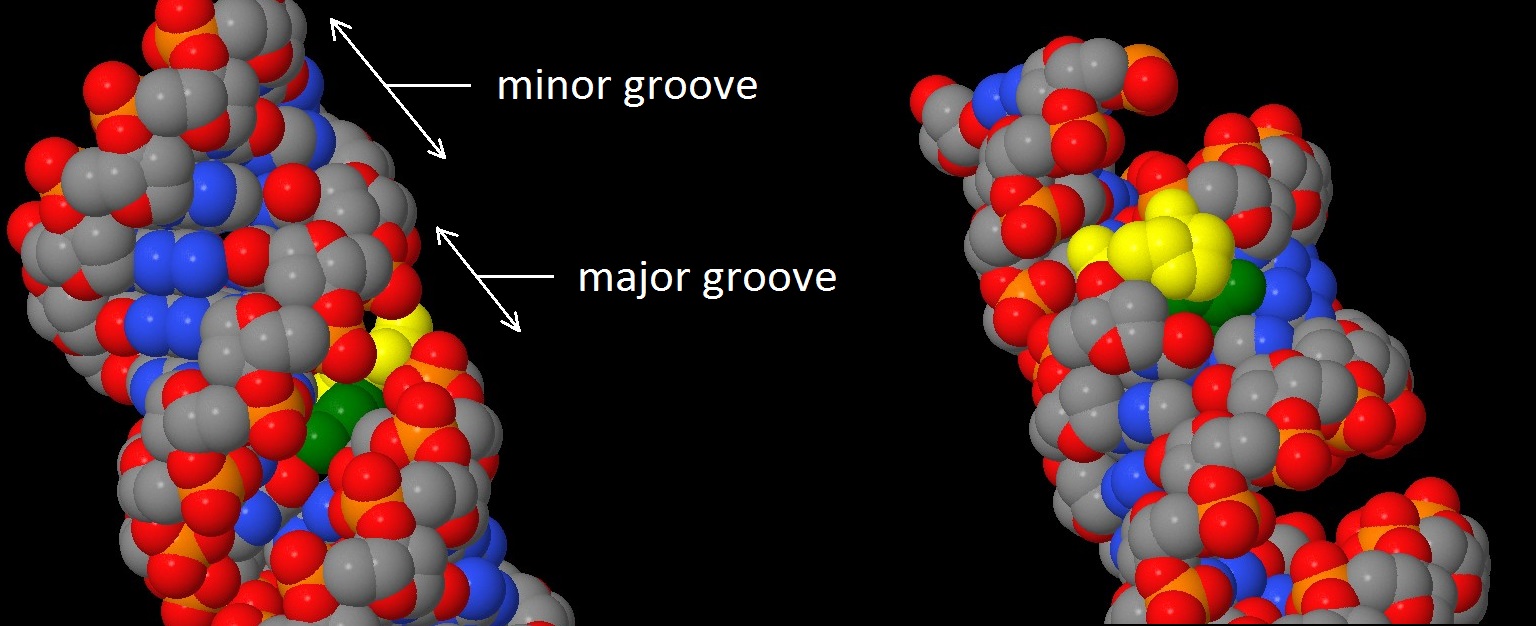
Рис.12
Z-форма ДНК:
В сторону большой бороздки обращены атомы C5, C6, N4.
В сторону малой бороздки обращены атомы O2.
Остальные атомы основания (C6, C2, N1, N3) не обращены явно в какую-либо из бороздок и находятся в плоскости витка спирали.
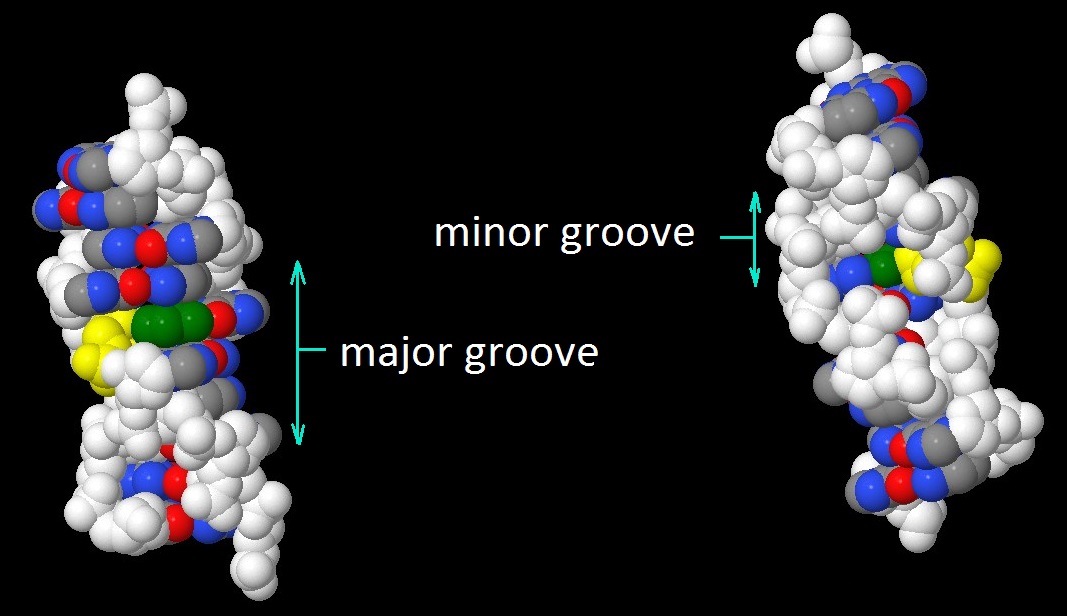
Рис.13
Также было необходимо с помощью ChemSketch получить изображение данного основания, выделить красным цветом атомы, смотрящие в сторону большой бороздки, и синим - в сторону малой. Результаты приведены ниже:
Упражнение 2.
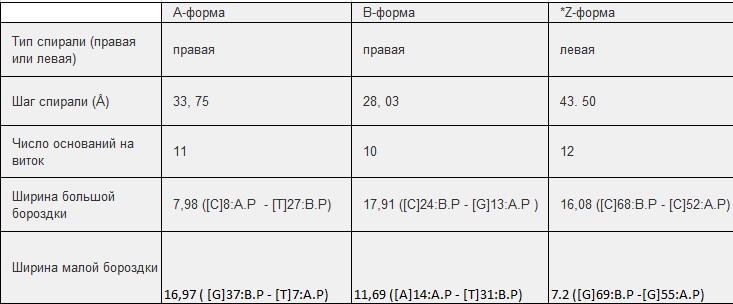
Требовалось сравнить основные спиральные параметры разных форм ДНК. Результаты представлены в таблице 1.
Таблица 1. Параметры разных форм ДНК
Упражнение 3.
Нужно было сравнить торсионные углы в структурах А- и В-форм. С помощью команды Settings->Torsion JMol были измерены торсионные углы нуклеотида, содержащего цитозин, в А- и В-форме.
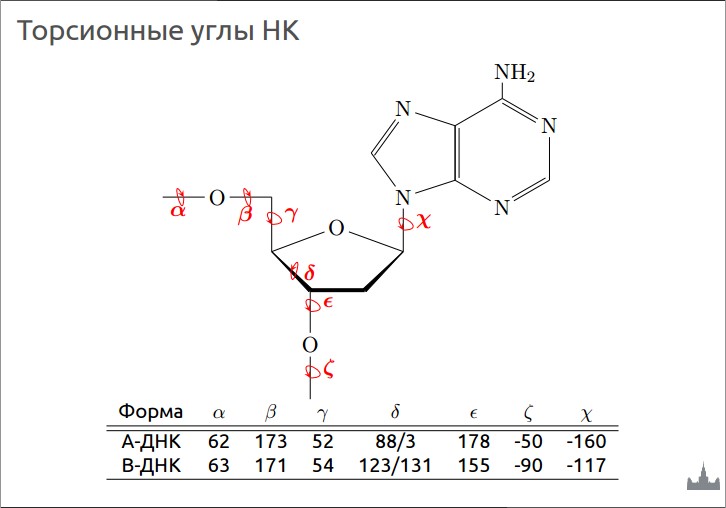
Рис.14 Шпаргалка по торсионным углам из презентации.
Результаты:
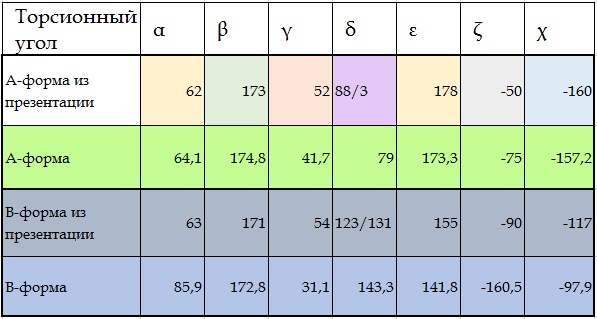
Рис.15 Торсионные углы цитидилового нуклеотида в разных формах ДНК.
Задание 4.
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Использовались программы find_pair и analyze. Была применена команда find_pair -t XXXX.pdb stdout | analyze .
Результатом работы программ являлись несколько файлов, в том числе файлы с расширением .out, откуда и бралась большая часть информации для отчета.
Упражнение 1.
Требовалось определить торсионные углы в заданной структуре ДНК и тРНК; с помощью Excel определить среднее значение каждого из торсионных углов (краевые нуклеотиды не рассматривать).
Результаты:
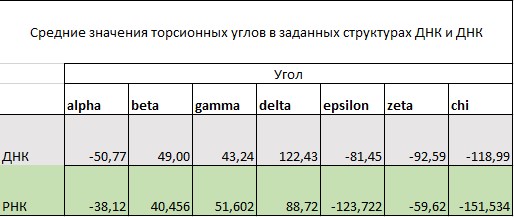
Также необходимо было определить номер самого "деформированного" нуклеотида (с наиболее отклоняющимся значением одного или нескольких угловторсионных углов).
В ДНК это нуклеотиды С5 цепи 1 и С15 цепи 2 (нумерация соответствует PDB-файлу TRO).
В РНК это G21,G11(цепь 1) и g16 и G23 (цепь 2) (нумерация соответствует прикрепленному файлу 1F7U_old.out).
Упражнение 2.
Требовалось научиться определять структуру водородных связей.
1. Определить номера нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК.
Результаты:
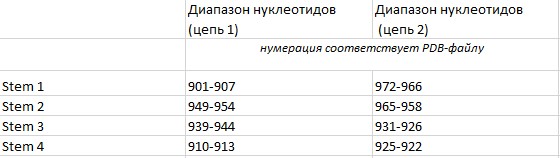
Как видно из таблицы, в структуре данной тРНК присутствуют 4 стебля. Это совпадает с канонической вторичной структурой тРНК.
2. Определить неканонические пары оснований в структуре тРНК.
Результаты:

Рис.12 Неканонические пары оснований в тРНК.
Структура содержит 9 неканонических пар оснований. Присутствуют пары с участием модифицированных (в данном случае метилированых) оснований и псевдоуридина, а так же не-уотсон-криковские пары с участием нормальных нуклеотидов.
3. Определить, есть ли дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру (для этого следует рассмотреть комплементарные пары, не имеющие отношения к стеблям).
Результаты:

Рис.12 Водородные связи в тРНК. Пары оснований, входящие в стебли, выделены зеленым цветом, не входящие в стебли - красным.
Итого, согласно результатам работы программ find_pair и analyse, обнаружено 4 пары оснований ([PSU]955 -- [G]917, [A]914 -- [U]908, [A]915 -- [U]948, [G]918 -- [C]956), образующих водородные связи и при этом не входящих в стебли. Таким образом, эти водородные связи участвуют в стабилизации третичной структуры тРНК.
Нужно заметить, что JMol не показывает водородные связи между A914 и U908. Скорее всего, проблема заключается в настройках JMol. Водородные связи между остальными 3 парами, действительно, видны в JMol.
Упражнение 3.
Требовалось научиться находить возможные стекинг-взаимодействия.
Cтекинг-взаимодействия - это взаимодействия, возникающие при «стопочном» расположении нескольких ароматических структур. Они появляются за счет межмолекулярного перекрывания p-орбиталей в π-сопряженных системах, какими являются ароматические структуры, в том числе пуриновые и пиримидиновые кольца азотистых оснований. Стекинг-взаимодействия обычны для ДНК и также встречаются в РНК и белках.
Что было сделано:
В файле с расширением .out в разделе "Overlap area" были найдены три последовательные пары с наибольшим перекрыванием:
12 Gu/aC 8.02( 2.73) 0.00( 0.00) 0.00( 0.00) 5.32( 1.87) 13.34( 4.60)
13 uP/Ga 6.43( 2.46) 0.00( 0.00) 0.00( 0.00) 7.33( 2.23) 13.77( 4.69)
То есть пары [G]953 - [C]961, [5MU]954 - [1MA]958, [PSU]955 - [G]917, соответственно нумерации исходного PDB-файла.
Для них попарно были получены изображения стекинга с помощью программ ex_str и stack2img:
1. [G]953 - [C]961 и [5MU]954 - [1MA]958
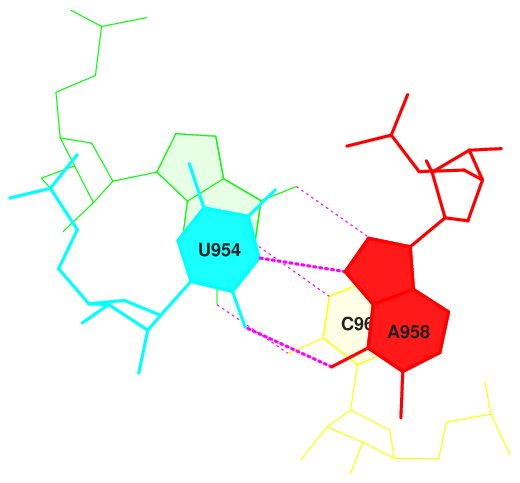
Рис.13 Изображение стекинг-взаимодействия между парами [G]953 - [C]961 и [5MU]954 - [1MA]958 (step 12).
2. [5MU]954 - [1MA]958, [PSU]955 - [G]917
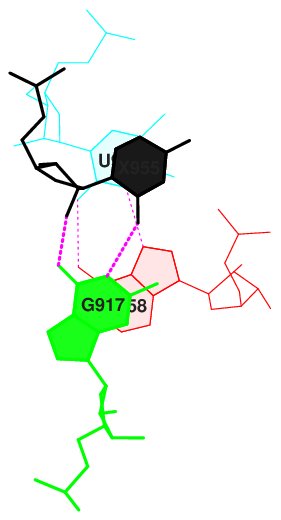
Рис.14 Изображение стекинг-взаимодействия между парами [5MU]954 - [1MA]958, [PSU]955 - [G]917(step 13).
Также были получены изображения данных пар из JMol, на которых хорошо видно перекрывание оснований.
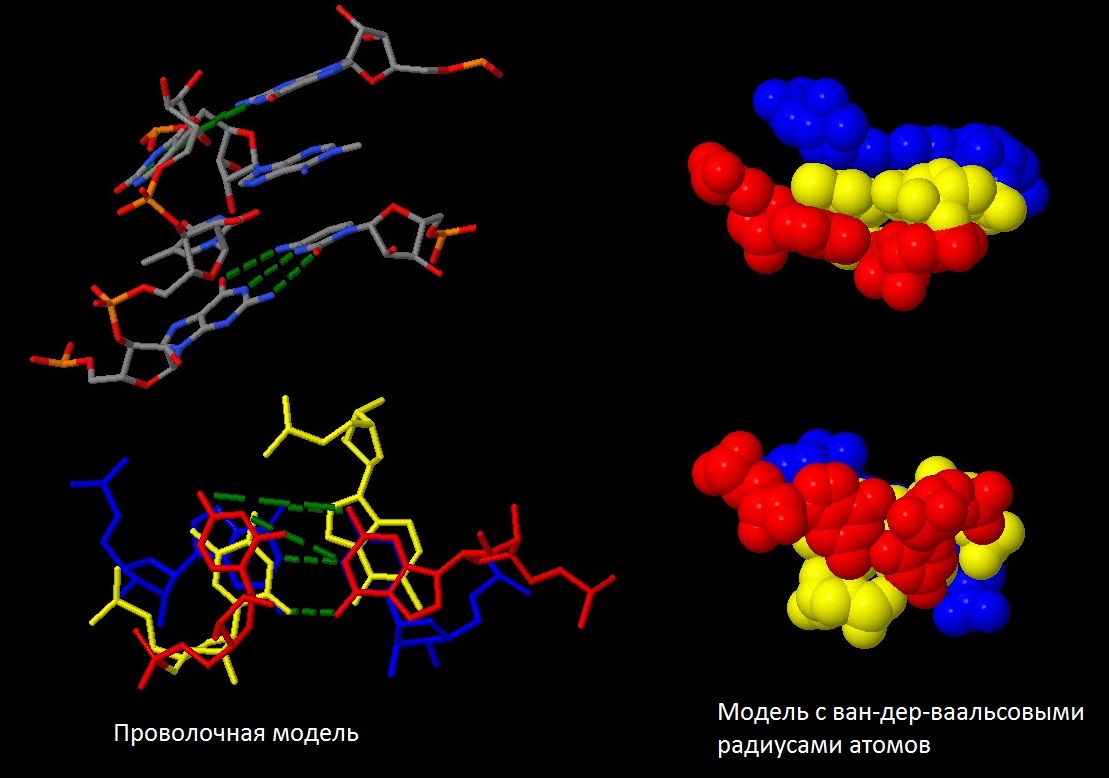
Рис.14 Взаимное расположение пар [G]953 - [C]961, [5MU]954 - [1MA]958, [PSU]955 - [G]917, между которыми предполагается стекинг-взаимодействие.