Практикум 3.Предсказание вторичной структуры заданной тРНК.
Упр.1 Предсказание вторичной структуры тРНК путем поиска инвертированных повторов.
Поскольку тРНК имеют более-менее консервативную вторичную структуру, так называемый "клеверный лист", мы вправе рассчитывать на то, что в данной аргининовой тРНК, взятой из PDB-файла 1F7U, будут присутствовать характерные стебли и петли.
Рис.1 Вторичная структура тРНК. Рисунок взят из статьи О.О. Фаворовой "Строение транспортных РНК и их функция на первом (предрибосомном) этапе биосинтеза белков".
Использованные программы:
1. einverted
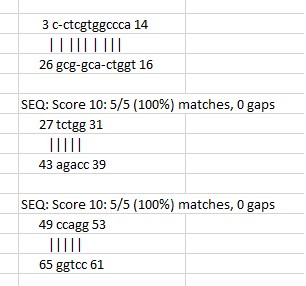
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. Требовалось найти возможные комплементарные участки в последовательности исследуемой тРНК и сравнить их с описанием, полученным ранее с помощью find_pair.
Примечание: в последовательности тРНК в формате fasta пришлось поменять инозин на гуанин, поскольку программа не распознавала букву I как обозначение нуклеотида. Гуанин в качестве замены был выбран сообразно сходству химического строения.
Лучший результат работы einverted:
2. find_pairs
3. RNAfold, реализующая алгоритм Зукера
Образец обращения к программе: cat trna.fasta | RNAfold --MEA
Лучший результат работы RNAfold (структура, наиболее похожая на реальную):
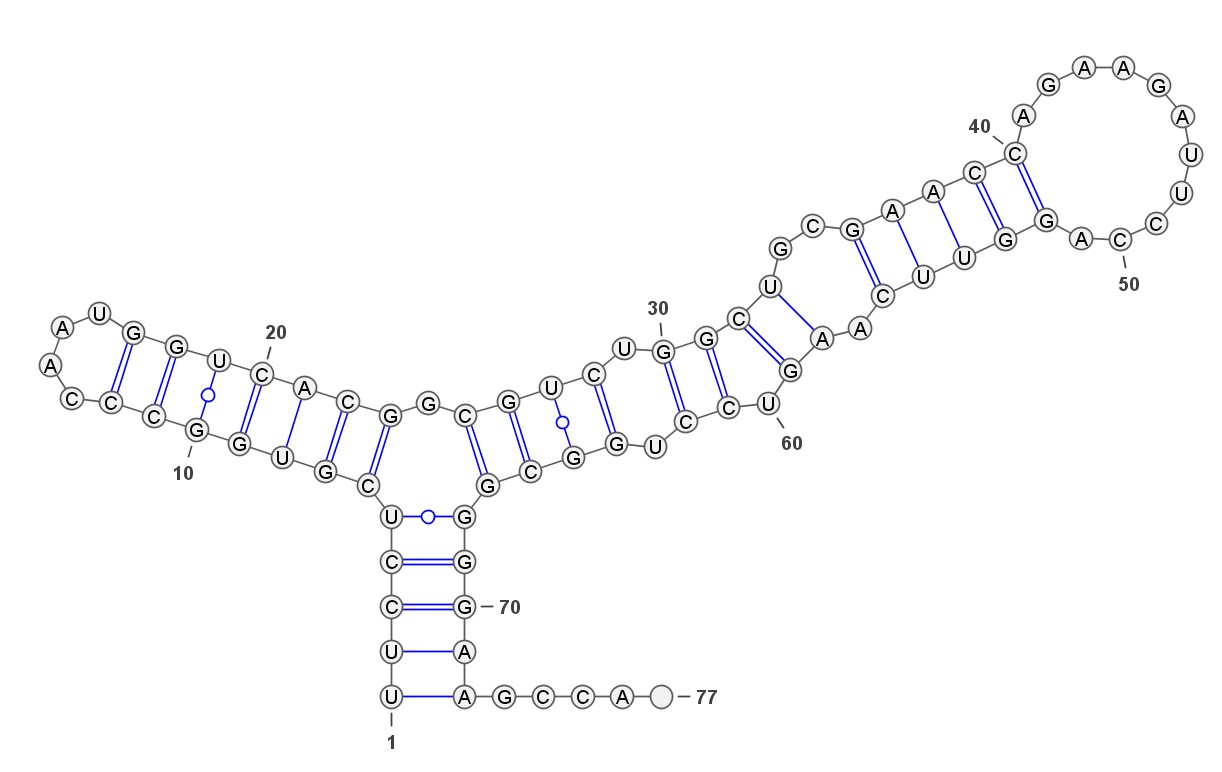
Рис.2 Предсказание вторичной структуры тРНК программой RNAfold. Изображение получено с помощью пакета VARNA.
Увы, данная структура имеет мало общего с реальной. Результат с минимальной энергией выглядит еще хуже:
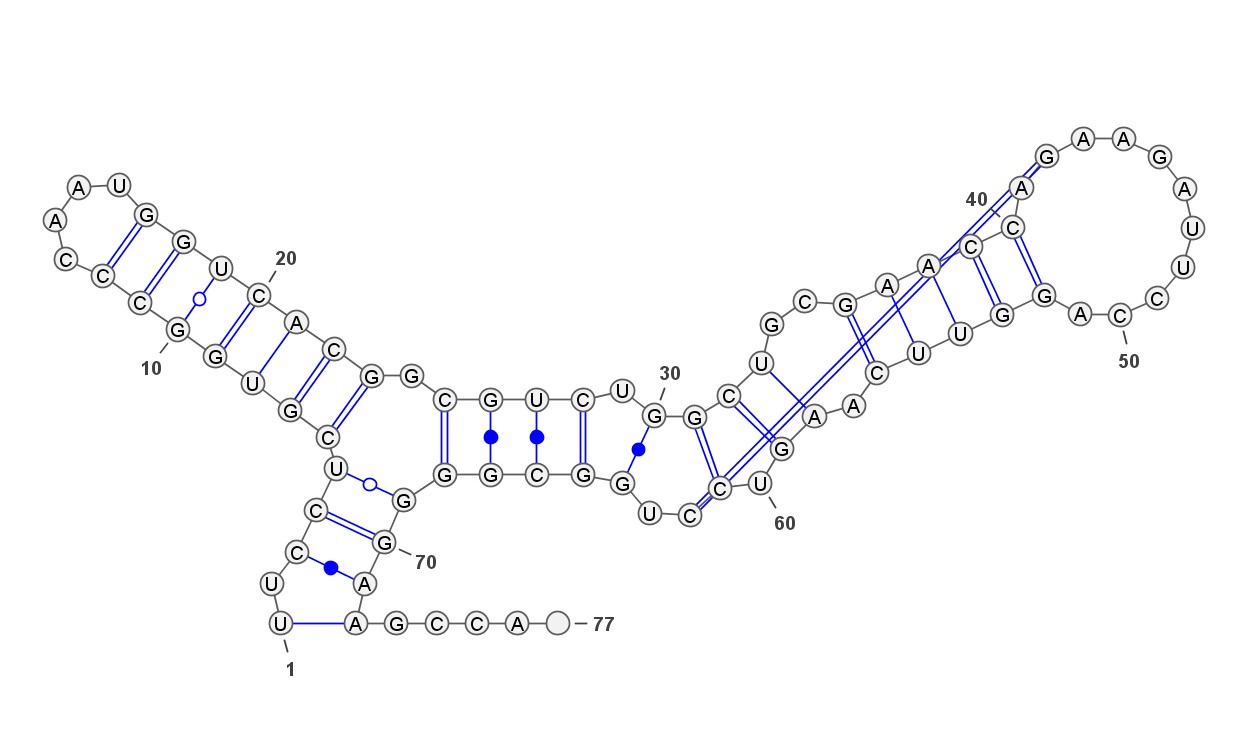
Рис.3 Предсказание вторичной структуры тРНК программой RNAfold. Структура с минимальной энергией. Изображение получено с помощью пакета VARNA.
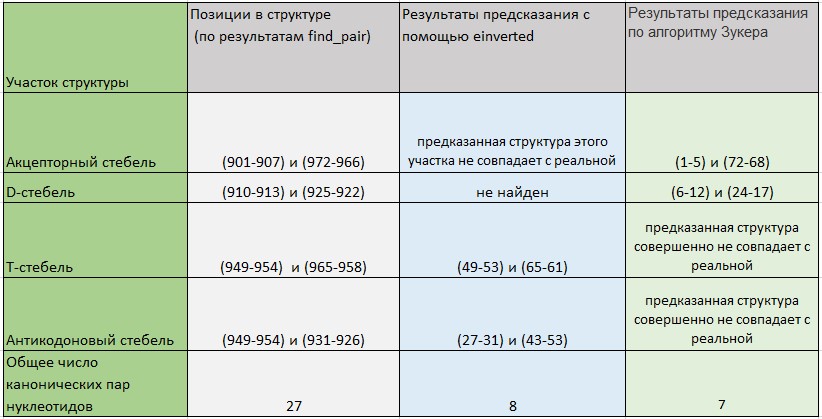
Сводная таблица результатов:
Задание 2. Поиск ДНК-белковых контактов в заданной структуре
Упр.2 Описание ДНК-белковых контактов в заданной структуре и сравнение количества контактов разной природы.
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
С помошью команды define в JMol были получены требуемые множества атомов ДНК, затем с помощью команды select within (расстояние, множество) были получены множества атомов белка, контактирующие с данными атомами ДНК.
Результаты:
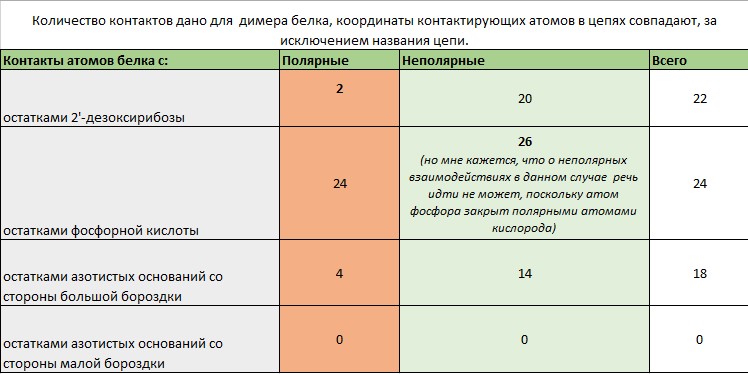
Из таблицы видно, что подавляющая часть контактов ДНК-белок в данном случае неспецифична относительно последовательности ДНК( присутствуют в основном контакты с остовом ДНК - 46 шт против 18 контактов с атомами азотистых оснований). За специфичное распознавание последовательности ДНК в данном случае, по-видимому, отвечают контакты белка с атомами малой бороздки ДНК.
Кажется странным, что нет контактов атомов белка с атомами большой бороздки. При этом в Jmol видно, что часть аминокислотных остатков белка все же заходит в большую бороздку ДНК.
Упр.3. Получить популярную схему ДНК-белковых контактов с помощью программы nucplot.
Результат:
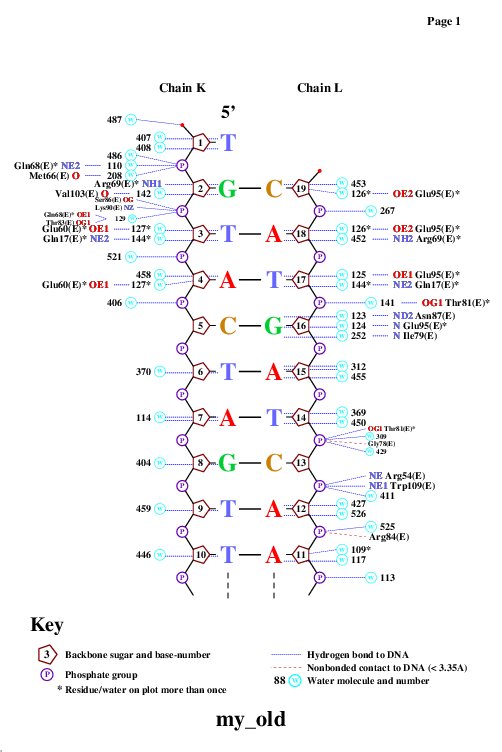
Рис.3 Результат работы nucplot #1
Примечание: в pdb-файле странная нумерация атомов воды, в частности, там есть [hoh]125:E и [hoh]125:L, две молекулы с одинаковыми номерами, но приписанные к разным цепят ДНК или белка. При этом nucplot, похоже, считает их за одну молекулу, из-за чего выдает неправильные результаты. В частности, на данном рисунке видно, что [GLU]95:E взаимодействует с [T]17 через [HOH]125. На самом же деле эти остатки находятся очень далеко друг от друга. См. рис. 4:
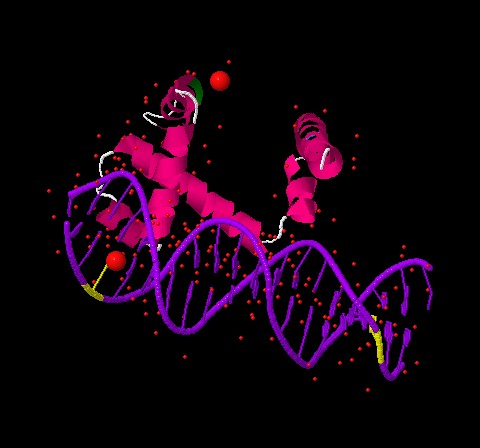
Рис.4 Взаимное расположение [GLU]95:E и [T]17. Желтым отмечен [T]17, зеленым - [GLU]95:E, большими красными шариками - две молекулы [hoh]125:E и [hoh]125:L.
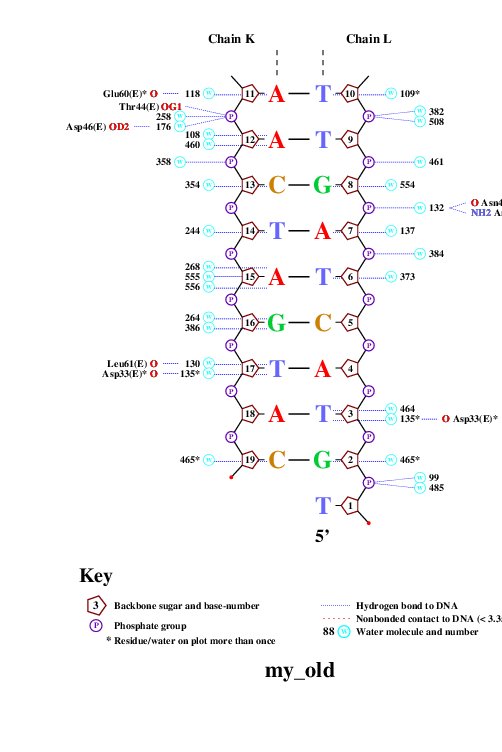
Рис.5 Результат работы nucplot #2
Примечание: не уверена, что следует безоглядно рассматривать все приведенные контакты атомов через воду, поскольку, если смотреть через JMOL, эти атомы расположены довольно далеко друг от друга, в некоторых случаях на расстоянии более 4 нанометров.
Упр.4.
На полученной схеме выбрать:
1) аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК:
Все аминокислотные остатки имеют не более одного прямого (не через воду) контакта с атомами ДНК. Контакты через воду кажутся мне подозрительными, я бы вообще не считала их за контакты. Поэтому выбрать требуемый остаток не могу. Привожу изображение одного их остатков, [TRP]109:E.
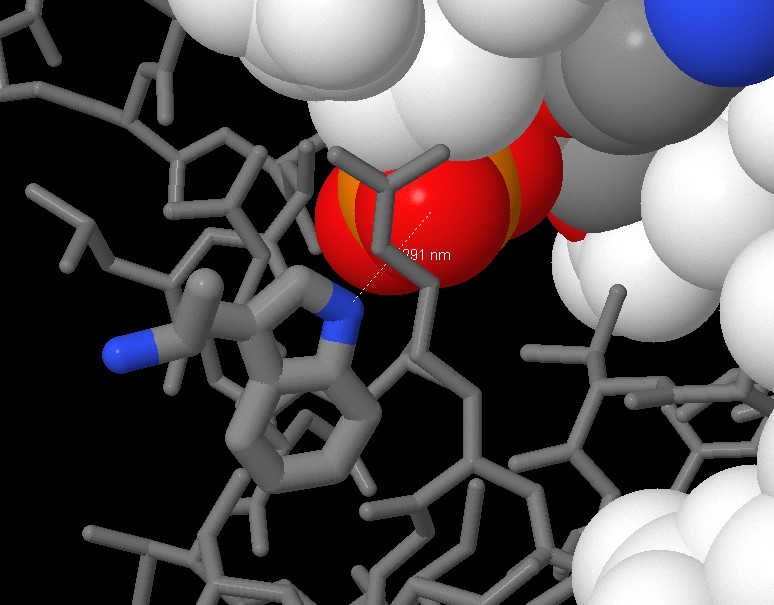
Рис. 6 Взаимодействие между [TRP]109:E (толстая проволока, раскрашен по типам атомов) и [C]13:L.O2P (толстая проволока, раскрашен по типам атомов).
2)аминокислотный остаток, по-вашему мнению, наиболее важный для распознавания последовательности ДНК.
Я выбрала [ARG]69:E, поскольку он единственный связан (образует водородную связь) с атомами азотистого основания (а не остова ДНК) напрямую (не через молекулы воды).
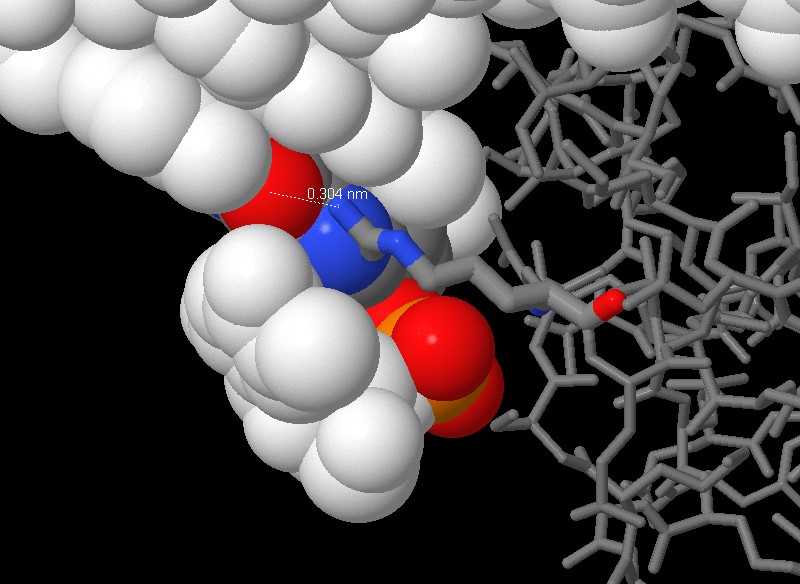
Рис.7 Взаимодействие между [ARG]69:E (толстая проволока, раскрашен по типам атомов) и [G]2 (большие шарики, раскрашен по типам атомов). Белок в виде серой проволочной модели, ДНК в виде белых ван-дер-ваальсовых радиусов.