Практикум 6. Чтение последовательностей по Сэнгеру
Файлы:
1. Исходные файлы с хроматограммами:
2. Выравнивание прямой и комплементарной к обратной последовательности в проекте JalView скачать
Измененные нуклеотиды отмечены маленькими буквами.
3. "Чистая" последовательность прямой цепи в формате fasta скачать
Задание 1. Получение последовательности фрагмента ДНК по результатам хроматограмм из капиллярного секвенатора по Сэнгеру и последовательности, сгенерированной программой.
Порядок выполнения работы:
Оба файла, с хроматограммой прямой и обратной цепи, были открыты в программе Chromos. Обратная цепь была изменена на комплементарную и перевернута (опция reverse complement), чтобы можно было сравнивать последовательности обеих цепей. С помощью опции find последовательности были выровнены относительно друг друга.
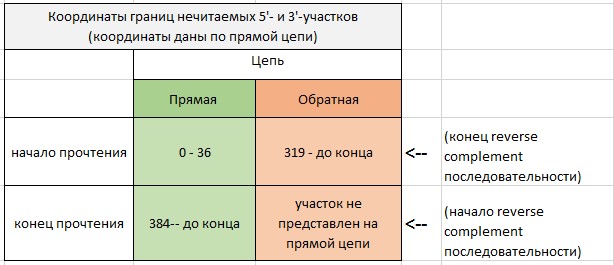
Затем были определены границы нечитаемых 5'- и 3'-участков в каждой последовательности. Координаты определялись по прямой цепи. Необходимо заметить, что после удаления нечитаемых участков нумерация нуклеотидов автоматически изменилась, так что приведенные координаты соответствуют исходным файлам, но не соответствуют файлам, полученным в ходе дальнейшей работы.
Границы нечитаемых участков:
Далее нечитаемые 5'- и 3'- концы были удалены.
Было охарактеризовано качество каждой хроматограммы:
Прямая цепь:
- В среднем сигнал превосходит шум в 6 раз.
- Средняя сила сигнала вдоль оси X равномерна, сила шума увеличивается к концу хроматограммы приблизительно в 2 раза.
- Сила у разных очевидных сигналов может различаться в 6 раз. При этом G и A чаще других нуклеотидов имеют очень сильный или очень слабый сигнал, в то время как T и C имеют меньший разброс силы сигнала.
- Другие собенности хроматограммы: 3'-концевой нечитаемый участок отсутствует, поскольку последовательность короткая (около 380 п.н.), поэтому нет эффекта размывания пиков.
- В районе 50-го нуклеотида - нечитаемая область, причем в начале хроматограммы обратной цепи тоже есть аналогичная "клякса". Думаю, виноват секвенатор.
- В целом, хроматограмма довольно хорошая.
Обратная цепь:
- В среднем сигнал превосходит шум в 10-12 раз.
- Средняя сила сигнала вдоль оси X практически равномерна, незначительно увеличивается к концу хроматограммы. Сила шума равномерна.
- Сила у разных очевидных сигналов может отличаться в 10 раз. Что касается зависимости силы сигнала от нуклеотида, высокие пики дает почти исключительно C.
- Другие собенности хроматограммы: очень маленький 3'-концевой нечитаемый участок, наличие нечитаемой области в районе 290-300 (нумерация по прямой цепи).
- Хроматограмма более-менее хорошая.
Редактирование последовательностей.
Для начала следует привести пример хорошего участка, прочтение которого однозначно:
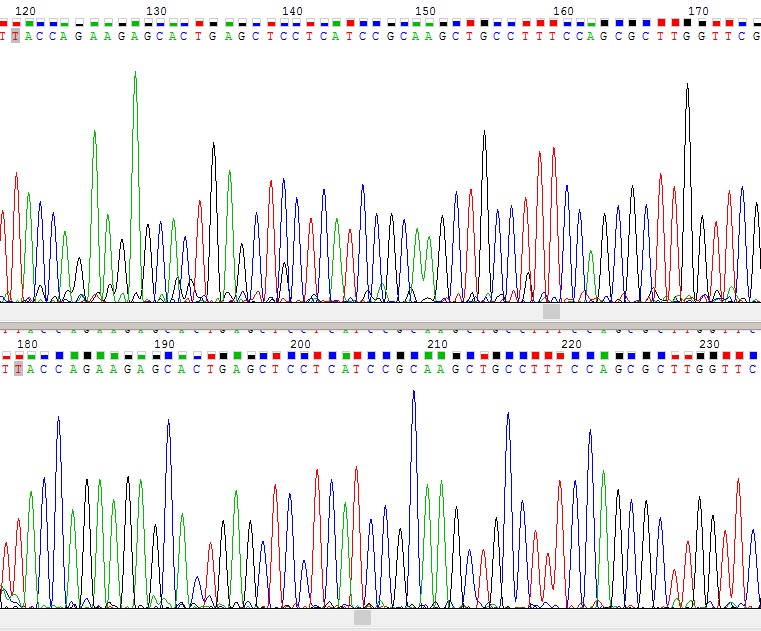 Рис.1 "Хороший" участок хроматограммы. Прямая цепь наверху, обратная внизу. Видно, что quality values высокие.
Рис.1 "Хороший" участок хроматограммы. Прямая цепь наверху, обратная внизу. Видно, что quality values высокие.
Редактирование прямой цепи
Проблема №1. Та самая клякса в области 45-55.
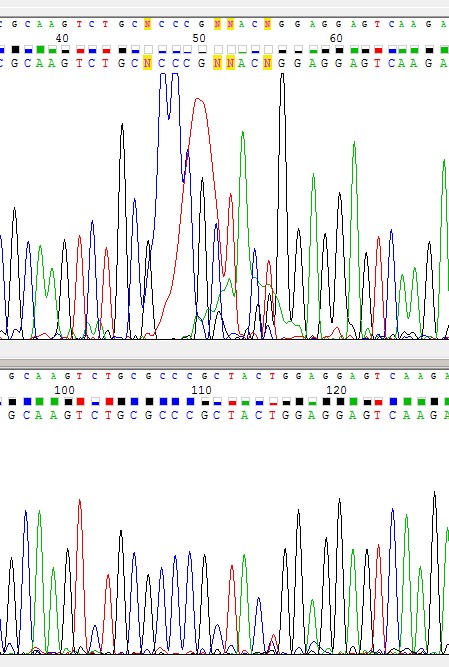
Рис.2 Проблемный участок на прямой цепи в области 45-55. Прямая цепь сверху, обратная снизу.
Пики накладываются друг на друга, также слишком силен шум. К счастью, на обратной цепи проблем в этом месте нет, и последовательность полностью восстанавливается. Хотя на обратной цепи T 155 (на прямой цепи это N 55) имеет плохой quality value из-за слабого сигнала.
Проблема №2. Сильный шум или наложение пиков в позиции 76?
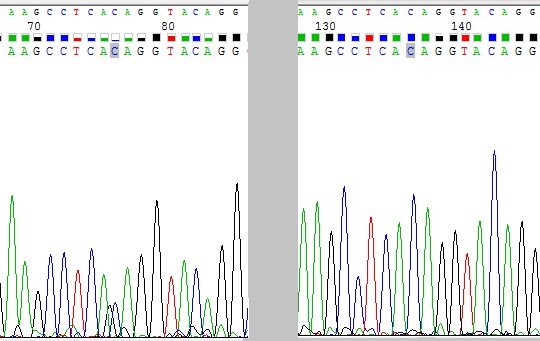
Рис.3 Неоднозначное место на прямой цепи в позиции 76. Слева прямая цепь, справа обратная.
Обратная цепь позволяет понять, что в позиции 76 на прямой цепи должен стоять C и наложения пиков там нет, просто сильный шум. Программа прочитала последовательность правильно.
Аналогичная проблема была в позиции 175, но там из-за сильного шума программа вообще не смогла прочитать нуклеотид и поставила N. Согласно обратной цепи, N было исправлено на t.
Проблема №3.
Слабый сигнал на фоне сильного шума в области, не представленной на обратной цепи.
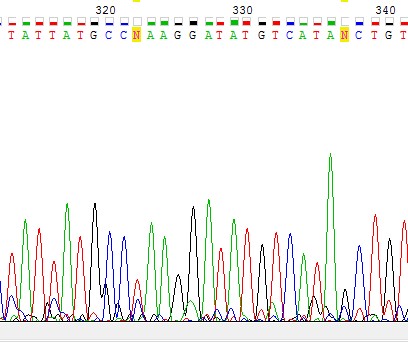
Рис.4 В позициях 322 и 337 слабый сигнал на фоне сильного шума на прямой цепи.
С обратной цепью эти позиции сравнить нельзя, потому что прочтение кончается раньше. Программа поставила в обоих случаях N, но мне кажется, что вполне можно распознать, какой из пиков - это сигнал, а какой шум. Так что я поставила определенные буквы.
Редактирование обратной цепи
Проблема №1. Сильный шум в области, не продублированной прямой цепью.
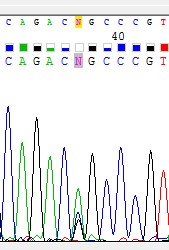
Рис.5 Сильный шум на фоне слабого сигнала в позиции 37.
Программа не смогла распознать нуклеотид. Я оставила N, поскольку шум представлен сразу несколькими высокими пиками, и мне кажется, что прочитать однозначно нельзя.
Проблема №2. Еще одна "клякса".
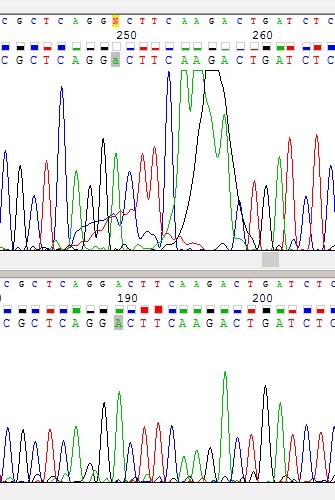
Рис.6 Проблемная область на обратной цепи в районе 250-260. Наверху обратная цепь, внизу прямая.
В области 250-260 пики имеют большую высоту и накладываются друг на друга. Большую часть этой области программа прочитала правильно. Сравнение с прямой цепочкой позволило полностью восстановить последовательность.
Проблема №3. И еще одна "клякса".
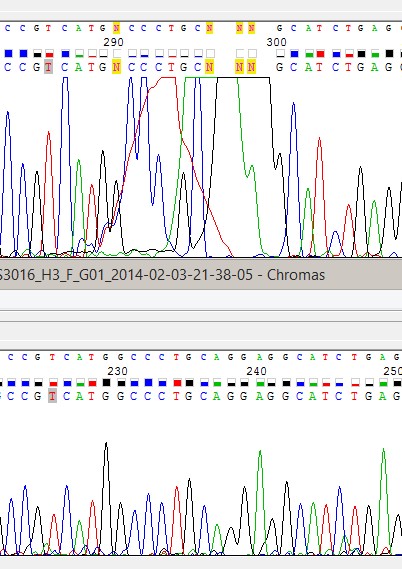
Рис.6 Проблемная область на обратной цепи в районе 290-300. Наверху обратная цепь, внизу прямая.
Эта "клякса" располагается приблизительно в той же области, что и аналогичная ей на прямой цепи. Последовательность восстанавливается по прямой цепи.
Задание 2.
2. Привести пример нечитаемой хроматограммы
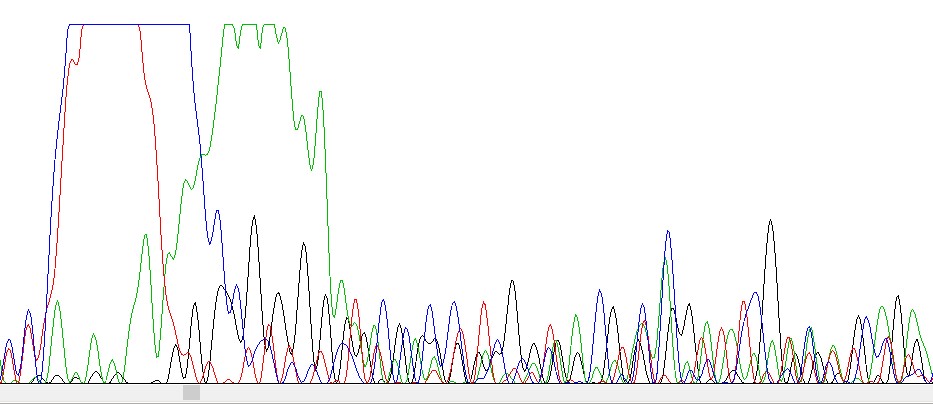 Рис.7 Нечитаемая хроматограмма. Взята из файла .../bad/WSV23_COI_F_A01_WSBS-Seq-1-08-15.ab1 с сервера kodomo.
Рис.7 Нечитаемая хроматограмма. Взята из файла .../bad/WSV23_COI_F_A01_WSBS-Seq-1-08-15.ab1 с сервера kodomo.
Программа не смогла прочитать ни один нуклеотид из данной области, даже не ставила буквы N.
На хроматограмме невозможно разделить шум и сигнал, пики накладываются друг на друга. Прочитать последовательность нельзя.