Практикум 7. Банки нуклеотидных последовательностей
Задание 1. Охарактеризовать качество сборки генома эукариотического организма.
В качестве организма была выбрана Apis mellifera (медоносная пчела), которая имеет большое практическое значение и, соответственно, является предметом многих исследований.

Рис.1 Apis mellifera
Как обстоят дела с секвенированием ее генома:
-
Число сборок генома = 2.
Одна из них завершенна на уровне сборки хромосом (Assembly level: Chromosome). Вторая находится на уровне сборки контигов.
-
Число проектов по секвенированию организма = 2.
-
Число образцов = 2.
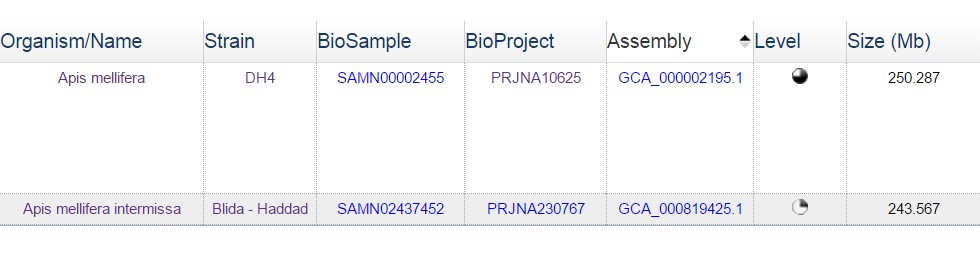
Рис.2 Отчет о сборке генома Apis mellifera(Genome Assembly and Annotation report).
Итак, была выбрана одна из двух сборок генома пчелы медоносной.
Некоторая информация о сборке:
Assembly ID - GCA_000002195.1
BioSample ID - SAMN00002455
BioProject ID - PRJNA230767
Описание образца (BIOSAMPLE ID - SAMN00002455):
Ссылка на страницу BioSample SAMN00002455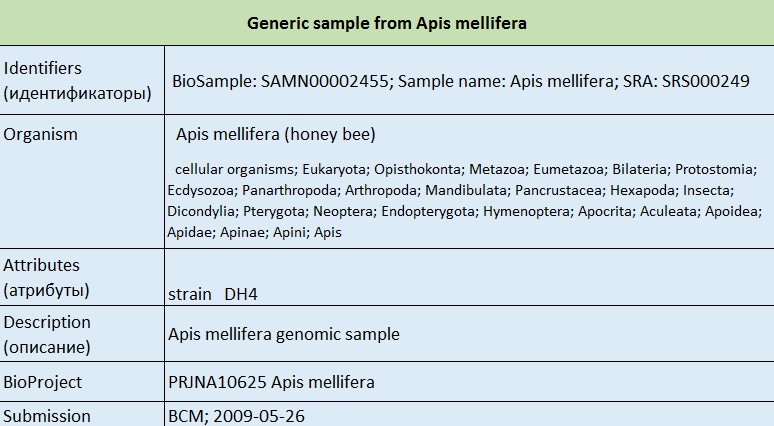
Рис.3 Описание BioSample SAMN00002455 (генетический образец из Apis mellifera).
Описание проекта (BIOPROJECT ID - PRJNA10625):
Ссылка на страницу BioProject PRJNA230767На странице приведена аннотация к проекту, ссылки на связанные с пчелами базы данных и публикации, в которых использованся данный геномный проект.
Текст аннотации (перевод):
"Геном медоносной пчелы, размером около ~200 Mb, был секвенирован командой из Baylor College of Medicine с использованием «метода дробовика» (Whole Genome Shotgun, WGS). Секвенированная последовательность, сборка генома и ее анализ были опубликованы в Nature в октябре 2006 года. Ссылки на публикацию в Nature, других журналах и иных ресурсах можно найти на Honey Bee Genome Publication and Press Portal".
Также на странице проекта приведен ряд его параметров:
-
Project Data Type:Genome sequencing; Locus Tag Prefix:AMEL.
-
Attributes:Scope: Monoisolate; Material: Genome; Capture: Whole; Method type: Sequencing.
Параметры сборки (Assembly GCA_000002195.1):
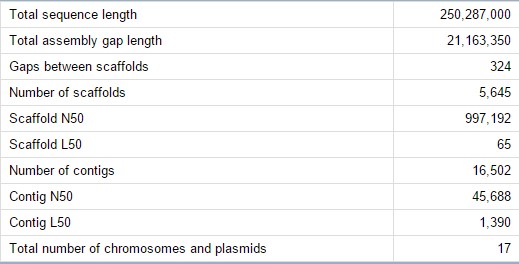
Рис.4 Параметры сборки генома пчелы медоносной(Assembly GCA_000002195.1).
N50 = 45,688 (т.е. не менее половины генома покрывается контигами, длина которых не меньше значения N50).
L50 = 1,390 (число контигов, покрывающих собой не менее половины генома, т.е. длиной не меньше значения N50).
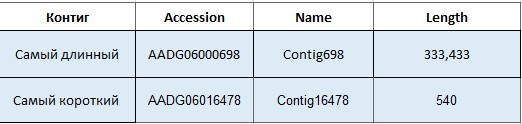
Параметры некоторых контигов:
Задание 2. Составление таблицы митохондриальных генов указанного мха.
Данный мне вид мха - Funaria hygrometrica.

Рис.5 Funaria hygrometrica
Требовалось найти полный геном митохондрий указанного мха в базе данных Nucleotide (NCBI), для чего нужно было составить поисковый запрос. Результат:
((Funaria hygrometrica[Organism]) AND mitochondrion) AND complete genome
По этому запросу было найдено две записи. Обе удовлетворяли требованиям :
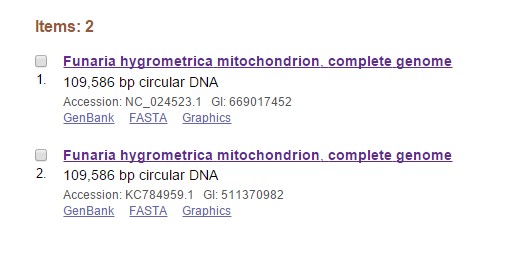
Рис. 6 Записи, найденные по вышеуказанному запросу
Первая запись лежит в NCBI, вторая - в GenBank. Судя по одинаковому названию исследования и авторам, обе записи содержат результаты одного и того же секвенирования. В комментариях к описанию записи в NCBI говорится, что референсная последовательность совпадает с приведенной в GenBank. Запись в NCBI датируется более поздним временем, поэтому я выбрала для дальнейшей работы именно ее.
Далее нужно было получить список всех генов и информацию об их количестве. Для этого я перешла по ссылкам Genome и Gene в разделе Related information.
Результаты:

Рис. 7 Информация о данном геноме и количестве генов в нем.
Как видно из таблицы, в геноме митохондрий Funaria hygrometrica присутствует 67 генов, из которых 40 кодируют белки, 3 - рибосомальные РНК и 24 - тРНК.
Чтобы получить таблицу генов, отсортированных по положению на хромосоме, я скачала искомый файл на странице Gene, предварительно отсортировав (Sort by Chromosome).
Ссылка на таблицу митохондриальных генов Funaria hygrometrica
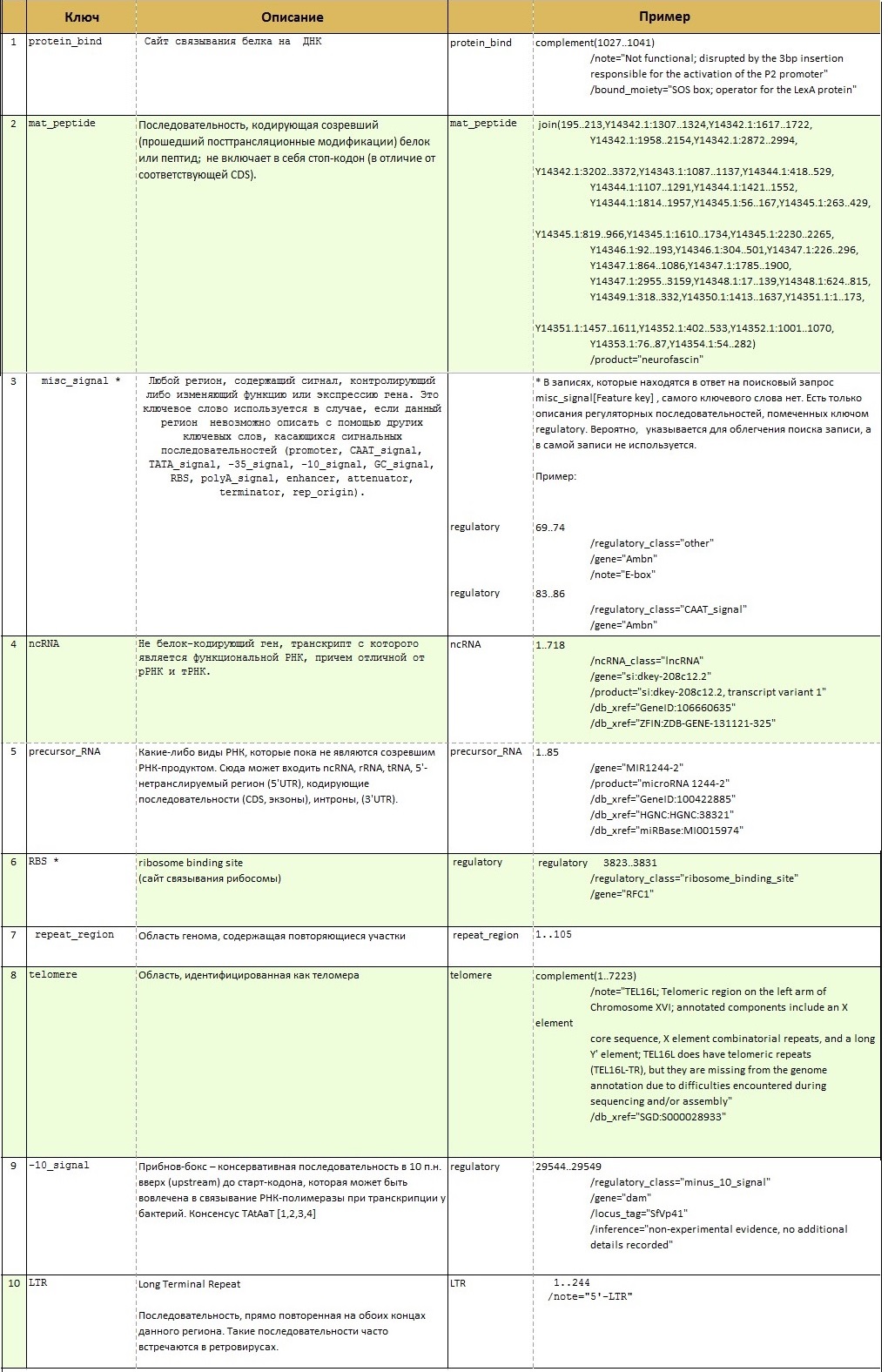
Задание 3. Опиcать десять ключей, используемых в таблицах особенностей.
Для получения ключей и их описания я воспользовалась таблицей особенностей (feature table) с сайта INSDC (The International Nucleotide Sequence Database Collaboration).
Ссылка на таблицу особенностей с сайта INSDC
Результаты:
Задание 4. Установить, какому гену принадлежит последовательность, полученная в практикуме 6, и таксономию организма.
Для выполнения этой задачи использовался BLASTN.
На вход была дана нуклеотидная последовательность, полученная в практикуме 6.
Скачать данную последовательность в формате fasta
Первые несколько результатов из выдачи BLASTN:
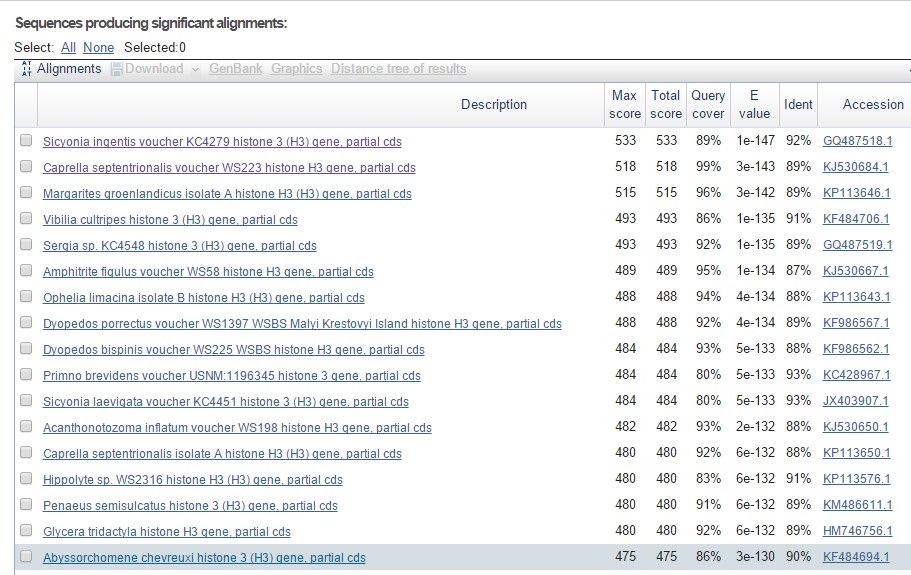
Рис. 8 Несколько первых находок BLASTN по запросу последовательности, полученной в практикуме 6.
Из результатов поиска можно определить ген, которому принадлежит последовательность - это histone 3 (H3) gene, partial cds (ген гистона 3, часть кодирующей последовательности ДНК). Все находки - это части гистона 3.
С определением организма, которому принадлежит данная последовательность, возникли проблемы. Из рисунка 8 видно, что ни одна из находок не идентична заданной более чем на 93%. Далее приведена таблица с характеристиками нескольких лучших находок:
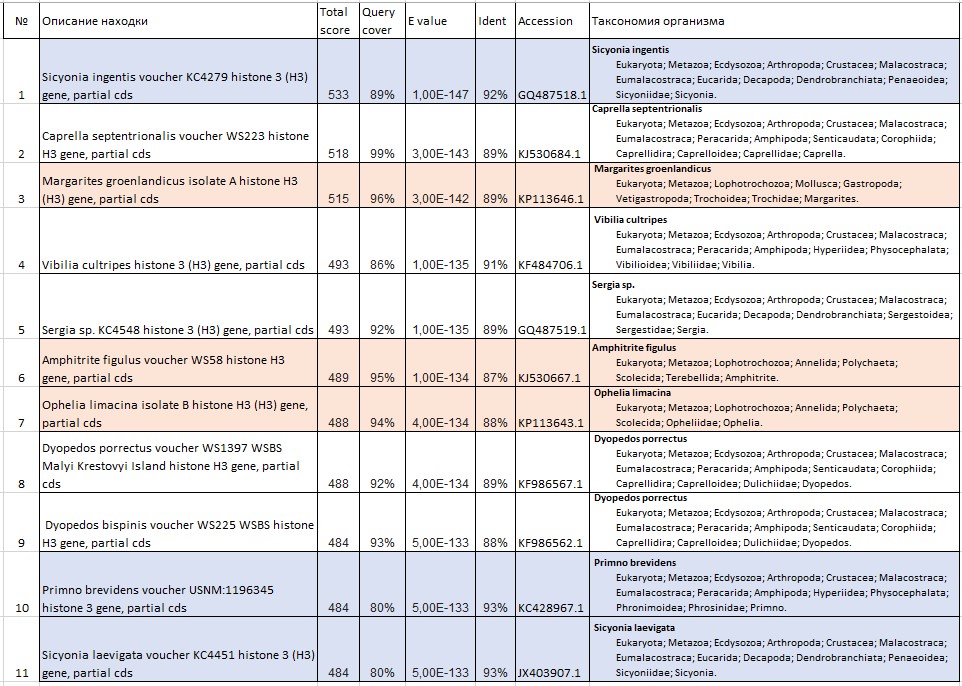
Рис. 9 Несколько первых находок BLASTN с таксономией организмов, которым принадлежат найденные последовательности. Находки отсортированы по E-value. Находки с самой высокой идентичностью данной выделены синим. Красным выделены организмы, принадлежащие к иному классу, нежели чем большинство находок.
Также было построено выравнивание нашей последовательности с 6-ю, у которых самые высокие значения идентичности . Ссылка на выравнивание
В данном выравнивании последовательности отсортированы по попарному сходству.
Рассмотрим 11 лучших находок. Все они имеют очень низкое значение E-value (самое высокое - 5E-133), то есть ни одна из них не случайна. Ни одна из находок не сходна с нашей последовательностью более чем на 93%, поэтому нельзя утверждать, что наша последовательность принадлежит организму какого-либо из найденных видов.
8 из 11 находок принадлежат организмам с таксономией Eukaryota; Metazoa; Ecdysozoa; Arthropoda; Crustacea; Malacostraca; Eumalacostraca, то есть это ракообразные подкласса Eumalacostraca. Одна из оставшихся принадлежит моллюску Margarites groenlandicus, еще две - многощетинковым кольчатым червям Ophelia limacina и Amphitrite figulus. Эти три находки имеют меньшее значение идентичности, чем остальные из лучших 11-ти.

Рис. 10 Организмы, которым принадлежат некоторые из найденных последовательностей.
Мне кажется, из всего вышесказанного можно заключить, что наш организм принадлежит к Eumalacostraca. Процент идентичности находок и их E-value различаются слишком мало, чтобы можно было выбрать какой-либо из эти таксонов, поэтому я остановлюсь на уровне надотряда.
Надо сказать, что в число 11 лучших находок попали 2 организма из одного рода - креветки Sicyonia (находки 1 и 11). Эти находки имеют самую большую идентичность с нашей последовательностью - 92% и 93% (8 и 7 нуклеотидных замен на 100 п.н.), соответственно. Это может означать, что наш организм может принадлежать к этому же роду. Но, мне кажется, свидетельств в пользу этого недостаточно.
Итак, можно заключить, что:
1) аша последовательность является частью гена гистона 3.
2) с высокой вероятностью она принадлежит организму из таксономических групп Eukaryota; Metazoa; Ecdysozoa; Arthropoda; Crustacea; Malacostraca; Eumalacostraca. Таксон более низкого уровня с уверенностью определить нельзя.