Практикум 1. Работа с филогенетическим деревом нескольких бактерий
Я выбрала следующие бактерии:
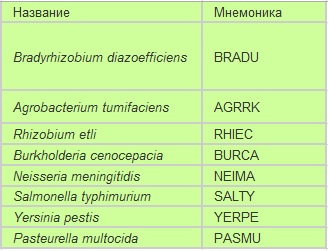
1.Cкобочная формула дерева
((BRADU,(AGRRK,RHIEC)),((BURCA,NEIMA),((SALTY,YERPE),PASMU)));2. Изображение дерева

Рис.1
3. Список нетривиальных ветвей как разбиений множества листьев
1){BRADU, AGRRK, RHIEC, BURCA, NEIMA, PASMU} vs{SALTY, YERPE}
2){BRADU, AGRRK, RHIEC, BURCA, NEIMA} vs {SALTY, YERPE, PASMU}
3){BRADU, AGRRK, RHIEC, SALTY, YERPE, PASMU} vs{BURCA, NEIMA}
4) {BRADU, AGRRK, RHIEC} vs {BURCA, NEIMA, SALTY, YERPE, PASMU}
5){AGRRK, RHIEC} vs {BRADU, BURCA, NEIMA, SALTY, YERPE, PASMU}
4. Из практикума 2:
Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, требовалось определить, к каким таксонам относятся выбранные бактерии.
Результат:


Рис.2 Филогенетическое дерево выбранных бактерий. Нетривиальные ветви соответствуют таксонам с теми же номерами.
1 - Alphaproteobacteria; Rhizobiales
2 - Rhizobiaceae; Rhizobium/Agrobacterium group
3 - Betaproteobacteria
4 - Gammaproteobacteria
5 - Enterobacteriales; Enterobacteriaceae
5. Из практикума 3: yкоренение в среднюю точку
Требовалось укоренить в среднюю точку дерево, построенное при выполнении задания 4 предыдущего занятия (методом Neighbor-Joining using % identity), рис.3. Метод Neighbor-Joining строит неукорененные деревья.

Рис.3 Филогенетическое дерево данных бактерий, построенное по выравниванию рибосомных белков S4 методом Neighbor-Joining using % identity.
Для этого нужно было воспользоваться программой retree пакета PHYLIP.
Результат:
Рис.4 Дерево, полученное в результате укоренения в среднюю точку дерева с рисунка 3

Вывод: при укоренении в среднюю точку получилось дерево, топология которого совпадает с топологией правильного филогенетического дерева данных бактерий (рис.2).