Практикум 11. Поиск сигналов
Задание 3. Определение сайтов связывания данного транскрипционного фактора в данном участке хромосомы человека
Я работала с файлом chipseq_chunk4.fastq.
Ход работы:
1. Проверка качества ридов с помощью программы FastQC
Вместо файла chipseq_chunk55.fastq я взяла один из запасных, chipseq_chunk4.fastq, поскольку в старом качество ридов было довольно плохим (около 28) по всей длине рида. В файле chipseq_chunk4.fastq качество ридов хорошее(рис.1), и фильтрация не требуется.
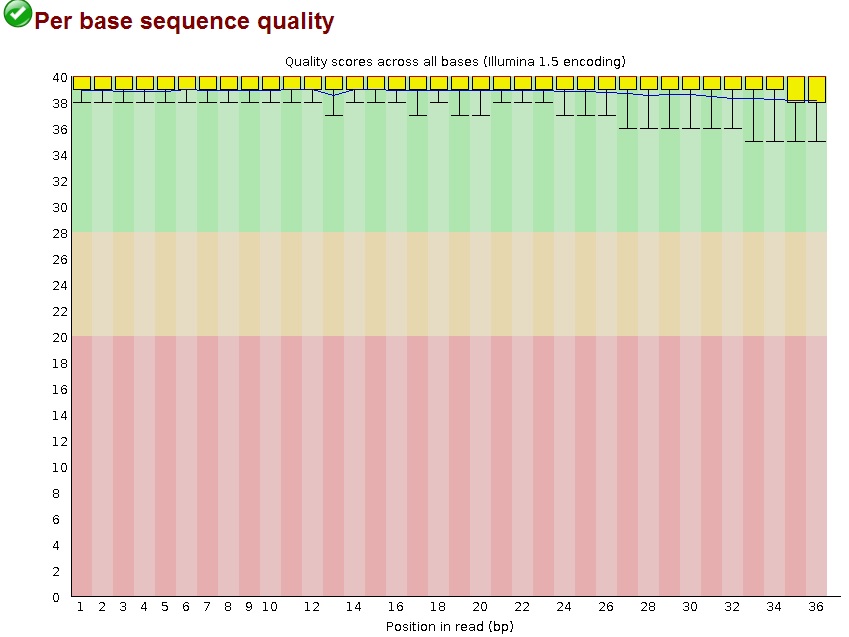
Рис.1 Per base quality (качество определения основания в каждой позиции рида) для ридов из файла chunk4
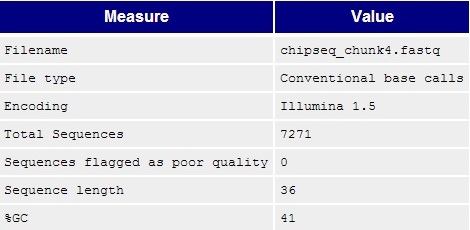
Рис.2 Основная информация о файле с чтениями
2. Картирование прочтений на проиндексированный геном человека hg19 и анализ результатов
Далее я последовательно выполняла команды из таблицы 1:
Таблица 1. Использовавшиеся в ходе работы команды:
Всего откартировалось 7271 чтения, то есть все.
Результаты картирования приведены в таблице 2:
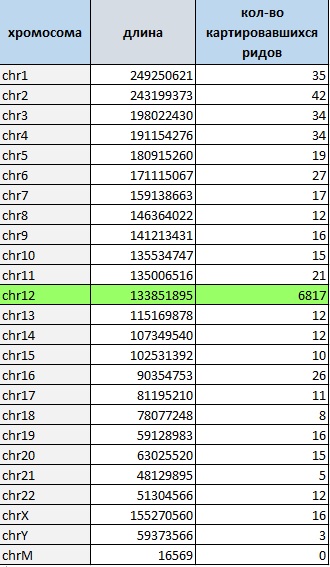
По результатам видно, что были предложены чтения с 12-ой хромосомы. На каждую хромосому, кроме митохондриальной, картировалось не менее 3-х чтений.
Поиск пиков (peak calling) с помощью программы MACS
Процедура поиска пиков:
1. Сначала я попробовала запустить команду macs2 callpeak -t chipseq_chunk4.sorted.bam, но программа выдала, что пиков слишком мало.
Тогда я воспользовалась программой macs2 callpeak -t chipseq_chunkX.sorted.bam --nomodel -n MACS
Опция -n позволяет задавать имя эксперимента.
Результатом работы программы являются три файла:
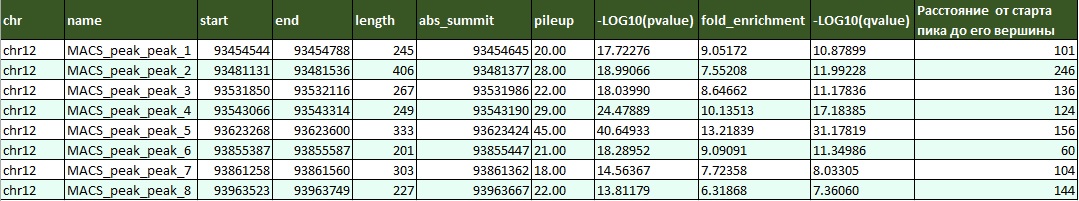
Было найдено 8 пиков. Все они расположены в одном регионе 12-й хромосомы - около 90 млн п.н. Ширина пиков варьируется от 201 до 406 нуклеотидов. На вид величина p-value (а значит, и достоверность) не зависит от ширины пика (см. таблицу 3). У среднего по длине пика (№5) самый маленький p-value, у остальных пиков он значительно меньше, в том числе у более длинных. Отсутствие зависимости p-value от длины пика наблюдается, даже если счесть пик №5 выбросом. Расположение вершин пиков относительно их крайних точек указано в таблице 1. В целом, вершина в большинстве случаев располагается примерно посередине пиков. Исключением является пик 6, вершина которого находится примерно на границе первой и второй его четвертей.
Таблица 3. Результаты поиска пиков программой MACS
Результаты визуализации пиков:
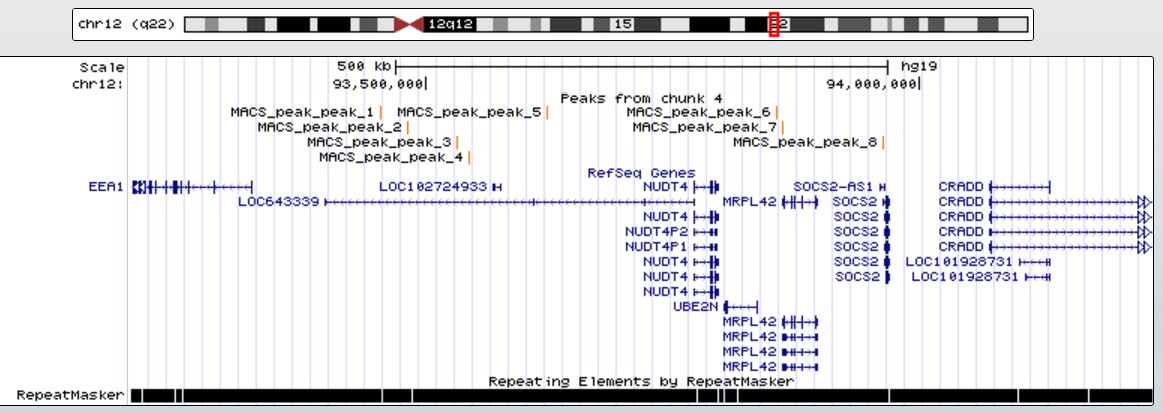
Рис. 3 Окно геномного браузера со всеми пиками из файла MACS_peaks.narrowPeak
Я использовала UCSC Genome Browser.
Расположение пиков в геноме (рисунок 3). Пики 1-5 перекрываются с длинной некодирующей РНК LOC643339. Пик 6 расположен перед началом (за 6 000 п.н. до) гена MRPL42 (mitochondrial ribosomal protein L42), пик 7 - в начале этого же гена, пик 8 - в начале гена suppressor of cytokine signaling 2 (SOCS2).
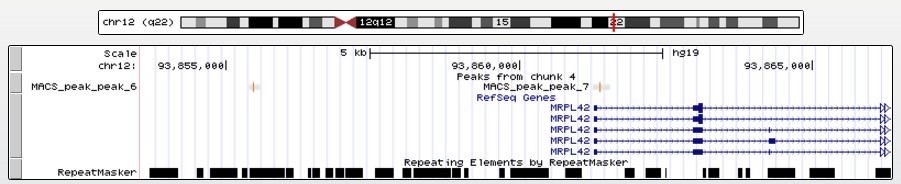
Рис.4 Расположение пиков 6 и 7 в геноме.
Более подробное описание пиков
Расмотрим поподробнее пик №7. Он находится в начале гена MRPL42 (mitochondrial ribosomal protein L42).
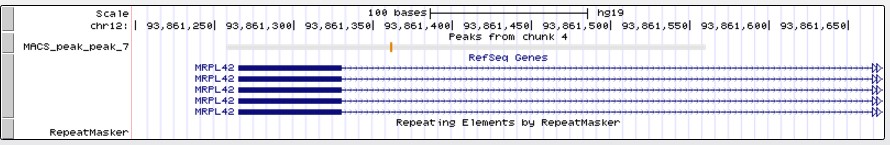
Рис.5 Расположение пика №7 в геноме.
В окне геномного браузера я включила отображение регуляторных элементов из ENCODE project. Результаты приведены на рисунке 6. Видно, что в области, где располагается пик 7, находятся сайты связывания множества регуляторных факторов, найденные с помощью ChIP-seq.
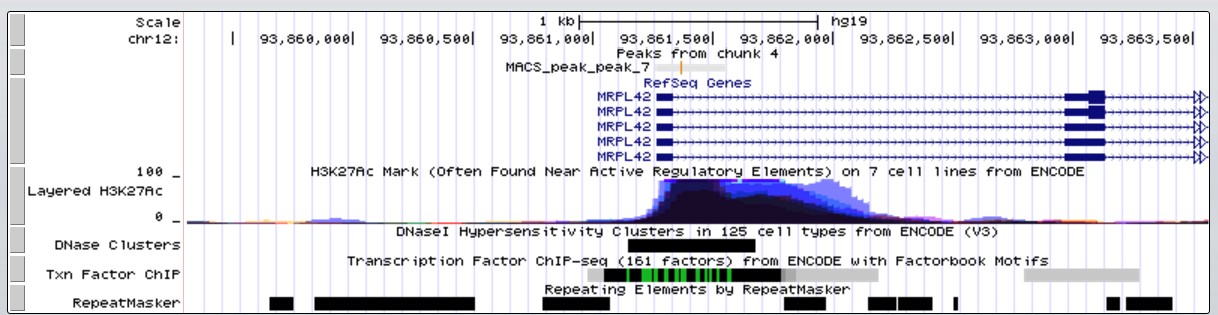
Рис.6 Расположение пика №7 в геноме и регуляция гена MRPL42.
На первый взгляд кажется, что пик №6 располагается слишком далеко от начала гена MRPL42 (за 6 000 п.н.), чтобы там располагались сайты регуляции этого гена. Однако, судя по данным из ENCODE, в этой области расположены сайты связывания сразу нескольких транскрипционных факторов.
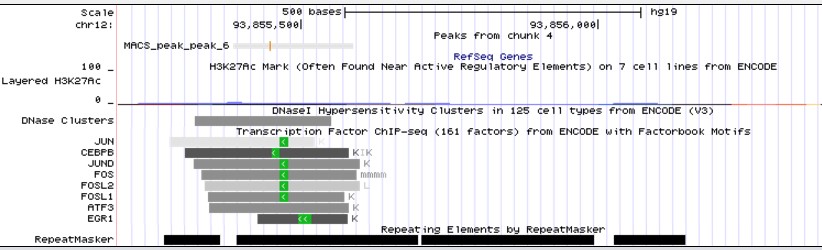
Рис.7 Расположение пика №6 в геноме и регуляторные элементы.
Рассмотрим также пик 5. У него самый маленький p-value из всех найденных пиков - около 10^(-40), что говорит о его высокой достоверности. На рисунке 8 изображено расположение пика №5 в геноме. Он попадает в область второго интрона длинной некодирующей РНК LOC643339.
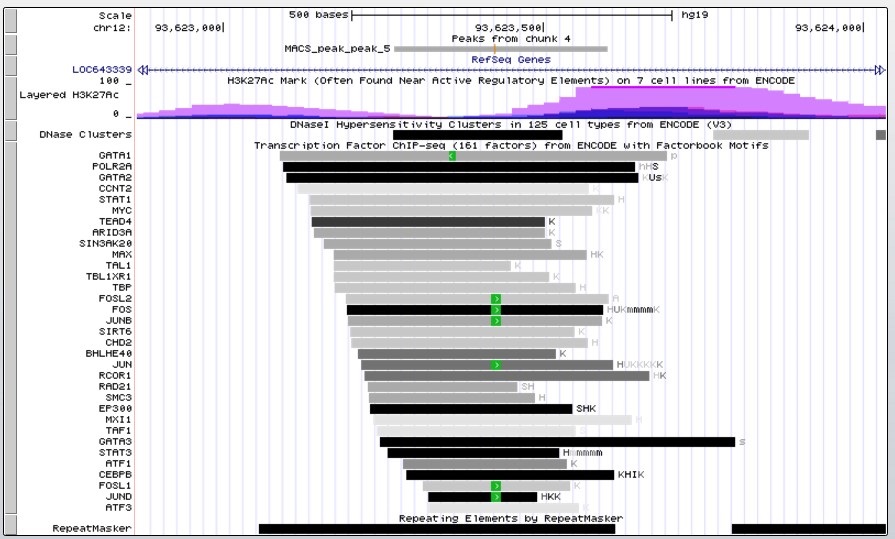
Рис.7 Расположение пика №5 в геноме и регуляторные элементы.
Пик №8 расположен рядом с геном suppressor of cytokine signaling 2 (SOCS2), причем перекрывается с одним из альтернативных транскриптов этого гена, а также длинной некодирующей антисмысловой РНК (Homo sapiens SOCS2 antisense RNA 1 (SOCS2-AS1)). По-видимому, эта РНК является альтернативным продуктом с этого участка и экспрессируется в результате регуляции транскрипции гена SOCS2).
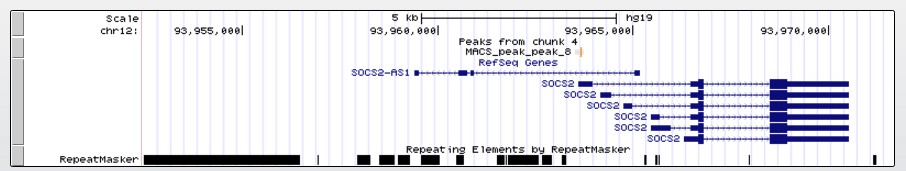
Рис.8 Расположение пика №8 в геноме.
Судя по расположению сайтов связывания на минус-цепи (рис. 9), регуляторные факторы из пика 8 функционально связаны с экспрессией SOCS2 antisense RNA 1, которая тоже располагается на минус-цепи. Хотя я не уверена, что между расположением гена на определенной цепи и расположением сайта связывания его регуляторного фактора есть прямая зависимость.

Рис.9 Расположение пика №8 в геноме и близлежащие регуляторные элементы.
Задание 4. Поиск сайтов связывания TBP
Требовалось найти в геноме человека три гена, транскрипция которых инициируется с помощью TATA-бокс связывающего белка, и один - без сигнала TATA-бокса в промоторной области.
Консенсусной последовательностью ТАТА-бокса является 5'-TATAAA-3'. Прочитать обзор, посвященный полиморфизму ТАТА-бокса в человеческом геноме, можно здесь: http://protein.bio.msu.ru/biokhimiya/contents/v74/full/74020149.html .

Рис.11 LOGO ТАТА-бокса в человеческом геноме. Рисунок позаимствован с сайта http://www.cbs.dtu.dk/
Чтобы найти ТАТА-боксы в последовательностях генов, требовалось воспользоваться UCSC GenomeBrowser , в котором отображаются результаты экспериментов по иммунопреципитации хроматина (ChIP), связанного с белком TBP (транскрипционный фактор, связывающий ТАТА-бокс и являющийся частью комплекса инициации транскрипции у эукариот). Для этого на странице геномного браузера с необходимыми настройками был выбран TPB и один из экспериментов. Я выбрала тот, в котором использовались мышиные поликлональные антитела (рис. 10).

Рис.11 Параметры выбранного эксперимента
Результаты:
Ген G6PC3
Название : glucose 6 phosphatase catalytic subunit 3
This gene encodes the catalytic subunit of glucose-6-phosphatase (G6Pase). G6Pase is located in the endoplasmic reticulum (ER) and catalyzes the hydrolysis of glucose-6-phosphate to glucose and phosphate in the last step of the gluconeogenic and glycogenolytic pathways. Mutations in this gene result in autosomal recessive severe congenital neutropenia. Alternative splicing results in multiple transcript variants. [provided by RefSeq, Feb 2016].
Положение в геноме: chr17:42148098-42153712, +
Координата старта транскрипции: chr17:42148098
Длина 6515 п.н.
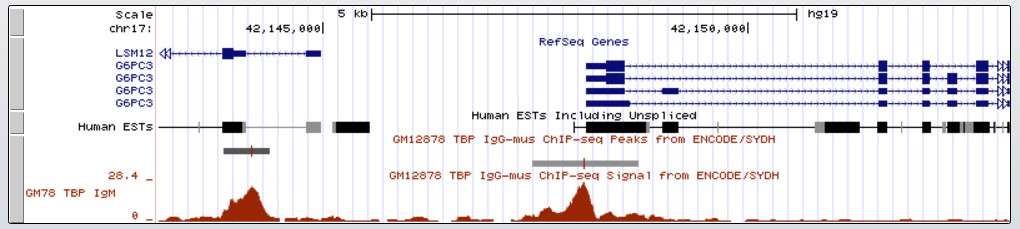
Рис. 11 Изображение положения гена G6PC3. Виден пик в его промоторном регионе.
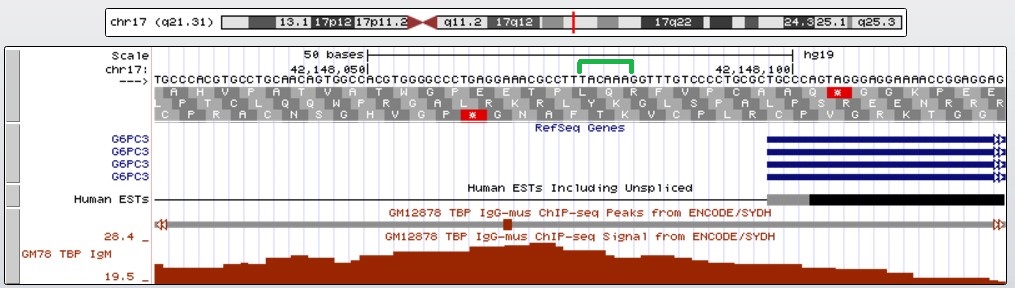
Рис. 12 То же в нуклеотидном разрешении. Видна последовательность ТАТА-бокса, близкая к консервативной (подчеркнута зеленым).
Обсуждение
Найденный пик хорошо отличается от шума и имеет довольно большую силу, что свидетельствует о его достоверности. Действительно, в промоторной области гена была найдена последовательность TACAAA примерно за 20 п.н. до старта транскрипции.
Вероятно, этот ген имеет сильный промотор (о чем свидетельствует близкий к консервативному ТАТА-бокс). Возможно, причина этого состоит в том, что глюкозо-6-фосфатаза является ферментом глюконеогенеза и должна присутствовать во многих клетках в большом количестве.
Ген CNTN6
Название: Homo sapiens contactin 6
The protein encoded by this gene is a member of the immunoglobulin superfamily. It is a glycosylphosphatidylinositol (GPI)-anchored neuronal membrane protein that functions as a cell adhesion molecule. It may play a role in the formation of axon connections in the developing nervous system. Alternative splicing results in multiple transcript variants. [provided by RefSeq, Jan 2014].
Положение в геноме: chr3:1134342-1445292, +
Координата старта транскрипции: chr3:1134342
Длина - 310951 н.
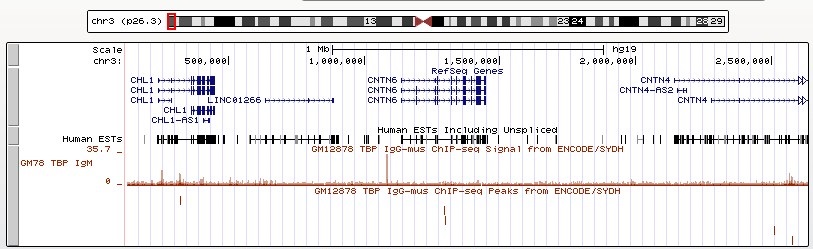
Рис. 13 Изображение геномного окружения CNTN6. Виден высокий пик далеко upstream и отсутствие сильных сигналов в области гена CNTN6.
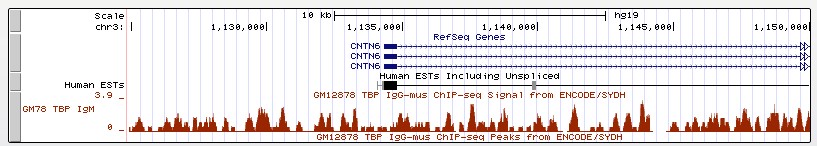
Рис. 14 То же более крупно. Виден высокий уровень шума в промоторной области гена CNTN6 и отсутствие достоверных пиков.
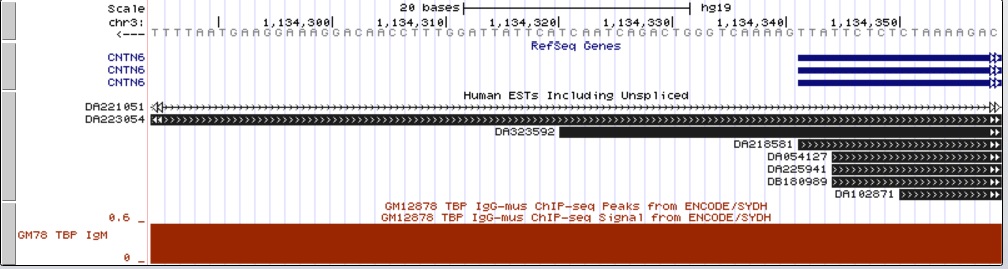
Рис. 15 Промоторная область CNTN6 в нуклеотидном разрешении. Последовательности ТАТА-боксов отсутствуют.
Обсуждение
По-видимому, в промоторе этого гена нет ТАТА-бокса. Об этом свидетельствует отсутствие пиков в промоторной области и, собственно, отсутствие последовательности ТАТА-бокса в +- 200 нуклеотидах от старта транскрипции (я искала и не нашла). Надо сказать, что начало гена представляет из себя AT-богатую последовательность. Также в промоторном регионе присутствуют несколько тринуклеотидов ТАТ (думаю, они и обеспечивают высокий уровень шума).
Ген ITPR1
Название : inositol 1,4,5-trisphosphate receptor type 1
This gene encodes an intracellular receptor for inositol 1,4,5-trisphosphate. Upon stimulation by inositol 1,4,5-trisphosphate, this receptor mediates calcium release from the endoplasmic reticulum. Mutations in this gene cause spinocerebellar ataxia type 15, a disease associated with an heterogeneous group of cerebellar disorders. Multiple transcript variants have been identified for this gene.
Положение в геноме: chr3:4535032-4889524 +
Координата старта транскрипции: chr3:4535032
Длина 354493
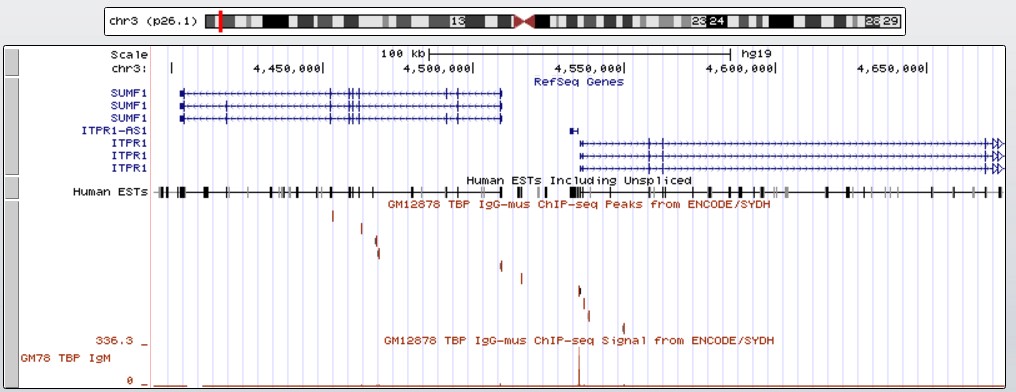
Рис.16 Изображение положения гена ITPR1. Виден высокий пик в его промоторном регионе.
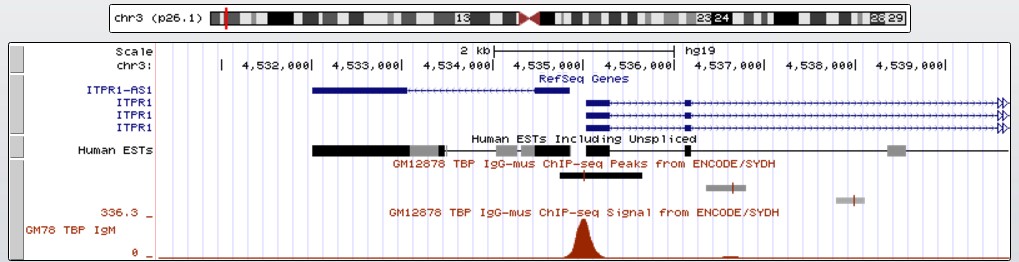
Рис.17 То же с большим увеличением.
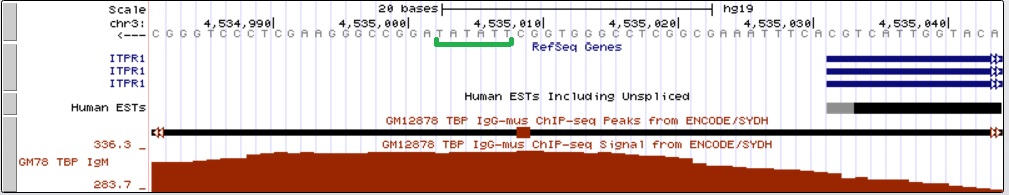
Рис.18 Промоторный регион гена ITPR1. Предполагаемая последовательность ТАТА-бокса подчеркнута зеленым.
Обсуждение
Пик связывания антител в промоторной области данного гена очень высокий (336.3, рис. ??), на два порядка выше уровеня шума. Это свидетельствует о его высокой достоверности и, возможно, последовательности ТАТА-бокса, близкой к консенсусной. Действительно, прмерно за 30 п.н. до старта транскрипции есть последовательность ТАТАТТ. Там же находится вершина пика. Можно заключить, что там действительно находится ТАТА-бокс. Хотя в запис данного гена в БД Nucleotide ( http://www.ncbi.nlm.nih.gov/nuccore/NM_001168272?report=GenBank ) об этом ничего не сказано.
Ген GH1
Название : Homo sapiens growth hormone 1
The protein encoded by this gene is a member of the somatotropin/prolactin family of hormones which play an important role in growth control. The gene, along with four other related genes, is located at the growth hormone locus on chromosome 17 where they are interspersed in the same transcriptional orientation; an arrangement which is thought to have evolved by a series of gene duplications. The five genes share a remarkably high degree of sequence identity. Alternative splicing generates additional isoforms of each of the five growth hormones, leading to further diversity and potential for specialization. This particular family member is expressed in the pituitary but not in placental tissue as is the case for the other four genes in the growth hormone locus. Mutations in or deletions of the gene lead to growth hormone deficiency and short stature. [provided by RefSeq, Jul 2008].
Положение в геноме: chr17:61994553-61996212, -
Координата старта транскрипции: chr17:61994553
Длина 1660
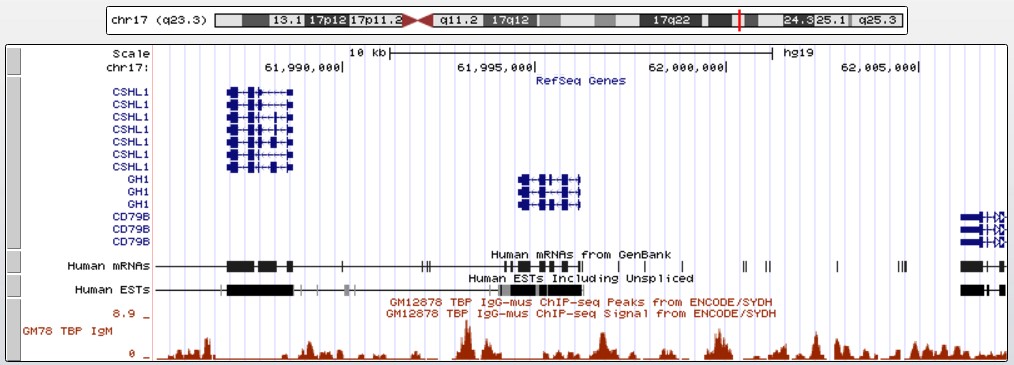
Рис. 19 Положение гена GH1 в геноме. Видно, что в данном регионе сильный шум, и сигнал периодически обрывается.
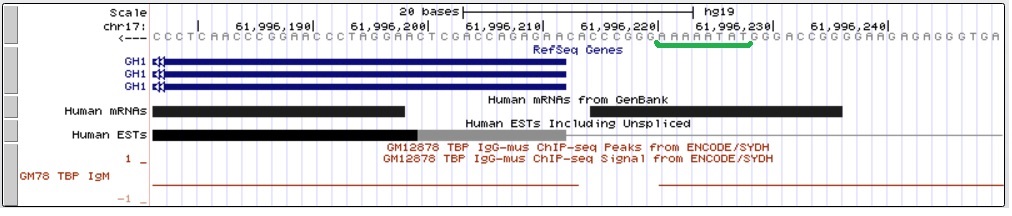
Рис. 20 Промоторный регион гена GH1. Присутствует последовательность ТАТА-бокса (подчеркнута зеленым). Пики отсутствуют. Видно, что сигнал обрывается.
Обсуждение
В промоторе данного гена присутствует ТАТА-бокс, я узнала об этом из обзора, посвященного полиморфизмам ТАТА-бокса (ссылка в начале отчета по заданию 4). Однако в выбранном мной эксперименте ChIP эта последовательность не была найдена. Сигнал в этом месте крайне плохой, периодически обрывается и имеет высокий уровень шума. Стоит сделать вывод, что результаты экспериментов не всегда надежны, и лучше искать пики сразу по нескольким экспериментам.