Практикум 4.
Я работала со следующими бактериями:
1. Построение дерева по нуклеотидным последовательностям
Требовалось построить филогенетическое дерево (тех же бактерий, что в предыдущих заданиях), используя последовательности РНК малой субъединицы рибосомы (16S rRNA).
Получение последовательностей.
Последовательности были получены из базы полных геномов NCBI (ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/). Нужные нам 16S рРНК находится в файле с расширением .frn
Далее последовательности были выровнены в JalView с помощью Muscle. Затем импортированы в MEGA, где по ним было построено дерево методом Maximum likelihood.
Результат:
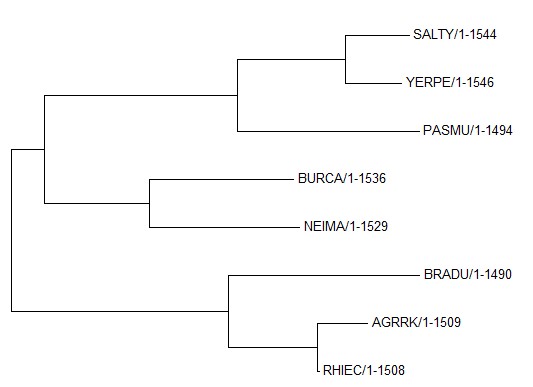
Рис.1 Филогенетическое дерево 16S рРНК данных бактерий, построенное методом ML
Анализ дерева. Дерево полностью совпадает с правильным - филогенетическим деревом самих бактерий. Качество реконструкции по рРНК по сравнению с деревьями, построенными по белкам, однозначно выше, если сравнивать с деревом из задания 2 этого практикума (см.ниже). Деревья, реконструированные по белкам, которые я получала в предыдущих двух практикумах, совпадали с филогенией бактерий. Вообще можно ожидать, что деревья, построенные по рРНК, будут ближе к правде, поскольку последовательности рРНК эволюционно очень консервативны и не претерпевают быстрой эволюции, связанной с расхождением видов, в отличие от некоторых белков.
Задание 2. Построение и анализ дерева, содержащего паралоги
Требовалось найти в своих бактериях достоверные гомологи белка CLPX_ECOLI и построить их филогенетическое дерево.
Чтобы получить последовательности гомологов CLPX_ECOLI, я использовала локальный blastp для поиска против базы из всех полных протеомов восьми моих бактерий. Пороговое значение e-value = 0.001.
Результаты работы blastp:
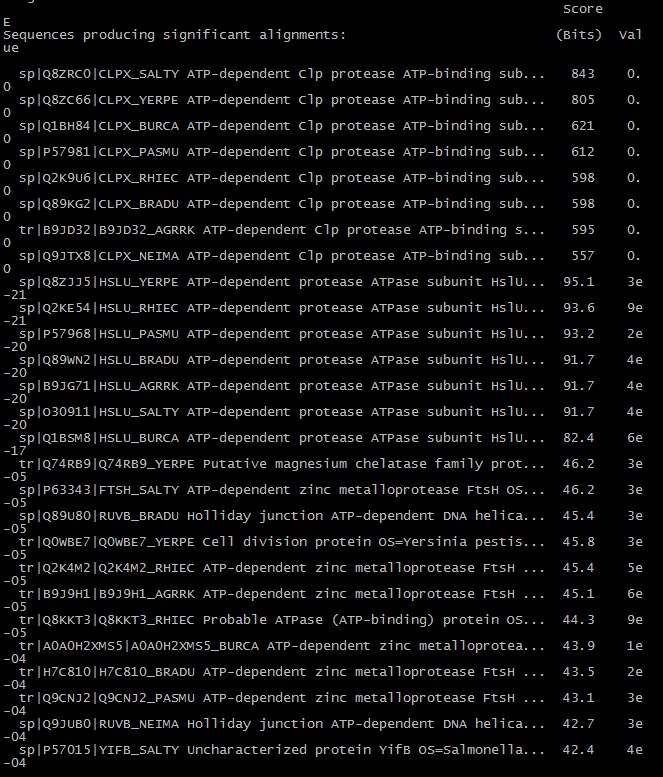
Я получила 27 находок, из которых 8 соответствуют белкам с мнемоникой CLPX (ATP-dependent Clp protease ATP-binding subunit ClpX). Еще 7 белков - это HSLU (ATP-dependent protease ATPase subunit HslU), какая-то другая протеаза, ее функция предложена из гомологии. Остальные белки имеют разные мнемоники.
Результат построения дерева:
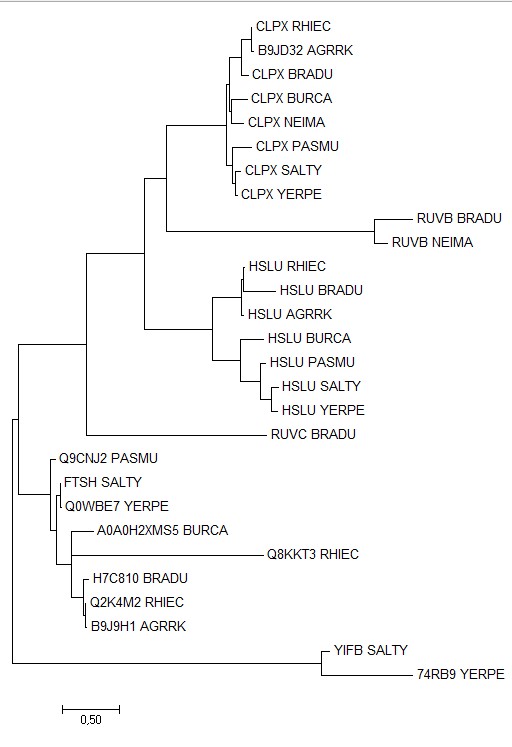
Рис.2 Филогенетическое дерево данных гомологичных белков, построенное методом Maximum likelihood.
Для удобства стоит рассмотреть рисунок, на котором совмещены филогенетическое дерево бактерий (1) и дерево данных белков (2).
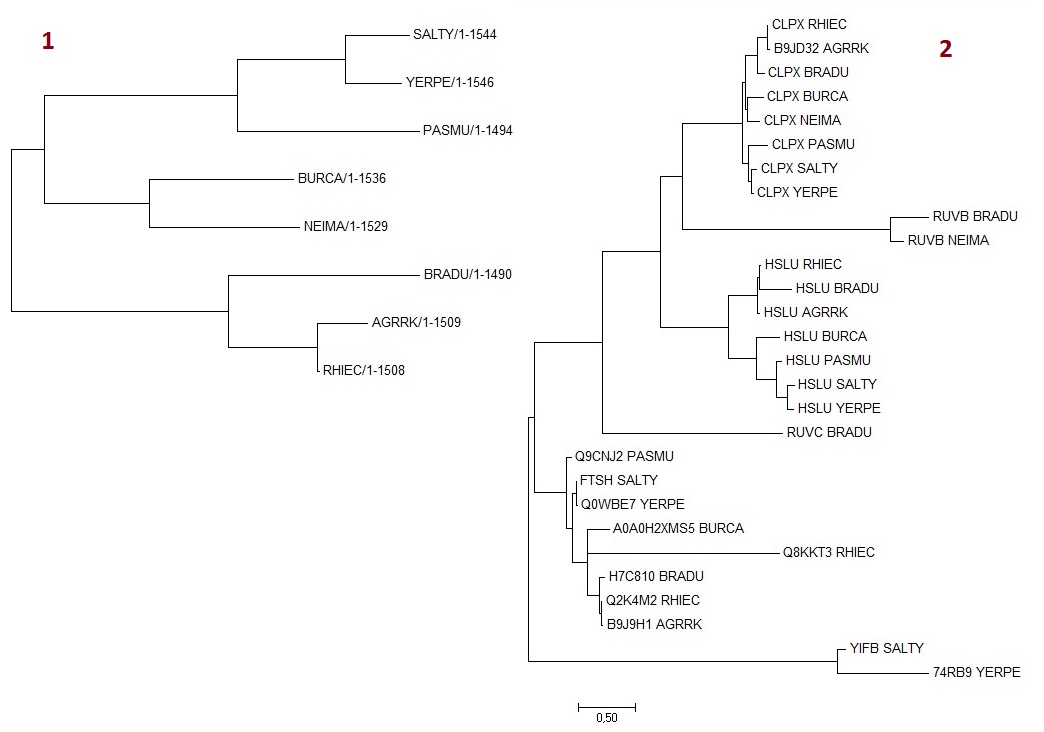
Рис.3
Также я решила провести бутстрэп-анализ полученного дерева, чтобы иметь представление о достоверности ветвей.
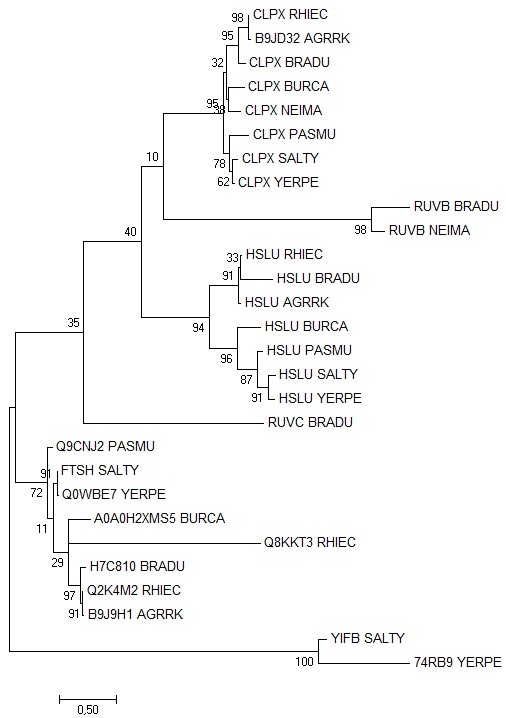
Рис. 4 Филогенетическое дерево данных гомологичных белков, построенное методом Maximum likelihood + бутстрэп-анализ.
Анализ результатов.
Примечание. Два гомологичных белка будем называть ортологами, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралогами.
Во-первых, видно, что белки CLPX всех восьми бактерий выделяются в отдельное дерево (белок с мнемоникой B9JD32_AGRRK имеет то же название, что и остальные CLPX). ПРи этом топология этого поддерева отличается от топологии филогенетического дерева бактерий (см. рис. 3), а именно наличием ветви (((PASMU,(YERPE,SALTY)),(NEIMA,BURCA))). Однако у ветки (((CLPX_RHIEC, B9JD32_AGRRK), CLPX_BRADU), (CLPX_BURCA, CLPX_NEIMA)) дерева белков низкая бутстрэп-поддержка (около 30%, см. рис. 4), так что она, по-видимому, не достоверна.
Во-вторых, у всех бактерий, кроме NEIMA, присутствует белок HSLU, который тоже выделяется в отдельное поддерево. Топология его дерева не совпадает с деревом бактерий внутри ветви ((RHIEC,AGRRK),BRADU)), поскольку, видимо, последовательности HSLU этих трех бактерий успели разойтись очень мало, и их филогению определить трудно. Думаю, бактерия NEIMA белок HSLU потеряла.
Паралоги: CLPX_BRADU и HSLU_BRADU, CLPX_BRADU и RUVB_BRADU, CLPX_YERPE и Q74RB9_YERPE, CLPX_NEIMA и RUVB_NEIMA.
Группы попарно ортологичных белков: 1)CLPX_BRADU,B9JD32_AGRRK, CLPX_YERPE, CLPX_RHIEC, CLPX_BURCA и т.д.
2) Белки с мнемоникой HSLU
3) RUVB_NEIMA и RUVB_BRADU
4)YIFB_SALTY и Q74RB9_YERPE
Дупликация и последующее разделение функций генов CLPX и HSLU произошло, вероятно, у общего предка выбранных восьми бактерий.
Пример разделения путей эволюции белков в результате видообразования присутствует в следующем поддереве:

Рис.5 Фрагмент дерева данных белков с описанием функций
Похоже, что белок Q0WBE7_YERPE изменил свою функцию после отделения от общего предка его и FTSH_SALTY. Ближайший родственник FTSH_SALTY и на дереве, FTSH_SALTY, как и почти все белки этого поддерева, является АТФ-зависимой цинковой металлопротеазой.