Практикум 7.
1. Геномное окружение, SEED
Цель задания: проверить консервативность геномного окружения заданного гена.
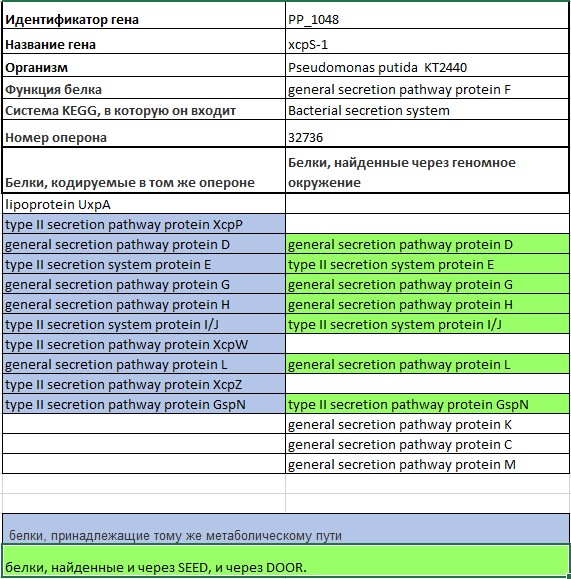
Идентификатор выбранного гена - PP_1048, ген xcpS-1, Gene ID: 1044322, организм Pseudomonas putida KT2440 (Грам-минус, почвенная, непатогенная). Продукт - General secretion pathway protein F.
Для получения картинки я получина вначале 40 гомологов (“Number of regions”), затем убрала похожие. При изменении длины региона новых генов, которые встречались бы больше чем у двух организмов, не нашлось. Также я увеличила параметр “e-value cutoff for coloring CDS sets” до 1e-10, поскольку при значении по умолчанию(1e-20) некоторые гомологи с одинаковыми названиями не красились в один цвет. Часть гомологов в S. boydii не нашлась даже при изменении e-value до 1e-7, поэтому я решила оставить так.
Результат:
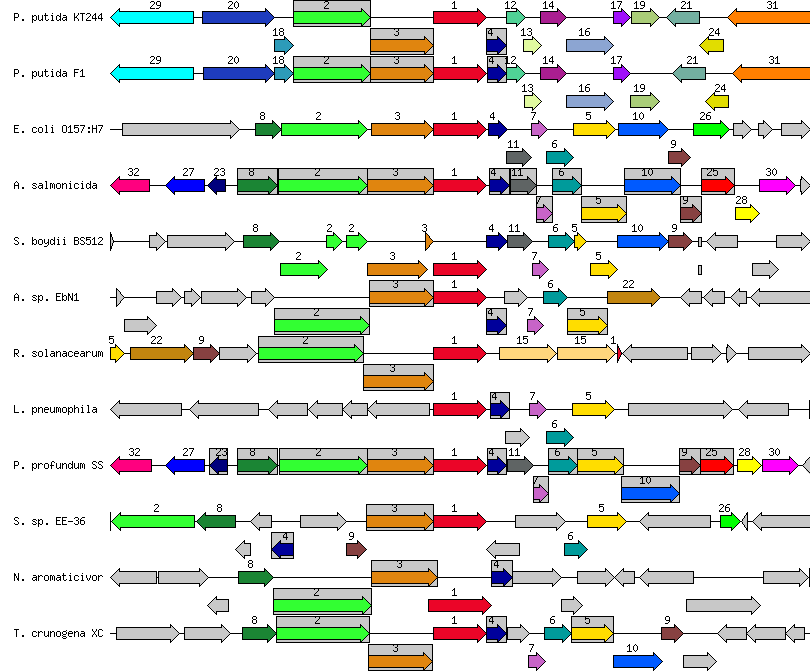
Рис.1 Геномное окружение гена xcpS-1 (general secretion pathway protein F) в разных организмах, полученное с помощью сервиса SEED. Данный ген покрашен в красный цвет. Гены консервативного геномного окружения обведены в рамочку.
Описание:
Данный ген относится к секреторному пути 2 типа (Type II secretory pathway).
-
Гены, появляющиеся в окрестности данного гена почти у всех данных организмов: general secretion pathway protein G general secretion pathway protein E (№3), general secretion pathway protein D (№2), gspK (№5, есть у двух третей); есть у половины: gspJ (№6), gspC (№8), gspL(№10), gspI(№7).
-
У меньшего количества организмов: general secretion pathway protein H (11), I(6), L(10), M (9), N (19, 25) и др. Соответствие генов белкам см. на рис. 3.
У некоторых организмов появляются гены, не относящиеся к консервативному геномному окружению нашего гена, продукты которых являются: белками теплового шока (Ribosome-associated heat shock protein implicated in the recycling of the 50S subunit(12), 33 kDa chaperonin (14), транспозазой, разнообразными ферментами и гипотетическими белками.
К генам консервативного геномного окружения xcpS-1 можно отнести:
2. Опероны, DOOR
Цель задания: найти гены, входящие в один оперон с заданным, и сравнить находки с результатами из SEED
На сайте DOOR я нашла оперон, в который входит заданный ген.
Operon ID: 32736

Рис.2 Изображение данного оперона из БД DOOR
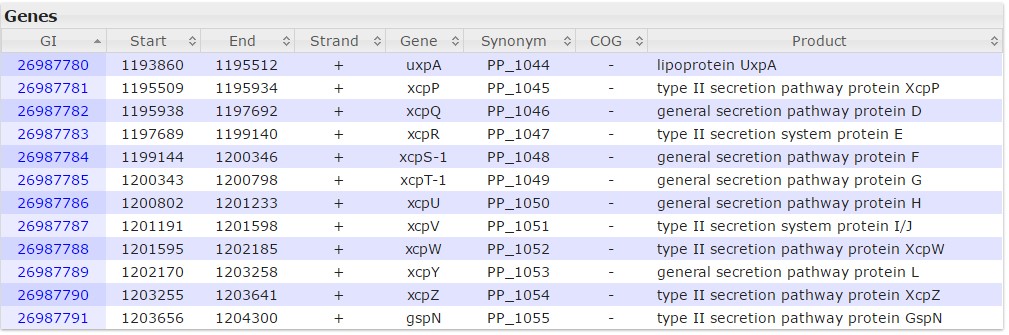
Рис.3 Гены, входящие в данный оперон.
Все эти гены нашлись с помощью SEED у Pseudomonas putida.
Большая часть этих генов входит в константное геномное окружение гена xcpS-1 у других организмов (найденных с помощью SEED). Однако у Pseudomonas putida не нашлось генов, кодирующих белки с названиями, соответствующими gspK, gspM и gspC(входящих в константное геномное окружение исходного гена), зато нашлись гены xcpP, xcpW и xcpZ, отсутствующие у других найденных организмов. Я подозреваю, что продукты этих генов функционально соответствуют gspK, gspM и gspC, поскольку данные из KEGG говорят об участии всех этих белков (gsp) в формировании одного комплекса (см. ниже в разделе про KEGG). Я пробовала поискать в SEED гомологи xcpP, но даже с обоими параметрами e-value 1e-5 с xcpU который соответствует нашлось только три организма с теми же самым названием этого гена. Впрочем, когда я проделала то же самое с gspH, согласно DOOR, результат был аналогичным. Потому не могу сделать никаких выводов.
Ген UxpA, кодирующий предшественник липопротеина, присутствует только у Pseudomonas putida.
3. Метаболические пути, KEGG
Требовалось выяснить, связаны ли найденные белки с заданным функционально. Для этого нужно было найти белок, кодируемый данным геном, в KEGG, и перейти на страницу метаболического пути, в который он входит.
Данный белок входит в метаболический путь Bacterial secretion system, а именно в систему секреции типа (type II secretion system).
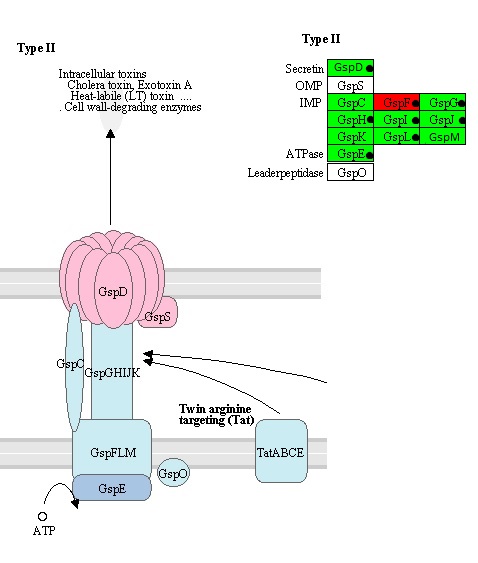
Рис.4 Изображение type II secretion system из KEGG. Исходный ген покрашен красным, гены из консервативного геномного окружения (наборы совпадают) - зеленым. Гены, точно присутствующие в нашем опероне, отмечены желтым.
Все белки из константного геномного окружения, найденные SEEDом, кроме gspN, входят в этот метаболический путь, являясь субчастицами одного большого мембранного транспортного комплекса (рис.4). Белки GspO и GspS, также функционально связанные (хоть и не столь тесно в случае GspO, судя по изображению 4) с данным комплексом, SEEDом не были найдены, и в нашем опероне из Pseudomonas putida не закодированы.
У Pseudomonas putida не обнаружилось генов, кодирующих белки с названиями, соответствующими gspK, gspM и gspC, зато нашлись xcpP, xcpW и xcpZ, вроде как отсутствующие у других найденных организмов. Как уже было сказано, я думаю, что продукты каких-то из этих генов функционально соответствуют gspK, gspM и gspC.
Итоговая таблица: