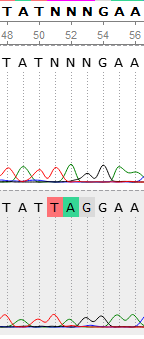
Для прямой хроматограммы (20_F.ab1):
Для обратной хроматограммы (20_R.ab1):
Далее при помощи программы UGene, выбрав инструмент для анализа данных, полченных при секвенировании по Сэнгеру, загрузил данные мне чтения. В качестве референсного использовалось прямое чтение, в параметре чтений были выбраны прямое и обратное чтения. Остальные параметры остались без изменений.
Выравнивание до корректировки лежит по ссылке - файл
Затем проводилась корректировка консенсуса. Ее описание кратко можно видеть ниже.

Данные полиморфизмы исправлялись в соответствии с сигналами хроматограммы и данными с обратного прочтения.
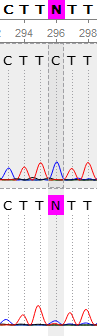
Данный полиморфизм исправлялся в соответствии данными с обратного прочтения.

Данные полиморфизмы исправлялись в соответствии с сигналами хроматограммы и данными с обратного прочтения, в итоге получилось, что один нуклеотид определяется однозначно, второй было решено оставить "К" в консенсусе, то есть кето.
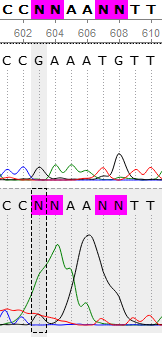
Данные полиморфизмы исправлялись в соответствии с сигналами хроматограммы и данными с обратного прочтения, в итоге получилось, что два нуклеотида однозначно определены, а два других соответствуют кето и пурину (было решено, что так будет точнее оставить).
Далее было выполнено выравнивание на референсную последовательность. В этом выравнивании спорны буквы были отредактированы вручную. Сам файл с консенсусной последовательностью можно найти по ссылке- файл
Ниже представлено изображение участка плохой хроматограммы (была выбрана обратная хроматограмма WS2950_H3).
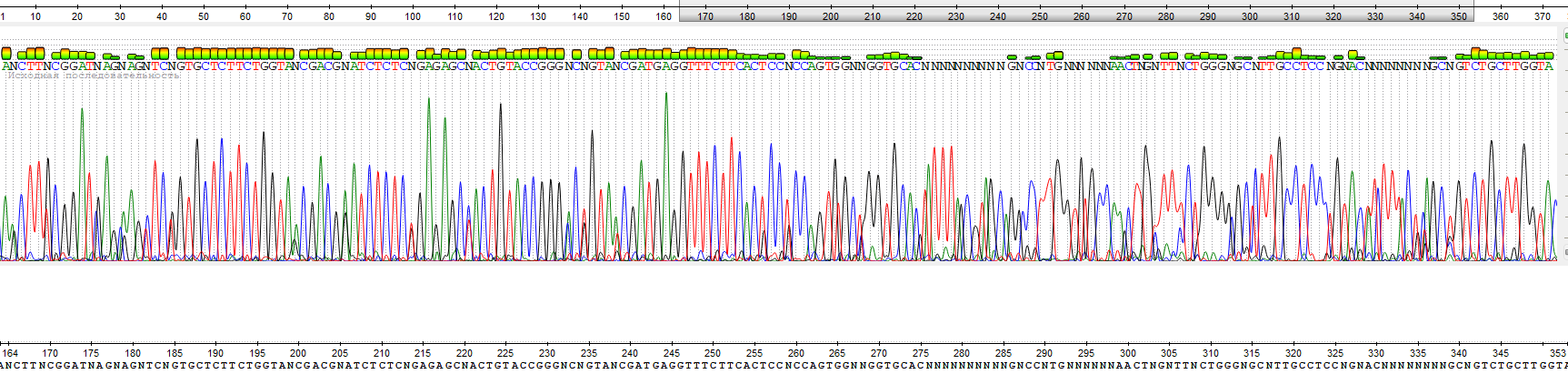
На данной хроматограмме, как и на любой другой, нечитаем начальный участок, однако этот участок длится до 120 позиции, что уже очень много. Но помимо этого в середине хроматограммы есть нечитаемые участки, изображение которых можно видеть выше. Также стоит отметить то, что во многих участках программа не сильно уверена в выборе нуклеотида из-за шумов, что дает поводы для сомнения и нам.