Для данной работы использовался пакет NPG-explorer, скачанный на персональный компьютер. Для анализа были выбраны 5 штаммов Mycobacterium tuberculosis, чьи аннотированные геномы можно найти на ncbi. Ключевой разницей штаммов был взят год составления сборки, сами годы можно найти во входном файле, а также в дальнейшем используется именно эти годы, как подписи организмов. Входной файл можно посмотреть по следующей ссылке - файл.
Далее для удобства был написан маленький скрипт, который выполняет все последовательные команды пакета NPG-explorer, включая изменение в файле npge.conf (стоит отметить, чтотак как на персональном компьютере стоит операционная система Windows, то синтаксис команд несколько отличается от предложенного в подсказках). сам скрипт можно посмотреть по ссылке - файл. Вся директория, в которой проводилась работа, затем была сопирована в директорию на kodomo term3/block2/credits.
Информация о полученном пангеноме была взята из файла pangenome.info. Ниже представлена основная информация:
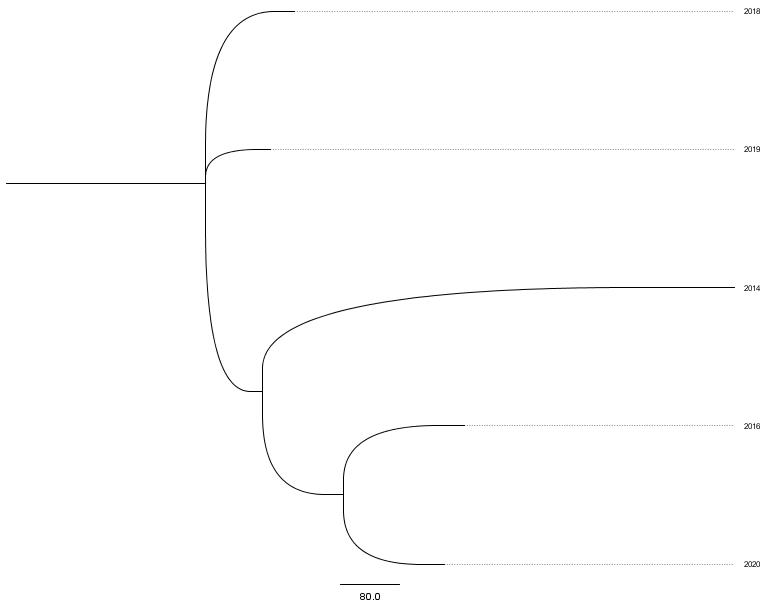
Также было построено дерево на осове схожести геномов штаммов. Как можно видеть, все штаммы в целом были близки (это видно из масштаба), но еще можно заметить, что штаммы 2018 и 2019 годов очень близки друг к другу, нежели другие.

Для начала было решено рассмотреть u-блоки. Для упрощения был взят файл partition-grouped.tsv и при помощи скрипта были отобраны такие блоки. Получилось, что есть 46 генов в таких блоках. Далее эти блоки были просмотрены в NPG-explorer, получилось, что в большинстве из блоков указано просто "potential protein", хотя в некоторых были и определенные белки, например, "polyketide synthase". Также стоит отметить, что такие блоки присутствуют в штаммах 2014, 2016 и 2018 года, что некоторым образом делает эти штаммы уникальней по сравнению с остальными. Итоговый файл с отобранными блоками - блоки
Далее я решил рассмотреть h-блоки. Для этого файл pangenome.bi был открыт с помощью Excel. Далее были рассмотрены строчки, которые начинаются только на "h", и они также были отсортированы по длине (по количеству колонок в блоке). Изучив затем результаты, можно заметить, что у штамма 2018 года произошла достаточно большая делеция (на 9618 нуклеотидов), однако, посмотрев этот блок в NPG-explorer, оказалось, что в ней не кодировался никакой белок. Этот факт был подмечен, так как, рассмотрев следующий h-блок, я увидел, что делеция на 8357 нуклеотидов у штамма 2019 года соответствовала участку, кодирующему хеликазу у двух других штаммов (у 2016 и 2018). Аналогично можно описать делецию на 3646 нуклеотидов у штамма 2014 года: для него есть информация о кодирующем участке (glycine/betaine ABC transporter substrate-binding) у штаммов 2016 и 2018 годов. Следующий блок - делеция в штамме 2016 года - интересен не столько, как делеция, а больше, как перемещенный блок, причем, судя по тому, что он отстоит у штамма 2014 года, а далее сохраняется у всех на одном и том же месте, можно говорить о том, что перемещение произошло между 2014 и 2018 годами, точнее сказать не получается, так как он отсутствует именно у штамма 2016 года. Наибольшая делеция у штамма 2020 года составляла 1064 нуклеотида. Стоит заметить, что для этой делеции есть анотация в других штаммах: FAD-containing monooxygenase в штаммах 2014 и 2016 года и FAD-dependent oxidoreductase в штамме 2018 года.
Хотелось бы подвести итоги и описать некоторые аспекты предыдущего абзаца. По построенному дереву, а также по инсерциям и делециям можно говорить, что штаммы разных годов очень близки (возможно, стоило бы взять штаммы, различающиеся сильнее во времени секвенирования, но изначально было решено, что более новые последовательности будут более достоверными, а после выполнения работы я пока что не стал проверять иные годы). Что касается u-блоков, то скорее всего, все неопределенные блоки NPG-explorer в целом считает инсерциями. Теперь стоит обсудить аннотацию блоков. Можно заметить, что в описании h-блоков говорилось о различиях в кодируемых последовательностях, но стоит вспомнить, что оксидоредуктаза и монооксигеназа - в некотором роде одно и то же (второе является подклассом первого). А также, судя по всему, сборки 2016 и 2018 года были аннотированы гораздо лучше остальных сборок.
Оказалось, что если запускать программу несколько раз, то могут получаться различные данные, а именно после первого запуска было получено 393 стабильных блоков, а для получения log-файлов запустил повторно, что привело к тому, что получилось 394 стабильных блоков. Такие же слова можно сказать и про остальные численные данные, которые указываются в данном практикуме. Хотя, вероятно, это справедливо для всей работы NPG-explorer.
Также хотелось бы сказать несколько слов про входной файл genomes.tsv. Для работы программы нужно верно указать базу, в которой искать, AC в этой базе данных, тип молекулы и номер хромосомы. А название штамма выбирается так, чтобы было удобно работающему.
По причине вышесказанного я исправил сам файл genomes.tsv, но не перезапускал работу программы, поэтому на данной странице не изменено название с просто "год" на "год и название штамма".