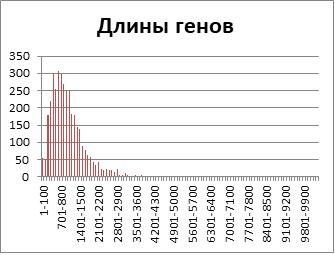
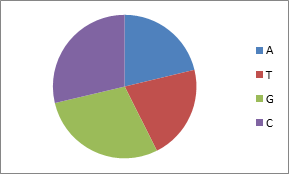
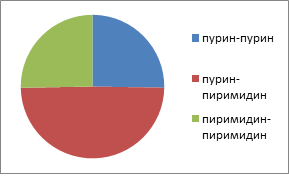
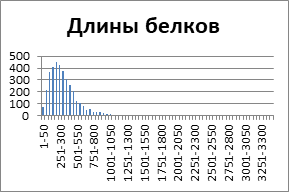
В данной работе хотелось бы рассмотреть протеом такой особи, как Marinobacter hydrocarbonoclasticus. Данная бактерия является представителем протеобактерий, а именно относится к гамма-протеобактериям. Что означает, что данная бактерия является не только грамм-отрицательной, но и палочковидной, при этом клетки этих бактерий имеют один жгутик. Однако способность к отращиванию жгутика зависит от концентрации NaCl в окружающей среде, а именно они способны к данному действию только при концентрации от 0,6 до 1,5 М.[1]
Стоит отметить уникальную особенность этих бактерий. А именно, они используют нециклические углеводороды как субстрат роста. Это вызвано тем, что в их клетках отсутствует один из важнейших ферментов для разложения циклических углеводородов.[1] Эта особенность раскрывает актуальность изучения данных бактерий, ведь линейные углеводороды являются ключевыми компонентами нефти и машинного топлива. Поэтому они очень важны с экологической точки зрения, так как могут помочь минимизировать ущерб окружающей среде от различных поломок на нефтедобывающих станциях или на различных судах, которые терпят крушения в мировом океане.[2]