Сначала был проведен локальный бласт при помощи скрипта. Скрипт можно посмотреть по следующей ссылке - скрипт. все выдачи локального бласта можно посмотреть по следующей ссылке - выдачи.
Далее были выбраны мнемоники белков, имеющих хорошее сходство с исходной последовательностью. Их загрузил в Jalview и провел выравнивание, а потом по выравниванию было построено дерево методом NeighborJoining. По следующей ссылке можно видеть дерево в Newick-формате - дерево. Ниже представлены два изображения дерева.
На данных изображениях ортологи объеденены при помощи квадратных скобок (рядом написаны названия этих белков), а для паралогов покрашены вытви ведущие к листам одним и тем же цветом.

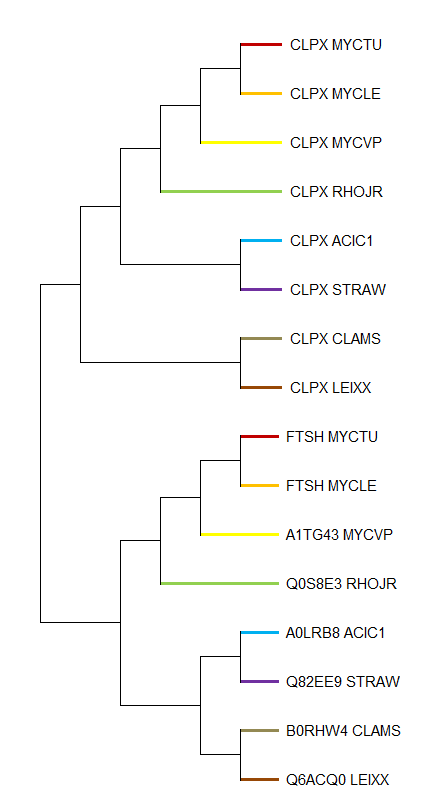
Как можно видеть из данных изображений, филлогения для металлопротеазы и простой протеазы полностью соответствуют филлогении бактерий. Единственное, что можно заметить,- для металлопротеазы входящая ветвь в группу совпадает с корнем у бактериального дерева, а у простой протеазы такого совпадения нет. Данное замечание дает повод для вывода о том, что исходно была протеаза, от нее отделилась металлопротеаза, для которой дальнейшее филлгенетическое разделение полностью соответствует таковому для бактерий.
Также ниже будет представлено дерево с схлопнутыи ветвями. Его вид позволяет утверждать, что прошло достаточно времени для того, чтобы в выравнивании эти два белка хорошо отличались и разбивались на дереве в отдельные ветви.