Сведения о бактерии Defluviicoccus vanus
Изгагин Михаил
Московский Государственный Университет имени М. В. Ломоносова, факультет биоинженерии и биоинформатики, г. Москва
1 ВВЕДЕНИЕ
Бактерия вида Defluviicoccus vanus рода Defluviicoccus семейства Geminicoccaceae отряда Rhodospirillales класса Alphaproteobacteria типа Pseudomonadota домена Bacteria – грамотрицательная бактерия, выделенный из образца биомассы с завода по производству активного ила EBPR [1].
Представители рода Defluviicoccus встречаются в очистных сооружениях сточных вод в виде кластерных или тетрадных колоний [1]. Особенностью представителей Defluviicoccus является накопление гликогена, что связано с проживанием в чередующихся условиях анаэробного питания [2].
2 МАТЕРИАЛЫ И МЕТОДЫ
Материалы про геном, кодирующие последовательности ДНК, последовательности аминокислот бактерии были взяты из источника {1}.
Гистограммы длин белков и расстояний между кодирующими последовательностями были построены с помощью Google Sheets {2}, {3}. Источник данных {1.1}.
Таблица старт кодонов была построена с помощью Google Sheets {4}. Результаты для этой таблицы были получены с помощью скриптов написанных в bash {5}. Источник данных {1.1}.
Результаты для GC-содержания (источник данных {1.3}); получение аминокислотных последовательностей из кодирующих последовательностей ДНК и сравнение с уже имеющимися данными (источник данных {1.1}, {1.2}); подсчета количества белков начинающихся с метионина и с других аминокислот (источник данных {1.2}) были получены с помощью программ на языке Python {6}, {7}, {8}.
3 РЕЗУЛЬТАТЫ
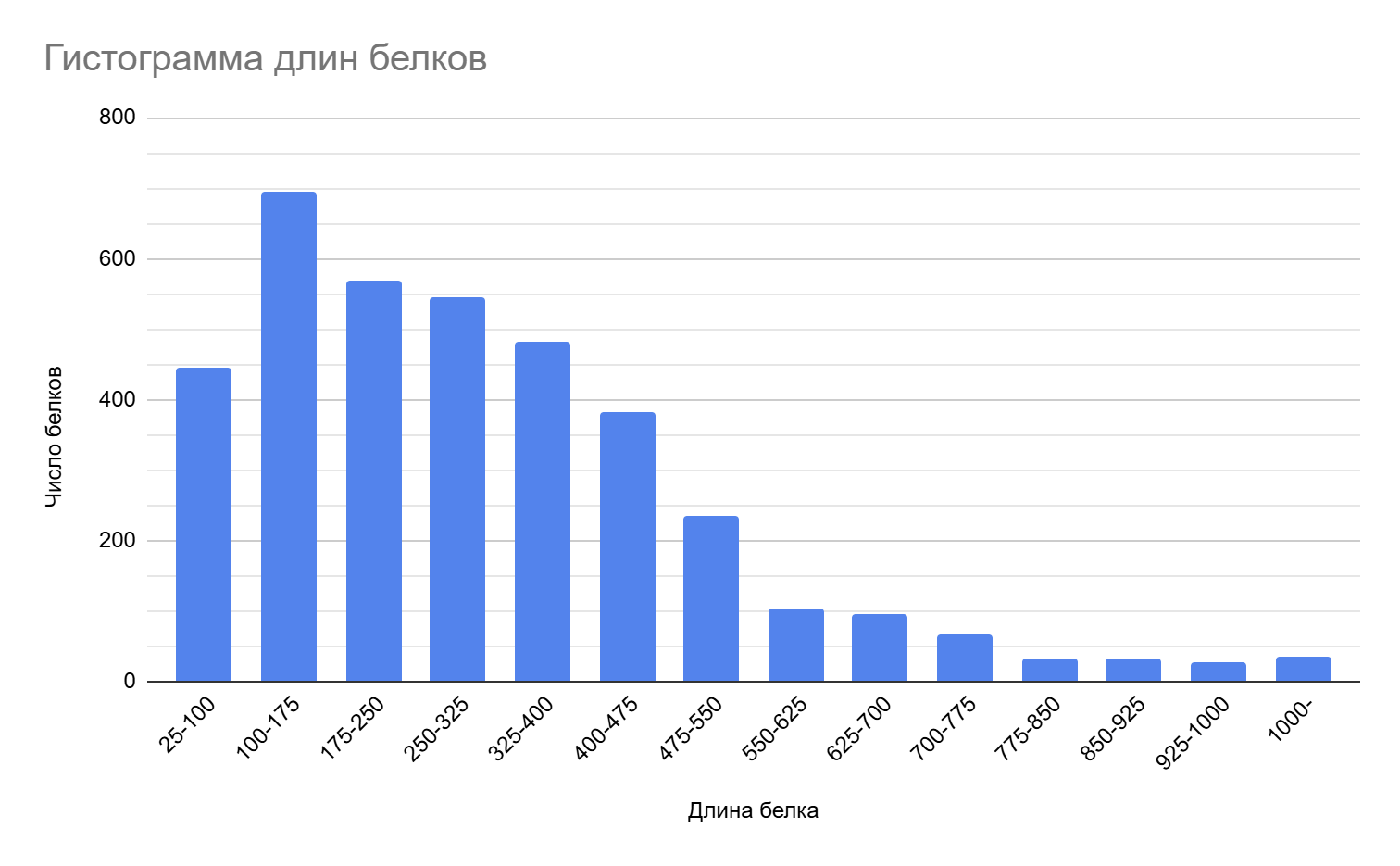
3.1 Дины белков, закодированных в геноме бактерии Defluviicoccus vanus
Из рис.1 видно, что наибольшее число белков состоит примерно из 25 - 550 аминокислот. Также средняя длина белка составляет 315 аминокислот, что в среднем оказывается немного больше, чем у других представителей Класса Alphaproteobacteria [3].
Полученный результат сходится со средней длинной белков у прокариот [4].
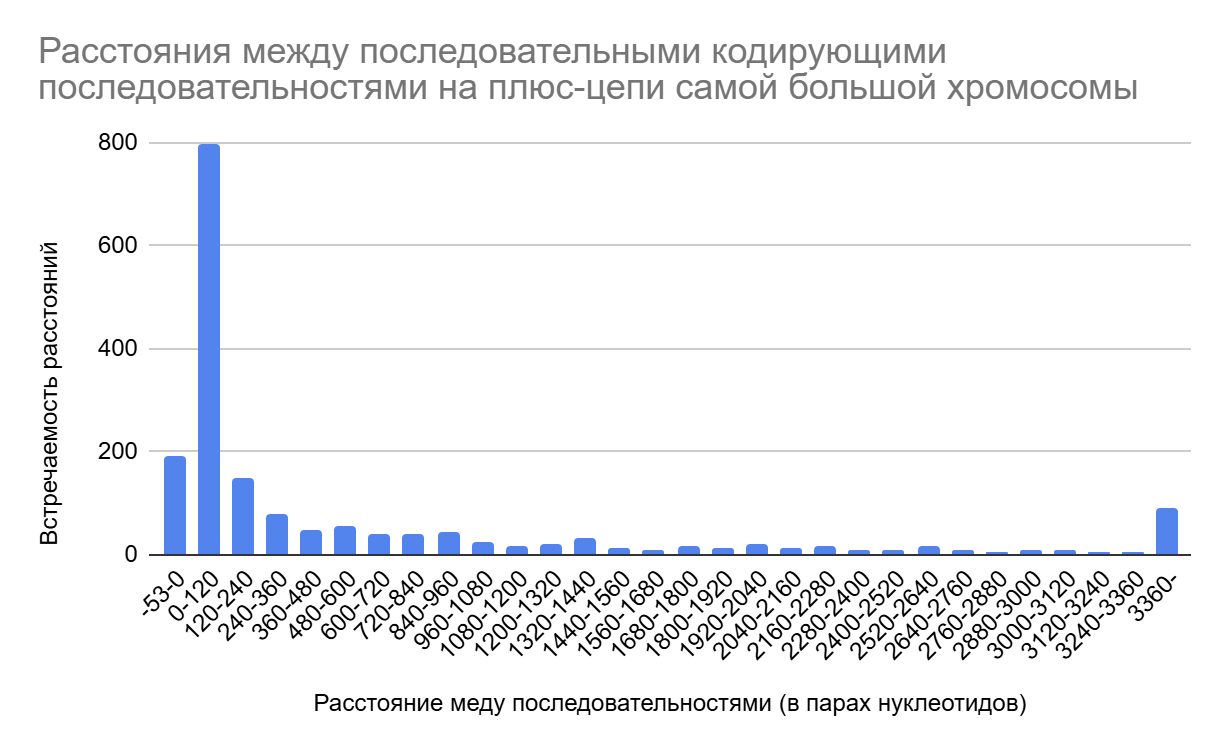
3.2 Расположение кодирующих последовательностей на плюс-цепи самой большой хромосомы
Из рис.2 можно заметить, что большая часть кодирующих последовательностей располагается рядом друг с другом. Также не маленькое количество последовательностей начинаются внутри предыдущих, что на гистограмме представлено самым левым столбцом, с отрицательными значениями расстояния. Из этого можно сделать предположение, что: либо одна из пересекающихся последовательностей является псевдогеном; либо механизм расположения кодирующих последовательностей устроен так, чтобы синтезировать максимальное количество белков с минимального по длине участка.
В рассмотрение брались все последовательности, кодирующие белки (не только сонаправленные).
3.3 Частота встречаемости разных старт-кодонов
Под остальными кодонами имеется ввиду: AAG, AAT, AAN, AGG, AGC, ACA, ACG, GAG, GAC, GGT, GTA, CAA, CAG, CAT, CCA, CCG, TAC, TGC, TCA. Каждый из них встретился в All CDS и Pseudo CDS ровно один раз.
Из табл. 1 видно, что большее количество генов имеет старт-кодон ATG, который является наиболее распространённым [5]. Но также встречаются и другие старт-кодоны [5], в том числе те, у которых не определены некоторые нуклеотиды. Также можно заметить, что многие нестандартные старт кодоны являются старт-кодонами псевдогенов.
Стоит отметить немалое количество встреченных старт-кодонов GTG и TTG.
3.4 GC-содержание в во всем геноме
GC-содержание генома составило 63,542%. Это является довольно большим показателем, так как у Alphaproteobacteria GC-содержание варьируется в среднем от 30% до более 60% [6].
Повышенное содержание GC с большей вероятностью не вызвано ошибкой секвенирования, трансляционным отбором, смещенной генной конверсией или горизонтальным переносом генов. А также говорит о потенциальном качественном естественном отборе в сторону увеличения GC-содержания в геноме, так как при увеличении содержания GC увеличивается количество мутаций GC→AT [6].
3.5 Выделение последовательности аминокислот из кодирующих последовательностей участков ДНК
Выявлено, что расхождения наблюдались только в старт кодонах. Последовательности аминокислот (в имеющемся источнике данных) начинаются с метионина (Met) независимо от того, с какого триплета начинается кодирующая последовательность. Это может быть связано с окружающими старт-кодон последовательностями [8], [9].
Кроме того частота встречаемости старт-кодонов GTG (15,822%) оказалась выше, чем в среднем, а TTG (6,929%) ниже [7], что подкрепляется GC-содержанием, определенном в прошлом пункте.
Данная программа выдаёт встречаемость старт кодонов с небольшой погрешностью (данные расходятся с результатами, полученными в 3.3), так как она показывает сколько раз метионин кодировался не триплетом ATG, а другим триплетом. Например триплет GTG в данном случае встретился только 596 раз, а не 597, так как в одном из случаев он кодировал именно свою аминокислоту (Val).
3.6 Количество белков начинающихся с метионина и с другой аминокислоты
Было выявлено, что все белковые последовательности начинающиеся не с метионина, которых оказалось 78 штук, оказались псевдогенами. Это может говорить о том, что организму невыгодно синтезировать белки с аминокислот отличных от метионина.
Все эти псевдогены начинались с триплетов отличных от ATG, то есть их старт кодоны кодировали именно тот белок, который и должны, а не подавлялись и не кодировали метионин [8], [9]. Эти данные объясняют небольшие расхождения в предыдущем пункте.
Таблица 1. Частота встречаемости разных старт кодонов
| Codons | All CDS {5.1} | Pseudo CDS {5.2} | Normal CDS |
|---|---|---|---|
| ATG | 2747 | 177 | 2570 |
| GTG | 597 | 23 | 574 |
| TTG | 257 | 9 | 248 |
| CTG | 40 | 5 | 35 |
| ATC | 35 | 3 | 32 |
| ATT | 17 | 3 | 14 |
| CGG | 8 | 8 | 0 |
| GGC | 7 | 7 | 0 |
| ATA | 4 | 1 | 3 |
| GCC | 4 | 4 | 0 |
| CGC | 4 | 4 | 0 |
| TTC | 4 | 4 | 0 |
| AAC | 2 | 2 | 0 |
| ACC | 2 | 2 | 0 |
| GGG | 2 | 2 | 0 |
| GCT | 2 | 2 | 0 |
| GTC | 2 | 2 | 0 |
| CCC | 2 | 2 | 0 |
| CTA | 2 | 2 | 0 |
| CTC | 2 | 2 | 0 |
| CTT | 2 | 2 | 0 |
| TAT | 2 | 2 | 0 |
| TCC | 2 | 2 | 0 |
| NTG | 2 | 0 | 2 |
| Остальные | 19 | 19 | 0 |
БЛАГОДАРНОСТИ
Выражаю благодарность Нагорному Даниилу Васильевичу, Аржаевой Елене Викторовне, Балакиной Ольге Александровне и Скоморохову Глебу Егоровичу, за поддержание морального духа и за некоторые правки по работе. Без их поддержки я бы не справился.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
- {1} Файлы (* - GCF_014672695.1_ASM1467269v1)
- {1.1} *_cds_from_genomic.fna.gz;
- {1.2} *_translated_cds.faa.gz;
- {1.3} *_genomic.fna.gz
- {2} Гистограмма и таблица длин белков.
- {3} Гистограмма и таблица расположения кодирующих участков на плюс-цепи самой большой хромосомы (страница: inter_cds_intevals-hist).
- {4} Таблица старт кодонов и частоты их встречаемости.
- {5} Путь до скриптов в баш:
- {5.1} /home/students/y24/iz-mi-al/term1/jojo/pep; Ссылка на скрипт на гугл диске.
- {5.2} /home/students/y24/iz-mi-al/term1/jojo/tet; Ссылка на скрипт на гугл диске.
- {6} Код для высчитывания GC-содержания во всём геноме.
- {7} Код для определения последовательности аминокислот по кодирующей последовательности ДНК.
- {8} Код для высчитывания количества белков начинающихся с метионина и с других аминокислот.
ЛИТЕРАТУРА
- [1] A. M. Maszenan; R. J. Seviour; B. K. C. Patel; P. H. Janssen; J. Wanner (2005) Defluvicoccus vanus gen. nov., sp. nov., a novel Gram-negative coccus/coccobacillus in the ‘Alphaproteobacteria’ from activated sludge.
- [2] Irina Bessarab; Abdul Majid Maszenan; Mindia A. S. Haryono; Krithika Arumugam; Nay Min Min Thaw Saw; Robert J. Seviour; Rohan B. H. Williams (2022) Comparative Genomics of Members of the Genus Defluviicoccus With Insights Into Their Ecophysiological Importance.
- [3] Luciano Brocchieri; Samuel Karlin (2005) Protein length in eukaryotic and prokaryotic proteomes.
- [4] Axel Tiessen; Paulino Pérez-Rodríguez; Luis José Delaye-Arredondo (2012) Mathematical modeling and comparison of protein size distribution in different plant, animal, fungal and microbial species reveals a negative correlation between protein size and protein number, thus providing insight into the evolution of proteomes.
- [5] J Biol Chem; D S Peabody (1989) Translation initiation at non-AUG triplets in mammalian cells.
- [6] Falk Hildebrand; Axel Meyer; Adam Eyre-Walker (2010) Evidence of Selection upon Genomic GC-Content in Bacteria.
- [7] Andre Villegas; Andrew M. Kropinski (2008) An analysis of initiation codon utilization in the Domain Bacteria – concerns about the quality of bacterial genome annotation.
- [8] C.Magnus Stenström; Erik Holmgren; Leif A Isaksson (2001) Cooperative effects by the initiation codon and its flanking regions on translation initiation.
- [9] Marilyn Kozak (1999) Initiation of translation in prokaryotes and eukaryotes.