Семейства белковых доменов. PFAM
Семейство домена Tryptophan halogenase
В предыдущем практикуме мною было выбрано семейство доменов с наименованиями представленными в таблице 1.
Триптофангалогеназа катализирует хлорирование триптофана. Связывает субстрат таким образом, чтобы открыть положение C5 для ключевых остатков лизина и глутамата в месте хлорирования. Это первый шаг в биосинтезе пирролнитрина, антибиотика с широким спектром противогрибковой активности. Ниже (рисунок 1) представлена его структура, где жёлтым выделен активный центр, розовым - остатки участвующие в связывании, оранжевым - важный участок для активности белка.
Рисунок 1. Структура триптофангалогеназы
| Таблица | Информация |
|---|---|
| AC pfam | PF04820 |
| ID pfam | Tryptophan halogenase |
| #SEED | 23 |
| #All | 5394 |
| #SW | 22 |
| #architectures | 70 |
| #3D | 63 |
| Taxonomy | |
| #eukaryota | 1013 |
| #archaea | 271 |
| #bacteria | 14360 |
Информация из этой таблицы может расходиться с информацией из предыдущего практикума, так как в предыдущей работе были использованы данные актуальные на 2023 год, а предсставленные здесь, взяты из Pfam на данный момент.
Белки из этого семейства имеют часть последовательности этого белка, и большинство из этих белков содержат именно ту часть, где есть активный участок. Вероятнее всего активный центр в этих белках сохраняет свою функцию. Но также было найдено и несколько белков с большим участком схожести, но не включающие участок с активным центром. Скорее всего у них участок данного домена сохранил в большей мере функцию связывания.
Данный домен по большей мере распространён у бактерий.
Карта локального сходства
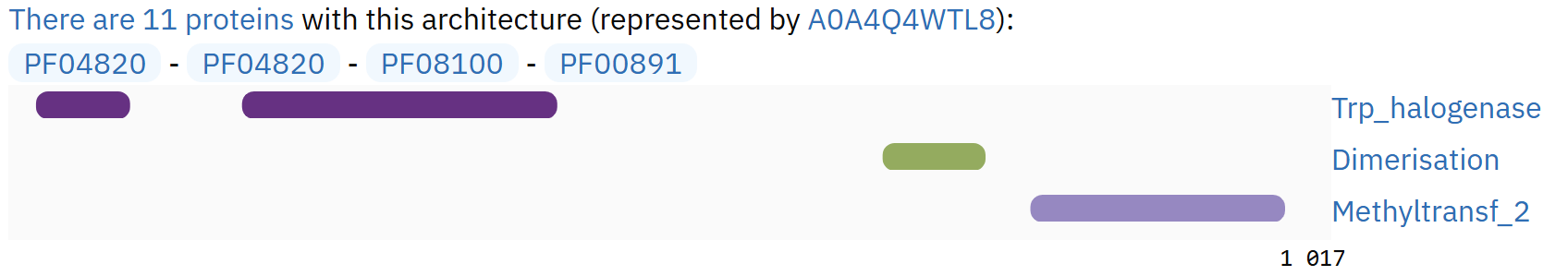
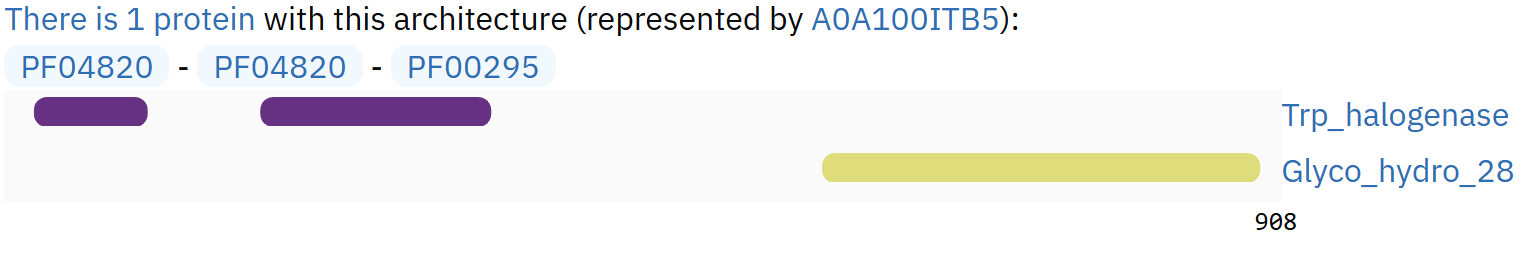
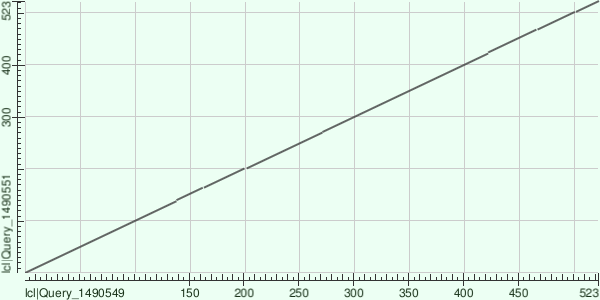
Для построения карты локального сходства были выбраны последовательности содержащие домен PF04820, но с разными доменными архитектурами. Данные представлены в таблице 2.
По карте локального сходства (рисунок 2) можно увидеть довольно сильное сходство. Это выражается незначительными разрывами и почти идеальной прямой. Вероятнее всего белки разошлись в эволюционном плане недавно. Также в обоих выбранных белках есть по 2 копии домена, однако выравнивание на карте одно. Это выравнивание до 523-го аминокислотного остатка, хотя домены в данных белках встречаются раньше. Выравнивание одно, вероятнее всего потому, что данные белки сильно похожи начальными участками, из-за чего области домена не видны. Значение E-value в данном выравнивании равно 0, что говорит о сильной достоверности результата.
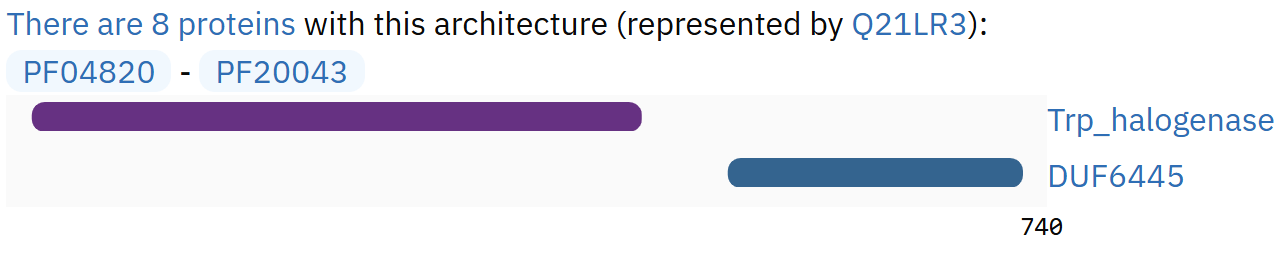
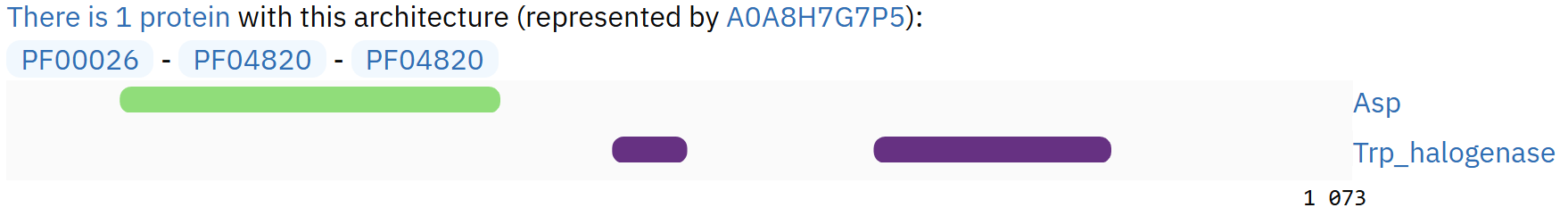
В противовес данному довольно точному результату можно посмотреть на другие архитектуры, а именно на PF04820 - PF20043 и на PF00026 - PF04820 - PF04820. На полученной карте (рисунок 3) видно огромную вставку.
| Наименование | Информация |
|---|---|
| доменная архитектура 1 | PF04820 - PF04820 - PF08100 - PF00891 |
| белок с архитектурой 1 | A0A4Q4WTL8 |
| доменная архитектура 2 | PF04820 - PF04820 - PF00295 |
| белок с архитектурой 2 | A0A100ITB5 |
.png)
Данные результаты подтверждаются и иллюстрацией с самой базы данных (рисунок 4 и рисунок 5 для первой карты локального сходства; рисунок 6 и рисунок 7 для второй карты локального сходства).