Комплексы ДНК-белок
Предсказание вторичной структуры заданной тРНК (1N78)
С помощью данной команды:
einverted -sequence rcsb_pdb_1N78.fasta -gap 12 -threshold 10 -match 3 -mismatch -3 -outfile outfile -outseq seqout
был получен файл outfile с содержимым:
EMBOSS_001: Score 15: 7/9 ( 77%) matches, 0 gaps
1 ggccccatc 9
|||| | ||
31 ccggcgcag 23
EMBOSS_001: Score 15: 10/15 ( 66%) matches, 0 gaps
34 ctcaaggccgaaacg 48
| | ||| ||| ||
69 ggggtcccccttagc 55
В результате команды:
remediator --old 1N78.pdb > 1N78_old.pdb
# Информация о 1N78
find_pair -t 1N78_old.pdb stdout | analyze
в предыдущем практикуме были получены результаты взаимодействий внутри РНК.
С помощью алгоритма Зукера было получено изображение возможной структуры тРНК (рис. 1)
В таблице 1 представлены результаты сравнения применённых методов.
| Участок структуры | Позиции в структуре (по результатам find_pair ) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | (501-507) - (572-566) | (1-9) - (31-23) | (1-7) - (65-71) |
| D-стебель | (510-513) - (525-522) | - | (9-13) - (22-26) |
| T-стебель | (549-553) - (565-561) | - | (48-52) - (60-64) |
| Антикодоновый стебель | (539-543) - (531-527) | (34-48) - (69-55) | (27-31) - (39-43) |
| Общее число канонических пар нуклеотидов | 20 | 17 | 22 |
Поиск ДНК-белковых контактов в заданной структуре
С помощью скрипта в Jmol были получены изображения (рис. 2, рис. 3, рис. 4, рис. 5, рис. 6).

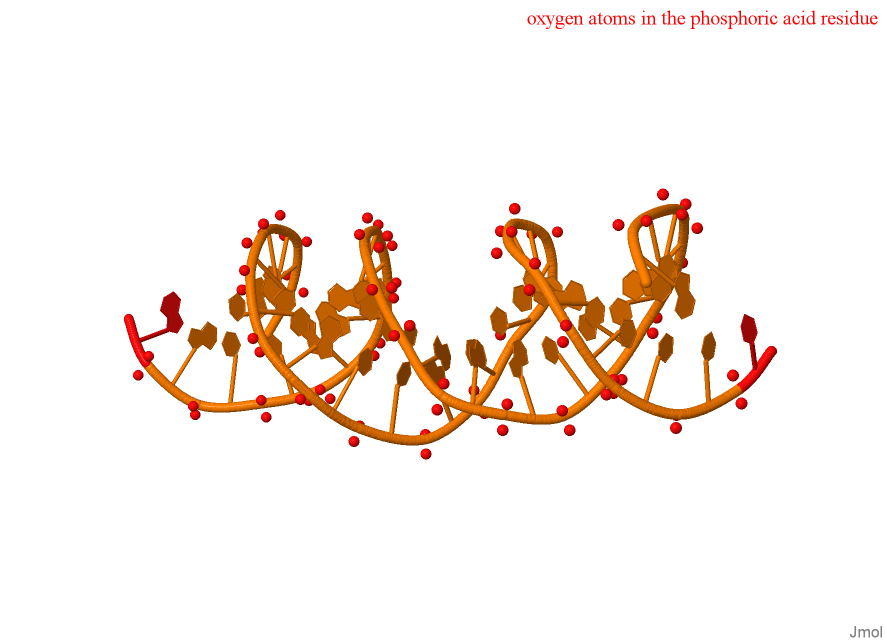
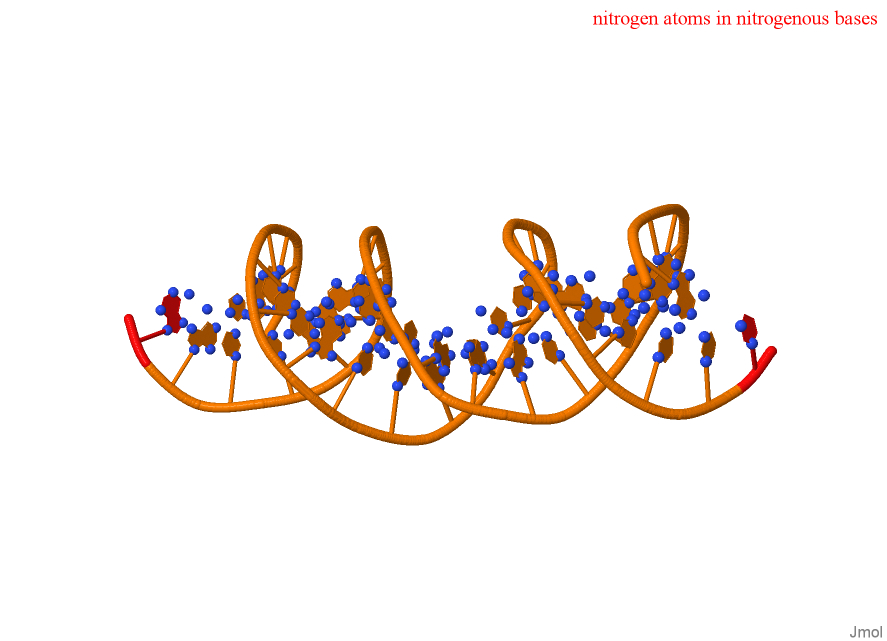
Контакты ДНК-белок
С помощью друго скрипта в Jmol были получены данные о полярных и неполярных взаимодействиях между ДНК и белком. Полученные результаты представлены в таблице 2.
| '''Контакты атомов белка с''' | '''Полярные''' | '''Неполярные''' | '''Всего''' |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 12 | 70 | 82 |
| остатками фосфорной кислоты | 21 | 27 | 48 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 16 | 16 |
| остатками азотистых оснований со стороны малой бороздки | 9 | 12 | 21 |
Схема ДНК-белковых контактов с помощью программы nucplot
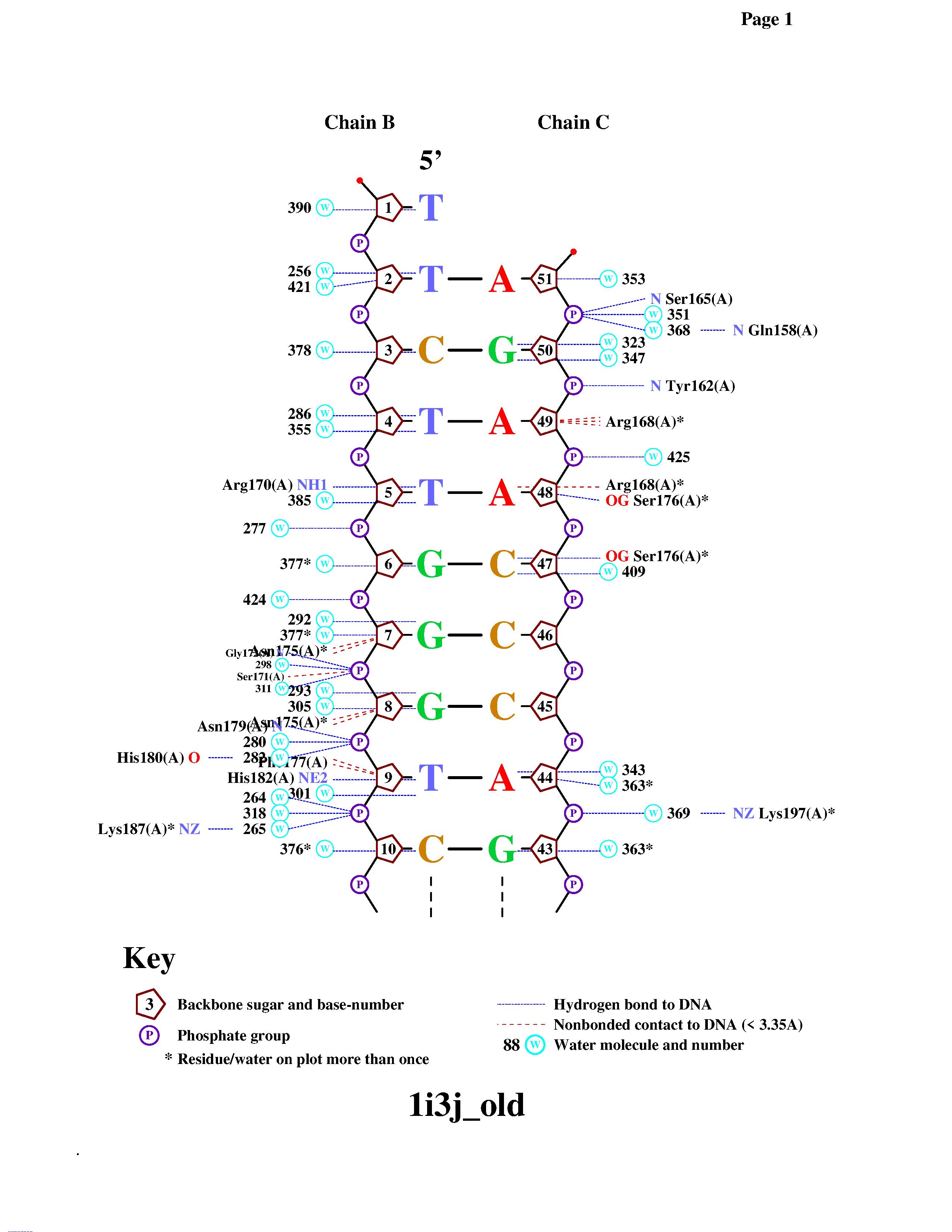
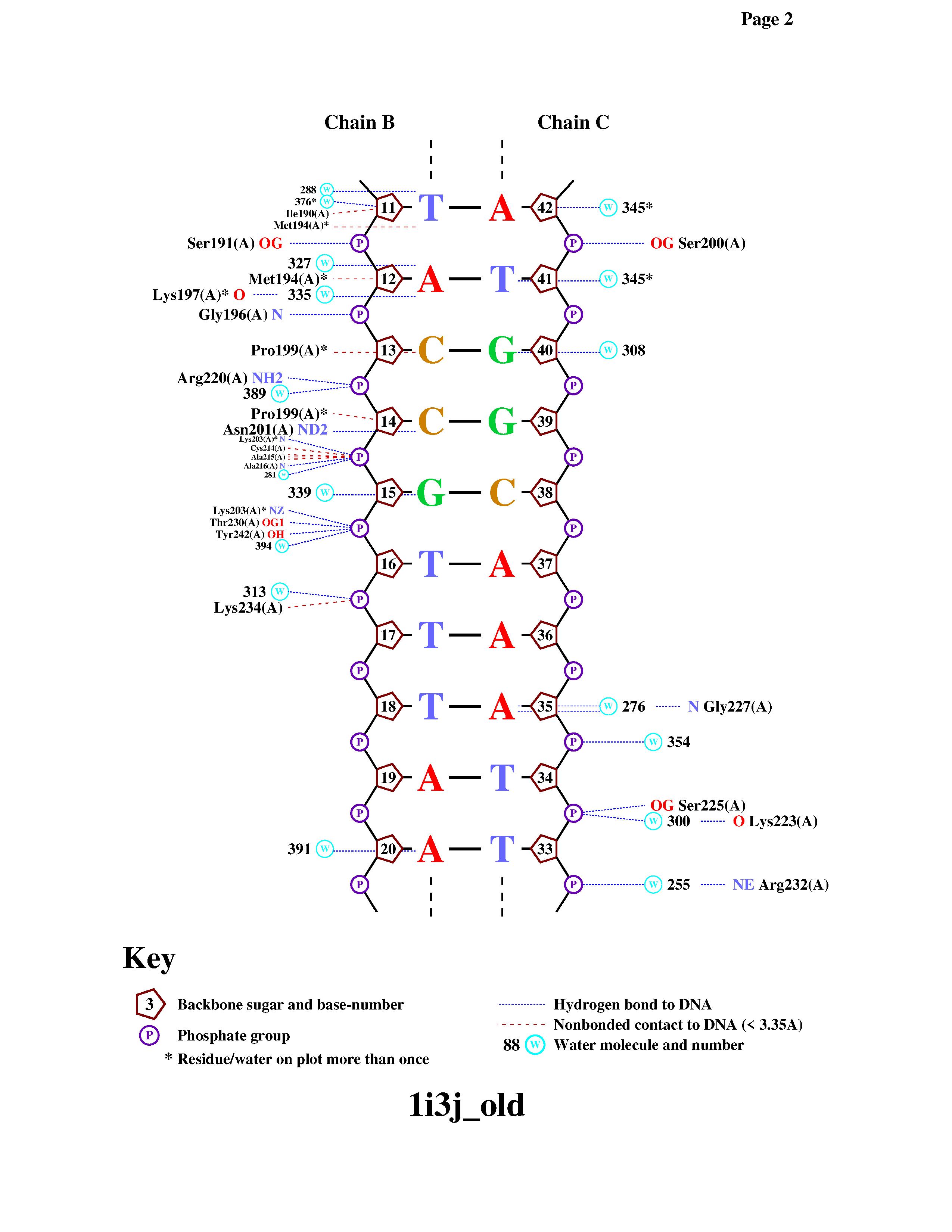
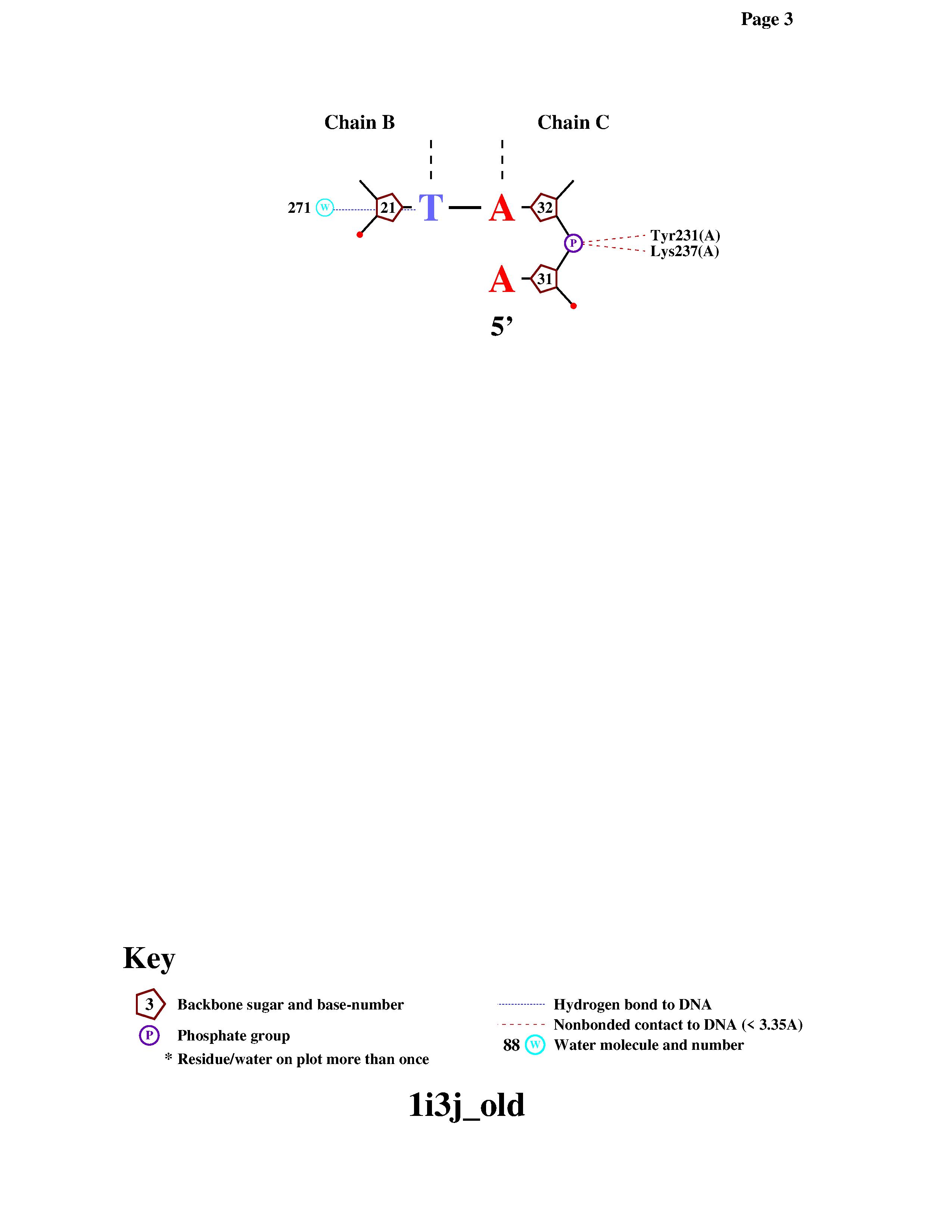
Аминокислотные остатки с наибольшим количеством контактов с ДНК:
- Arg168 - с 48 аденином и 49 остатками дезоксирибозы
- Asn175 - с 7 и 8 остатками дезоксирибозы
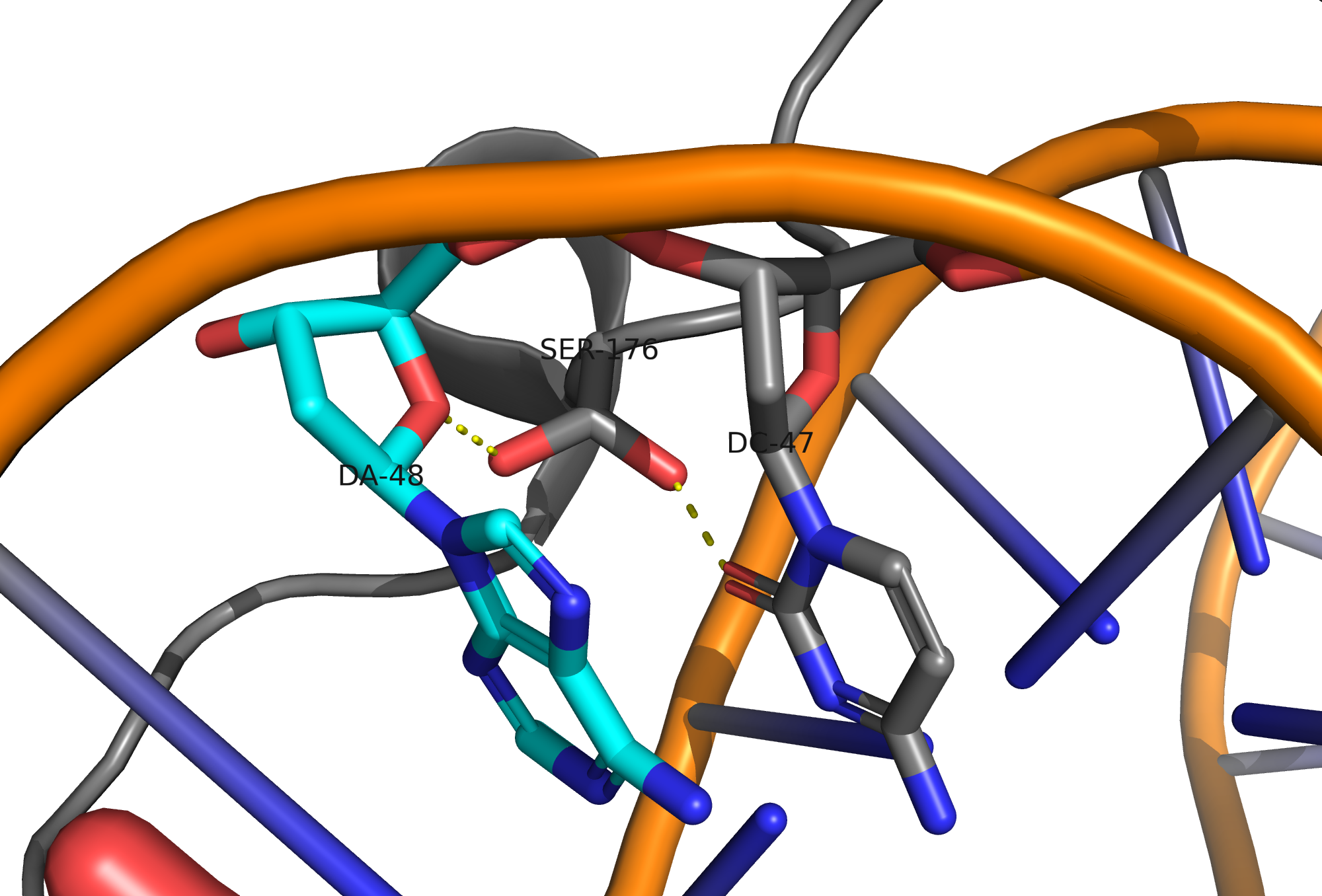
- Ser176 - водородные связи с 47 цитозном и 48 остатками дезоксирибозы
- Met194 - с 11 тимином и 12 остатком дезоксирибозы
- Pro199 - с 13 цитозином и 14 остатком дезоксирибозы
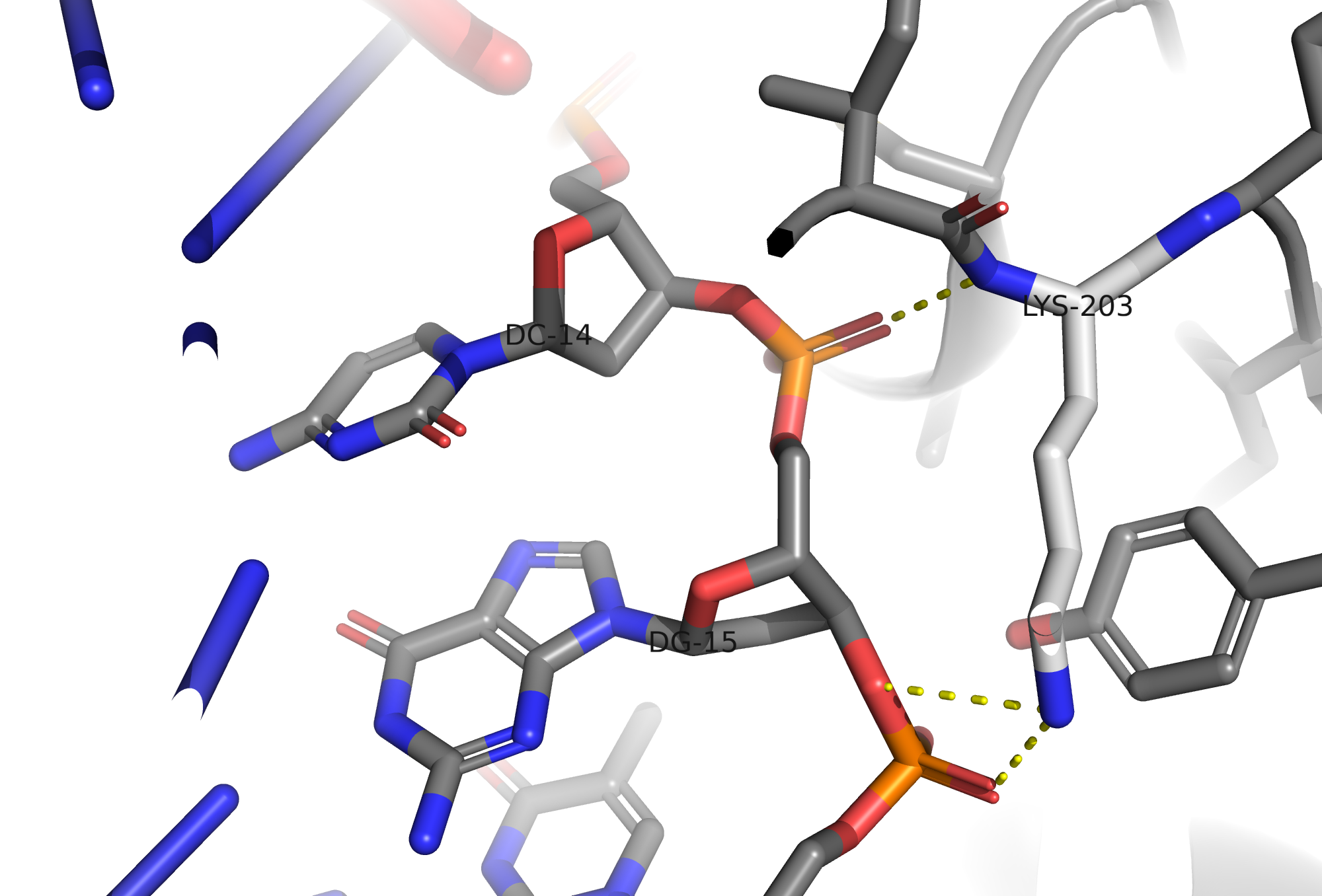
- Lys203 - водородные связи с фосфатами между 14 и 15 остатками дезоксирибозы и 15 и 16 остатками дезоксирибозы
Наиболее важным вероятнее всего является Ser176, так как он образует водородные связи с дезоксирибозой и цитозином. Ниже приведены рисунки некоторых взаимодействий (рис. 10, рис. 11)