РЕЗЮМЕ
В данной работе кратко описан геном бактерии Desulfitobacterium metallireducens DSM 15288. По ходу работы был проведен анализ длин белков организма, анализ типов генов, из которых состоит геном, их подсчет по матричной и комплементарной цепям ДНК, анализ димеров генома, поиск места начала и конца процесса удвоения ДНК (репликации), анализ межгенных промежутков по прямой и обратной цепям, а также выделены некоторые кластеры генов с сходной функцией.
КЛЮЧЕВЫЕ СЛОВА
DSM 15288, геном, белок, рибосома, ген, бактерия
ВВЕДЕНИЕ
Desulfitobacterium metallireducens получили свое название из-за того, что
они являются настоящими анаэробными организмами, и в процессе дыхания
окисляют различные органические вещества, а восстанавливают металлы (
metallireducens) (рис.1).[1]
Систематическое положение организма приведено в табл. 1. [3]
Desulfitobacterium metallireducens являются настоящими анаэробными
прокариотами. Бактерии также обладают следующими свойствами:
Грам-отрицательные, подвижные, образуют споры, выглядят как дугообразные
палочки размером 0,5 на 2-3 µm (рис. 2), являются мезофилами: оптимальная
температура для жизнедеятельности около 30 oC. Оптимальным pH для них
считается pH близкий к нейтральному (7). [1]
, [2]
Эти бактерии были впервые выделены из загрязненной ураном реки San Juan
(Shiprock, New Mexico, United States) в январе 2000 года K. T. Finneran’ом.
Ее геном состоит из одной хромосомы размеров 3176073 bp.
[2] В нынешней
работе кратко описан геном NZ_CP007032.1 (идентификатор записи с геномом
данной бактерии).
МАТЕРИАЛЫ И МЕТОДЫ
В процессе работы активно использовались электронные таблицы google sheet (
ЭТ) и команды на сервере kodomo.fbb.msu.ru,
а также сервис для определения мест начала и конца репликации в геноме: http://genskew.csb.univie.ac.at/
.
Из директории сайта NCBI с данными по бактерии были скачаны следующие
файлы: [4]
GCF_000231405.2_ASM23140v3_feature_table.txt.gz - геном бактерии, в виде,
который удобно импортировать в google sheets.
GCF_000231405.2_ASM23140v3_genomic.fna.gz - геном бактерии в расширении
fna, который можно конвертировать в fasta.
Команды на kodomo:
1. “wordcount -wordsize 1” - для подсчета нуклеотидов, из которых
состоит ДНК.
2. ”geecee” - вычисление частоты комплементарной пары G-С в геномной
ДНК.
3. “wordcount -wordsize 2” и “cbcalc -s word-2 -K” - команды были
использованы для расчета количества димеров в геноме, а также для расчета
отношения наблюдаемого количества димера к ожидаемому.
Функции, которые были задействованы в ЭТ:
1. Импортирование данных из txt-файла
2. Фильтр строк по значению
3. Создание новых листов, их связь между собой
4. Копирование и вставка (В том числе специальная вставка по значениям)
5. Перенос данных между листами с помощью функции ВПР
6. Сортировка
7. Оформление таблицы
8. Использование $ для распространения формул
9. Использование функций СЧЁТЕСЛИМН, МАКС и МИН
10. Построение диаграмм и их оформление
11. Функция СРЗНАЧ
12. Поиск по значению
13. Экранирование спец. символов в начале строки
14. “Сводная таблица”
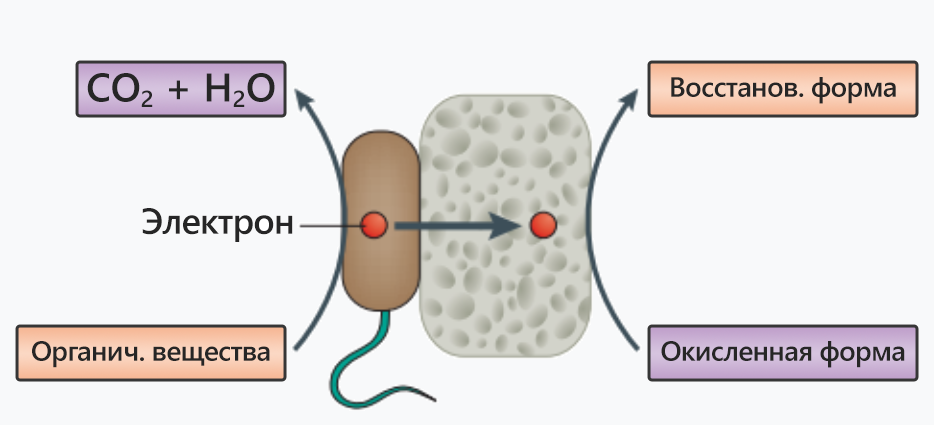
Рис. 1
Схема процесса дыхания.[2]
| Таксон | Название | ||
|---|---|---|---|
| Домен | Bacteria | ||
| Группа | Terrabacteria group | ||
| Тип | Firmicutes | ||
| Класс | Clostridia | ||
| Порядок | Clostridiales | ||
| Семейство | Peptococcaceae | ||
| Род | Desulfitobacterium | ||
| Вид | Desulfitobacterium metallireducens | ||
| Штамм | 15288 |
Табл. 1
Систематическое положение Desulfitobacterium
metallireducens DSM 15288
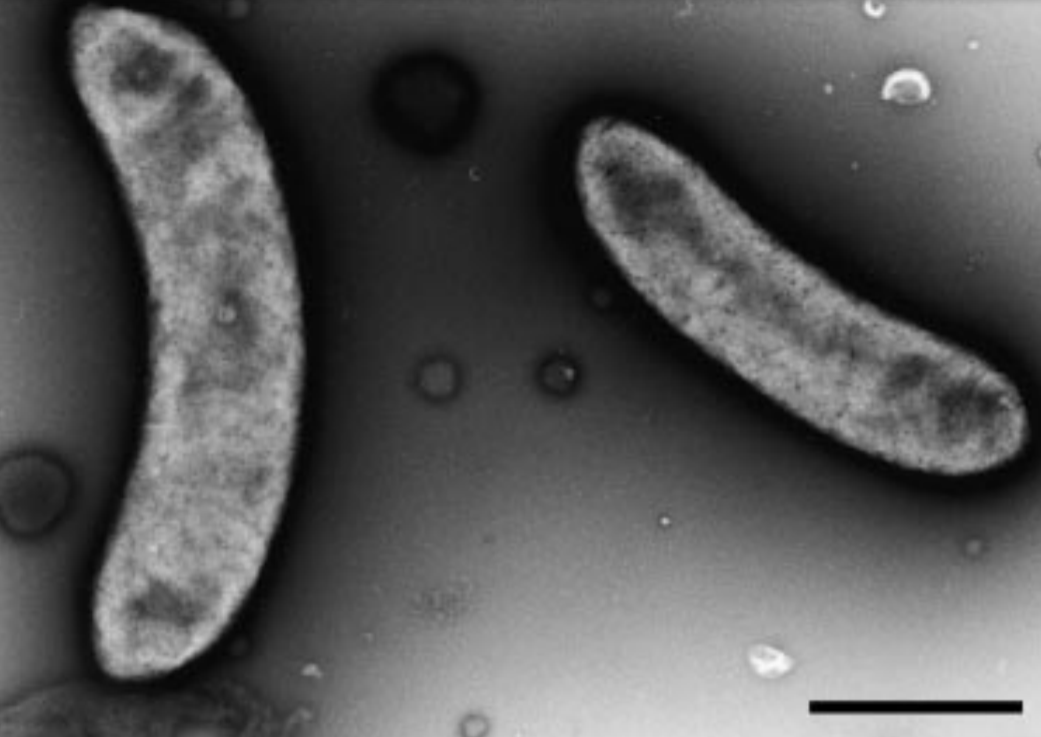
Рис. 2
Электронная микрофотография DSM 15288, на которой видна
типичная дугообразная форма данных бактерий. Полоса, 1 µm.
[1]
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Состав генома
Состав генома DSM 15288 представлен в таблице 2.
Как видно из таблицы 2, количество нуклеотидов A (аденин) примерно равно T (тимин), G (гуанин) примерно равно C (цитозин), а значит можно считать выполненным второе правило Чаргаффа.
Отклонение количества нуклеотида от идеального равенства между комплементарными парами A-T составляет 2418 нуклеотидов, а между G-C 8961 нуклеотид.
| Нуклеотид | Количество | ||
|---|---|---|---|
| A | 924473 | ||
| T | 921053 | ||
| G | 671610 | ||
| C | 658937 |
Табл. 2
Типы нуклеотидов в геноме бактерии, количество каждого нуклеотида
Типы генов
В таблице 3 наглядно показано, что геном состоит из 3105 генов, при этом 1719 генов представлены на обратной цепи ДНК, а 1386 на прямой. Очевидно, что на обратной цепи
генов больше, чем на прямой, однако, это может быть связано с тем, что на обратной цепи кодируются структуры, которые требуют большого количества генов, к примеру жгутик,
NADH-хинон оксидоредуктаза или кластер генов, который отвечает за кодировку тРНК для практически всех аминокислот, а также АТФ-синтаза. Также интересными являются гены,
которые хранят информацию про 5S, 16S, 23S рРНК. Все три гена кодируются друг за другом в порядке 16S, 23S, 5S на прямой цепи и, соответветственно, 5S, 23S, 16S на обратной
цепи. Всего в геноме встречаются 8 таких участков (см. Gukov_suppl, лист “somethings_from_genome”). Образование участков можно объяснить тем, что рРНК нужны только для
постройки рибосомы, в свою очередь она нуждается в каждом типе рРНК, это значит, что бактерии не выгодно экспрессировать только один тип рибосомальных РНК, потому что без
двух других она бесполезна, поэтому все три рРНК кодируются в непосредственной близости относительно друг друга. Нельзя не отметить присутствие в геноме псевдогенов.
Псевдоген - это аналог генов, который утратил свою функцию, а это значит, что они утратили способность экспрессировать что-то в бактерии. В геноме DSM наблюдается 34
псевдогена, то есть 1,1% генома. Возможно, в ходе эволюции у бактерии были белки, которые были полезными, но не были необходимыми для выживания, в случае мутации внутри
гена, который кодировал такой белок, он терял возможность к экспрессии, но при этом оставался в геноме. Так как без этого белка можно было жить, вид продолжил свое развитие.
| Класс | Обратная цепь | Прямая цепь | Обе | |||
|---|---|---|---|---|---|---|
| Число генов | Процент от цепи | Число генов | Процент от цепи | Число генов | Процент от цепей | |
| ncRNA | 1 | 0.07% | 1 | 0.03% | ||
| protein_coding | 1639 | 95.35% | 1331 | 96.03% | 2970 | 95.65% |
| pseudogene | 19 | 1.11% | 15 | 1.08% | 34 | 1.10% |
| RNase_P_RNA | 1 | 0.06% | 1 | 0.03% | ||
| rRNA | 9 | 0.52% | 15 | 1.08% | 24 | 0.77% |
| SRP_RNA | 1 | 0.07% | 1 | 0.03% | ||
| tmRNA | 1 | 0.06% | 1 | 0.03% | ||
| tRNA | 50 | 2.91% | 23 | 1.66% | 73 | 2.35% |
| Итого | 1719 | 100.00% | 1386 | 100.00% | 3105 | 100.00% |
Табл. 3
Типы генов в геноме DSM 15288 и их количество на обратной (2 и 3 столбцы, прямой (4 и 5 столбцы) и обеих цепях (6 и 7 столбцы). А также процент количества каждого типа гена
от общего количества генов на каждой цепи и обеих цепях.
Места начала и конца репликации
Репликация - процесс удвоения ДНК. В случае прокариотических организмов, ДНК имеет одно место начала репликации, то есть место, куда прикрепляется ДНК-зависимая ДНК-полимераза для копирования молекулы, а также одно место конца репликации, где ДНК-зависимая ДНК-полимераза заканчивает свою работу. Найти место прикрепления ДНК-полимеразы можно основываясь на неоднородности соотношения гуанинов G и цитозинов C внутри одной цепи ДНК. С одной стороны от места начала репликации число G на 1000 нуклеотидов меньше числа C на 1000 нуклеотидов (в среднем). А с другой стороны наоборот. На этом основан алгоритм вычисления GC-skew,в сервисе http://genskew.csb.univie.ac.at/. место начала репликации соответствует минимуму GC-skew cumulative, а место конца - максимуму (рис. 3). [4] Как можно видеть по графику, точка минимума находится в окрестности последнего нуклеотида, а это значит, что геном бактерии в формате .fasta начинается места начала репликации. Точка максимума находится в окрестности 1540361-ого нуклеотида, что соответсвует месту окончания репликации.
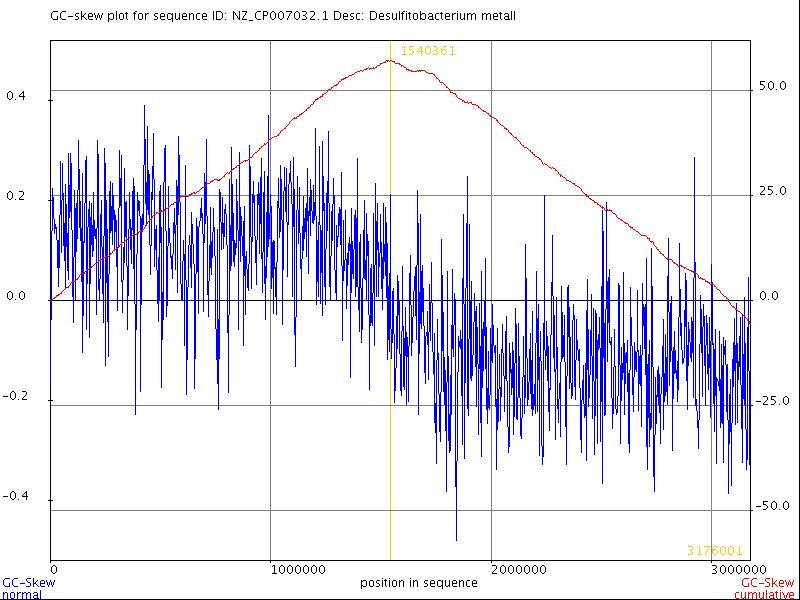
Рис. 3
График, который показывает неоднородность числа гуанинов G и цитозинов C внутри матричной цепи ДНК.
Типы генов
В таблице 3 наглядно показано, что геном состоит из 3105 генов, при этом 1719 генов представлены на обратной цепи ДНК, а 1386 на прямой. Очевидно, что на обратной цепи генов больше, чем на
прямой, однако, это может быть связано с тем, что на обратной цепи кодируются структуры, которые требуют большого количества генов, к примеру жгутик, NADH-хинон оксидоредуктаза или кластер
генов, который отвечает за кодировку тРНК для практически всех аминокислот, а также АТФ-синтаза. Также интересными являются гены, которые хранят информацию про 5S, 16S, 23S рРНК. Все три гена
кодируются друг за другом в порядке 16S, 23S, 5S на прямой цепи и, соответветственно, 5S, 23S, 16S на обратной цепи. Всего в геноме встречаются 8 таких участков (см. Gukov_suppl, лист
“somethings_from_genome”). Образование участков можно объяснить тем, что рРНК нужны только для постройки рибосомы, в свою очередь она нуждается в каждом типе рРНК, это значит, что бактерии не
выгодно экспрессировать только один тип рибосомальных РНК, потому что без двух других она бесполезна, поэтому все три рРНК кодируются в непосредственной близости относительно друг друга.
Нельзя не отметить присутствие в геноме псевдогенов. Псевдоген - это аналог генов, который утратил свою функцию, а это значит, что они утратили способность экспрессировать что-то в бактерии. В
геноме DSM наблюдается 34 псевдогена, то есть 1,1% генома. Возможно, в ходе эволюции у бактерии были белки, которые были полезными, но не были необходимыми для выживания, в случае мутации
внутри гена, который кодировал такой белок, он терял возможность к экспрессии, но при этом оставался в геноме. Так как без этого белка можно было жить, вид продолжил свое развитие.
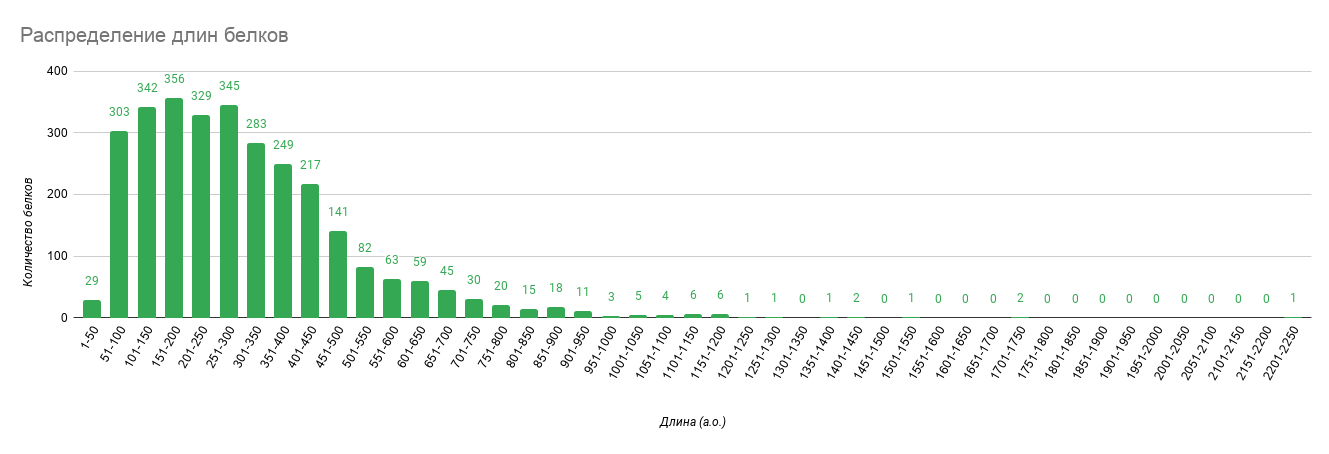
Рис. 4
Диаграмма распределения длин белков.
Межгенные промежутки
На рисунке 5 представлены межгенные промежутки на матричной цепи ДНК. Исходя из рисунка 5 можно сказать о том, что диапазон с наибольшим количество генов от 1 до 1000 нуклеотидов, в
котором находится 1009 генов. Стоит отметить, что 19 генов стыкуются друг с другом, то есть расстояние между ними равно 0 нуклеотидов, а также 112 генов имеют между собой пересечения, это
означает, что должны существовать системы транскрипции, которые отличают пересекающиеся гены между собой.
На рисунке 6 представлены межгенные промежутки на комплементарной цепи ДНК.
По рисунку 6 важно отметить, что, как и на матричной цепи, на комплементарной цепи диапазон от 1 до 1000 нуклеотидов содержит в себе большее количество генов среди всех диапазонов (1276
генов). Также 30 генов не имеют ни одного нуклеотида между собой, а еще 165 пересекаются.
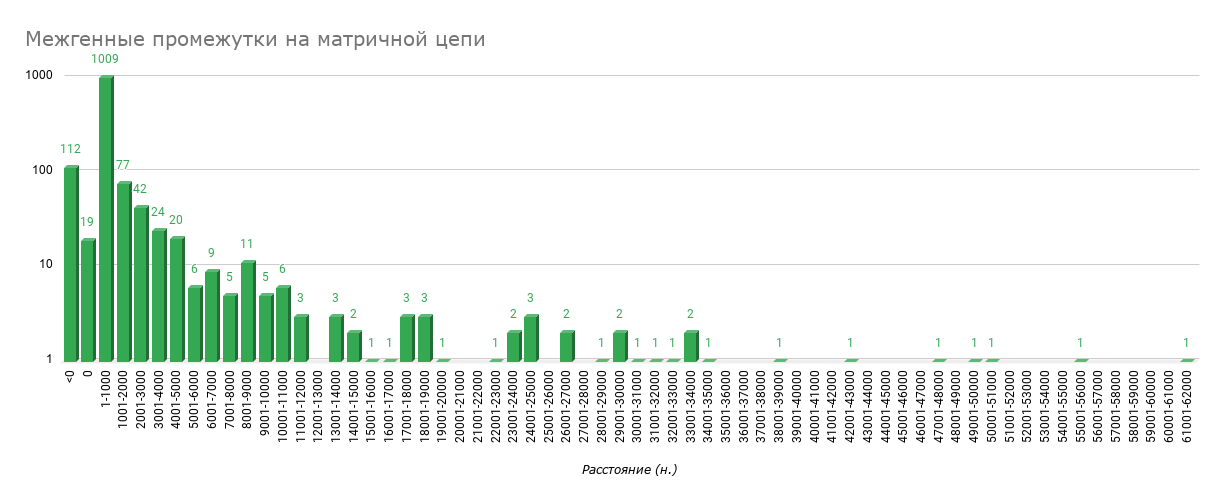
Рис. 5
Межгенные промежутки на матричной цепи ДНК в виде диаграммы, вычисленной по логарифмической шкале. Расстояние между генами измеряется в нуклеотидах (н.).
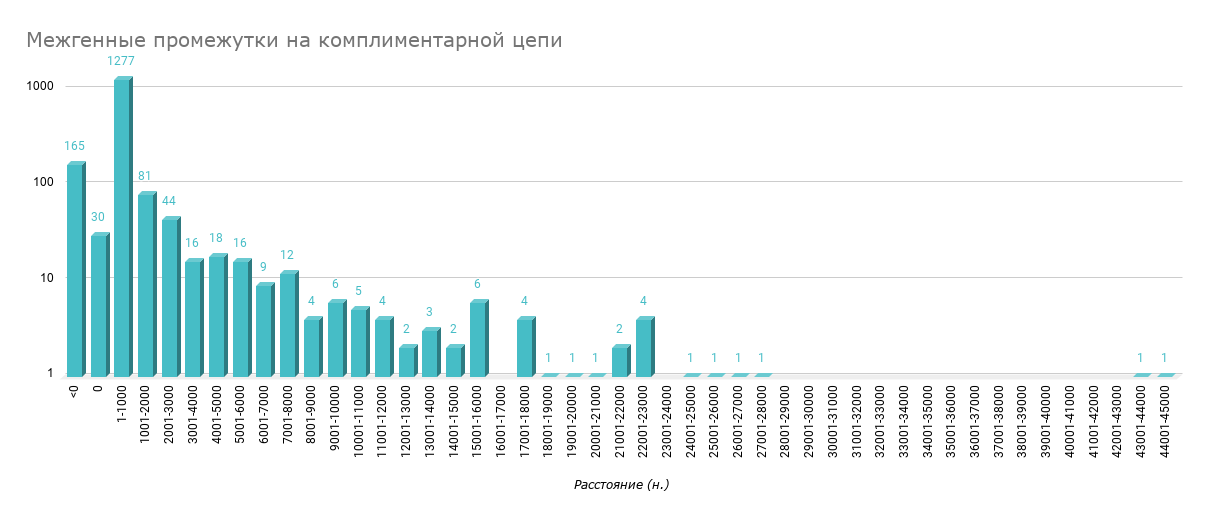
Рис. 6
Межгенные промежутки на комплементарной цепи ДНК в виде диаграммы, вычисленной по логарифмической шкале. Расстояние между генами измеряется в нуклеотидах (н.).
Ди-меры в геноме
Анализ ди-меров, которые содержаться в геноме, представлен на рисунке 7.
На рисунке 7 наблюдается тенденция того, что ди-меры, которые состоят из одинаковых нуклеотидов, встречаются более, чем на 10% чаще нежели ожидаемое количество, а наблюдаемое
количество GT, AC, TA более чем на 20% меньше, чем ожидалось. Анализ ди-меров можно использовать для сравнения геномов организмов между собой, для того, чтобы выстраивать филогенетические
деревья. Если одинаковые ди-меры имеют схожие частоты встречаемости, то можно говорить о том, что эти организмы произошли от одного предка.
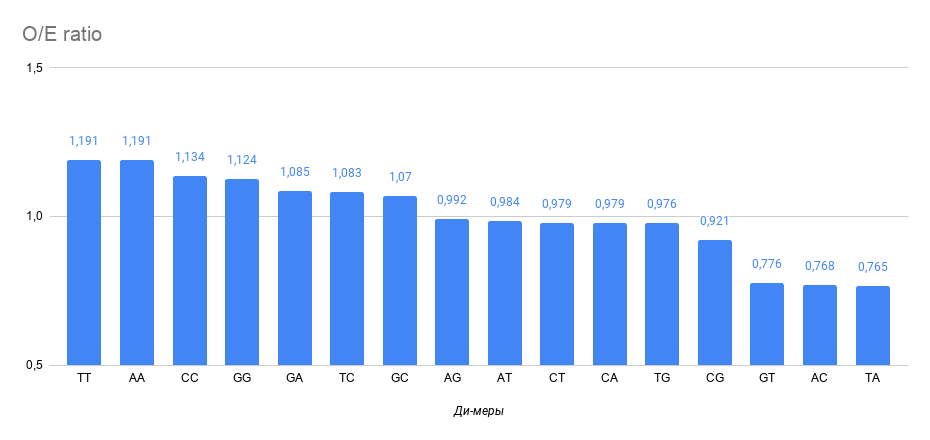
Рис. 7
Диаграмма отношений наблюдаемого количества (O, observed) ди-мера к ожидаемому (E, expected) количеству.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Папка с кратким обзором на google drive.
Таблица с исходными данными и их обработкой Gukov_suppl: table/Gukov_suppl
Геном: fasta/Gukov_genome.fasta
БЛАГОДАРНОСТИ
Я благодарен факультету биоинженерии и биоинформатики Московского государственного университета имени М. В. Ломоносова (ФББ МГУ) за возможность писать этот обзор.
ИЛЛЮСТРАЦИИ
1) Схема была преведена из статьи [2]
2) Микрофотография взята из исследования [1]
3) График был получен с помощью сервиса GC-skew http://genskew.csb.univie.ac.at/
4, 5, 6, 7) Рисунки были получены на основе данных из NCBI [4].
СПИСОК ЛИТЕРАТУРЫ
[1] - Kevin T. Finneran,† Heather M. Forbush, Catherine V. Gaw VanPraagh and Derek R. Lovley (2002). Desulfitobacterium metallireducens sp. nov., ananaerobic
bacterium that couples growth to the reduction of metals and humic acids as well as chlorinated compounds. International Journal of Systematic and Evolutionary Microbiology,
52, 1929–1935.
https://www.microbiologyresearch.org/docserver/fulltext/ijsem/52/6/0521929a.pdf?expires=1605692357&id=id&accname=guest&checksum=7E02C4AF5DD8E6AD13C07FE87A48ADB9
[2] - Liang Shi, Hailiang Dong, Gemma Reguera, Haluk Beyenal, Anhuai Lu, Juan Liu, Han-Qing Yu and James K. Fredrickson. Extracellular electron transfer
mechanisms between microorganisms and minerals. Nature, october 2016, 651-662.
https://drive.google.com/drive/
folders/184M_tTWuMM1hOXNo3PiFv1UX1heYioqF?usp=sharing
[3] - сайт NCBI - Desulfitobacterium metallireducens DSM 15288 chromosome, complete genome
https://www.ncbi.nlm.nih.gov/nuccore/NZ_CP007032.1.
[4] - сайт NCBI - директория с данными по бактерии DSM 15288.
ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/231/405/GCF_000231405.2_ASM23140