Для выполнения задания нужна была программа fiber, которая на вход принимает некоторые параметры (форма ДНК и последовательность), а выдает структуру ДНК данной формы в pdb. Однако, для Z-формы программа позволяет только повторять GC. Чтобы длины всех 3 цепей были одинаковыми, нужно было использовать 10 GC для построения Z-формы.
# Добавление значений переменных среды
%set_env PATH=/srv/conda/miniconda/bin:/srv/conda/miniconda/condabin:/usr/local/bin:/usr/bin:/home/preps/golovin/progs/x3dna-v2.4/bin
%set_env X3DNA=/home/preps/golovin/progs/x3dna-v2.4
env: PATH=/srv/conda/miniconda/bin:/srv/conda/miniconda/condabin:/usr/local/bin:/usr/bin:/home/preps/golovin/progs/x3dna-v2.4/bin env: X3DNA=/home/preps/golovin/progs/x3dna-v2.4
! fiber
===========================================================================
NAME
fiber - generate 56 fiber models based on Arnott and other's work
SYNOPSIS
fiber [OPTION] PDBFILE
DESCRIPTION
generate 56 fiber models based on the repeating unit from Arnott's
work, including the canonical A-, B-, C- and Z-DNA, triplex, etc.
-cif output structure coordinates in mmCIF format
-num a structure identification number in the range (1-56)
-m, -l brief description of the 56 fiber structures
-a, -1 A-DNA model (calf thymus)
-b, -4 B-DNA (calf thymus, default)
-c, -47 C-DNA (BII-type nucleotides)
-d, -48 D(A)-DNA poly d(AT) : poly d(AT) (right-handed)
-z, -15 Z-DNA poly d(GC) : poly d(GC)
-pauling triplex RNA model, based on Pauling and Corey
-rna RNA duplex with arbitrary base sequence
-seq=string specifying an arbitrary base sequence
-rep=number no. of repeats of the sequence specified via -seq
-single output a single-stranded structure
-h this help message (any non-recognized options will do)
INPUT
An structure identification number (or symbol)
EXAMPLES
fiber fiber-BDNA.pdb
# fiber -4 fiber-BDNA.pdb
# fiber -b fiber-BDNA.pdb
fiber -a fiber-ADNA.pdb
fiber -seq=AAAGGUUU -rna fiber-RNA.pdb
fiber -seq=AAAGGUUU -rna -single fiber-ssRNA.pdb
fiber -seq=AATTG -rep=5 B-AATTG-repeat5.pdb
# triplex model (RNA), based on Pauling and Corey
fiber -pauling triplex-C10C10C10.pdb # default: 10 Cs per strand
fiber -pauling -seq=AAA triplex-A3A3A3.pdb # 3 As per strand
fiber -pauling -seq=AAAA:CCCC:GGGG Pauling-triplex-A4C4G4.pdb
# triplex DNA model, with O2' atoms removed
fiber -pauling-dna -seq=ACTT -repeat=6 Pauling-DNA-triplex.pdb
OUTPUT
PDB file
SEE ALSO
analyze, anyhelix, find_pair
AUTHOR
3DNA v2.4.5-2021may19, created and maintained by xiangjun@x3dna.org
LICENSE
Creative Commons Attribution Non-Commercial License (CC BY-NC 4.0)
See http://creativecommons.org/licenses/by-nc/4.0/ for details
NOTE
*** 3DNA HAS BEEN SUPERSEDED BY DSSR ***
Please post questions/comments on the 3DNA Forum: http://forum.x3dna.org/
===========================================================================
# Команда для получения pdb A-формы ДНК
! fiber -a -seq=atgcatgcatgcatgcatgc src/fiber/atgc-a.pdb
Structure #1; Twist: 32.7 (degrees); Rise: 2.548 (Angstrom)
# Команда для получения pdb B-формы ДНК
! fiber -b -seq=atgcatgcatgcatgcatgc src/fiber/atgc-b.pdb
Structure #4; Twist: 36.0 (degrees); Rise: 3.375 (Angstrom)
# Команда для получения pdb Z-формы ДНК
! echo -n "10"| fiber -z src/fiber/atgc-z.pdb
Structure #15; Twist: -60.0 (degrees); Rise: 7.250 (Angstrom) Repeating unit: GC:GC Number of repeats (Dft: 10):
Задание выполнялось в PyMol над B-формой ДНК 1bna.pdb. Был выбран гуанин DG'4 для определения, того какие атомы смотрят в сторону большой бороздки, а какие атомы в сторону маленькой бороздки. На рисунке 1 представлена структура 1bna.pdb с выделеным DG'4.
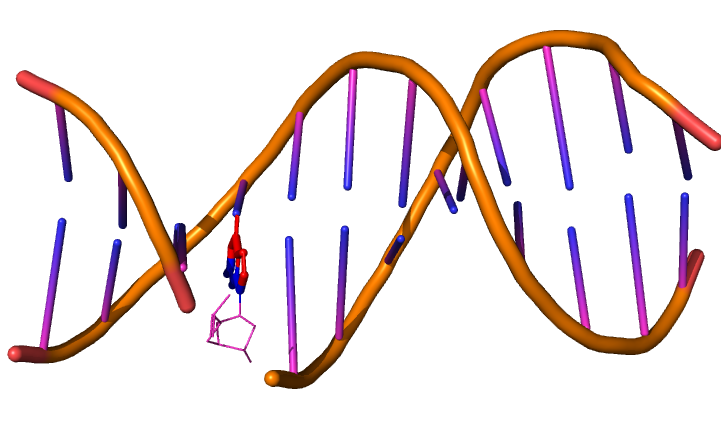
На рисунке 2 представлен гуанин с раскрасом по стороне, в которую смотрит атом. Если атом красный, то он смотрит в сторону большой бороздки, а если синий, то малой. Итого:
Красные:
N1, C5, C6, O6, N7, C8
Синие:
C2, N2, N3, C4, N9
В таблице 1 представлены основные парамеры каждой формы ДНК: тип спирали, шаг спирали, число оснований на виток, ширина большой бороздки, ширина малой бороздки.
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (Å) | 31 | 33.8 | 43.6 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 18.5 | 17.8 | 13.2 |
| Ширина малой бороздки | 9.6 | 9.9 | 9.9 |
import os
os.chdir('src//analyze//1ffy')
! find_pair -t 1ffy.pdb | analyze
os.chdir('..//..//..')
can not remove file: ref_frames.dat handling file <1ffy.pdb> Time used: 00:00:00:00
# Здесь какой-то непонятный код начинается. Идея в том, чтобы файлы *.out переделать в pandas.DataFrame и использовать seaborn,
# для построения распределения торсионных углов у 1ffy. А также для использования .mean(), чтобы посчитать среднее значение каждого типа угла.
import pandas as pd
import re
def mkdf(path):
with open(path + '.txt') as file:
with open(path + '_prob.txt', 'w') as out_file:
for line in file:
out_file.write(re.sub(r'\s+', ' ', line) + '\n')
data = pd.read_table(path + '_prob.txt', sep = ' ')
data = data.reset_index()
data = data.drop(['Unnamed: 9', 'index', 'id'], axis = 1).dropna()
return data
# Здесь программа группирует цепи 11ffy и описывает среднее значение по каждому углу.
ffy_data_1 = mkdf('src//analyze//1ffy//1ffy_strd1')
ffy_data_2 = mkdf('src//analyze//1ffy//1ffy_strd2')
ffy_data = pd.concat([ffy_data_1, ffy_data_2], ignore_index=True)
round(ffy_data.mean(), 2)
alpha -30.13 beta 35.25 gamma 39.39 delta 85.46 epsilon -131.73 zeta -51.93 chi -128.84 dtype: float64
На рисунке 3 представлены boxplot'ы для углов в структуре 1ffy. По рисунку видно, что все углы, кроме β имеют небольшой разброс, но, в основном, большое количество выпадов, в то время как угол β вообще не имеет выпадов, однако значения этого угла образует два кластера около 150 градусов и -150 градусов. На рисунке 4 изображены распределения для каждого каждого типа угла.
# Для работы seaborn удобно изпользовать long-form data (в одной строке полностью описано одно наблюдение). Чтобы переделать ffy_data нужно использовать
# .melt(), которая сдлеает long-form data.
# График - box plot'ы для каждого угла.
import seaborn as sns
ffy_data_melted = ffy_data.melt(id_vars=['base'],
var_name = 'angle', value_name = 'angle_value')
boxplots = sns.boxplot(x= 'angle', y = 'angle_value', data = ffy_data_melted, palette = 'Set3')
sns.stripplot(x= 'angle', y= 'angle_value', data = ffy_data_melted, size = 4, color = '.3')
<AxesSubplot:xlabel='angle', ylabel='angle_value'>
dist_angles = sns.displot( data=ffy_data_melted, x = 'angle_value', col = 'angle', kind = 'hist', height = 4, aspect=.7)
dist_angles.set_axis_labels('Градус', '')
<seaborn.axisgrid.FacetGrid at 0x7f74258e4ac8>
Судя по средним значениям тРНК 1ffy и данным по ДНКовым углам из лекции, была составлена таблица 2, в которой сравниваются углы 1ffy с A- и B-формами. Если угол не похож ни на A-, ни на B-, то тогда считается, что это не ДНКовый угол (1ffy не похожа на z-форму, это можно увидеть в PyMol). Таким образом, можно заключить, что структура тРНК 1ffy больше похожа на A-форму ДНК, нежели остальные формы.
| α | β | γ | δ | ε | ζ | χ | |
|---|---|---|---|---|---|---|---|
| 1ffy | не ДНК | не ДНК | A | A | не ДНК | A | B |
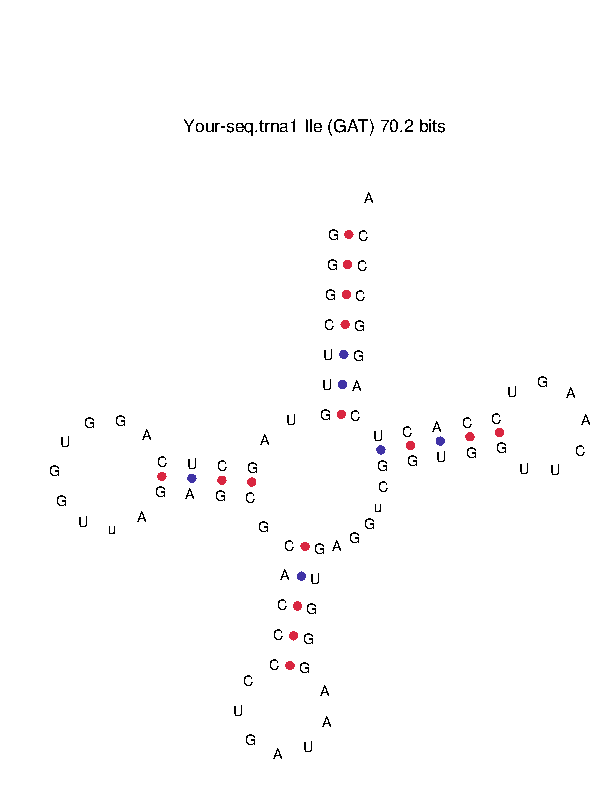
Внутри фигуры 1 представлен анализ вторичной структуры тРНК 1ffy. На рисунке 5 предсказана вторичная структура тРНК. Он был получен с помощью сервиса: http://lowelab.ucsc.edu/tRNAscan-SE/.
ФИГУРА 1.
Выдача программы analyze про водородные связи в тРНК 1ffy:
Strand I Strand II Helix
1 (0.003) ....>T:...1_:[..G]G-----C[..C]:..72_:T<.... (0.003) |
2 (0.008) ....>T:...2_:[..G]G-----C[..C]:..71_:T<.... (0.005) |
3 (0.007) ....>T:...3_:[..G]G-----C[..C]:..70_:T<.... (0.004) |
4 (0.009) ....>T:...4_:[..C]C-----G[..G]:..69_:T<.... (0.007) |
5 (0.007) ....>T:...5_:[..U]U-*---G[..G]:..68_:T<.... (0.009) |
6 (0.006) ....>T:...6_:[..U]U-----A[..A]:..67_:T<.... (0.004) |
7 (0.008) ....>T:...7_:[..G]G-----C[..C]:..66_:T<.... (0.002) |
8 (0.006) ....>T:..49_:[..G]G-*---U[..U]:..65_:T<.... (0.005) |
9 (0.005) ....>T:..50_:[..G]G-----C[..C]:..64_:T<.... (0.002) |
10 (0.003) ....>T:..51_:[..U]U-----A[..A]:..63_:T<.... (0.001) |
11 (0.004) ....>T:..52_:[..G]G-----C[..C]:..62_:T<.... (0.004) |
12 (0.007) ....>T:..53_:[..G]G-----C[..C]:..61_:T<.... (0.002) |
13 (0.005) ....>T:..54_:[..U]U-**--A[..A]:..58_:T<.... (0.007) |
14 (0.008) ....>T:..55_:[..U]U-**+-G[..G]:..18_:T<.... (0.011) x
15 (0.011) ....>T:..36_:[..U]U-**+-U[..U]:..33_:T<.... (0.005) |
16 (0.013) ....>T:..38_:[..A]A-**--C[..C]:..32_:T<.... (0.003) |
17 (0.012) ....>T:..39_:[..G]G-----C[..C]:..31_:T<.... (0.007) |
18 (0.004) ....>T:..40_:[..G]G-----C[..C]:..30_:T<.... (0.004) |
19 (0.004) ....>T:..41_:[..G]G-----C[..C]:..29_:T<.... (0.009) |
20 (0.007) ....>T:..42_:[..U]U-----A[..A]:..28_:T<.... (0.008) |
21 (0.007) ....>T:..43_:[..G]G-----C[..C]:..27_:T<.... (0.005) |
22 (0.009) ....>T:..44_:[..A]A-**--G[..G]:..26_:T<.... (0.011) |
23 (0.005) ....>T:..10_:[..G]G-----C[..C]:..25_:T<.... (0.005) |
24 (0.005) ....>T:..11_:[..C]C-----G[..G]:..24_:T<.... (0.010) |
25 (0.004) ....>T:..12_:[..U]U-----A[..A]:..23_:T<.... (0.008) |
26 (0.006) ....>T:..13_:[..C]C-----G[..G]:..22_:T<.... (0.004) |
27 (0.003) ....>T:..14_:[..A]A-**--U[..U]:...8_:T<.... (0.007) |
28 (0.006) ....>T:..15_:[..G]G-**+-C[..C]:..48_:T<.... (0.008) x
29 (0.005) ....>T:..16_:[..G]G-**--U[..U]:..17_:T<.... (0.003) +
30 (0.008) ....>T:..19_:[..G]G-----C[..C]:..56_:T<.... (0.004) +
Неканонические взаимодействия:
5 (0.007) ....>T:...5_:[..U]U-*---G[..G]:..68_:T<.... (0.009) |
8 (0.006) ....>T:..49_:[..G]G-*---U[..U]:..65_:T<.... (0.005) |
14 (0.008) ....>T:..55_:[..U]U-**+-G[..G]:..18_:T<.... (0.011) x
15 (0.011) ....>T:..36_:[..U]U-**+-U[..U]:..33_:T<.... (0.005) |
16 (0.013) ....>T:..38_:[..A]A-**--C[..C]:..32_:T<.... (0.003) |
22 (0.009) ....>T:..44_:[..A]A-**--G[..G]:..26_:T<.... (0.011) |
29 (0.005) ....>T:..16_:[..G]G-**--U[..U]:..17_:T<.... (0.003) +
Стебли РНК:
1 (0.003) ....>T:...1_:[..G]G-----C[..C]:..72_:T<.... (0.003) |
2 (0.008) ....>T:...2_:[..G]G-----C[..C]:..71_:T<.... (0.005) |
3 (0.007) ....>T:...3_:[..G]G-----C[..C]:..70_:T<.... (0.004) |
4 (0.009) ....>T:...4_:[..C]C-----G[..G]:..69_:T<.... (0.007) |
5 (0.007) ....>T:...5_:[..U]U-*---G[..G]:..68_:T<.... (0.009) |
6 (0.006) ....>T:...6_:[..U]U-----A[..A]:..67_:T<.... (0.004) |
7 (0.008) ....>T:...7_:[..G]G-----C[..C]:..66_:T<.... (0.002) |
23 (0.005) ....>T:..10_:[..G]G-----C[..C]:..25_:T<.... (0.005) |
24 (0.005) ....>T:..11_:[..C]C-----G[..G]:..24_:T<.... (0.010) |
25 (0.004) ....>T:..12_:[..U]U-----A[..A]:..23_:T<.... (0.008) |
26 (0.006) ....>T:..13_:[..C]C-----G[..G]:..22_:T<.... (0.004) |
17 (0.012) ....>T:..39_:[..G]G-----C[..C]:..31_:T<.... (0.007) |
18 (0.004) ....>T:..40_:[..G]G-----C[..C]:..30_:T<.... (0.004) |
19 (0.004) ....>T:..41_:[..G]G-----C[..C]:..29_:T<.... (0.009) |
20 (0.007) ....>T:..42_:[..U]U-----A[..A]:..28_:T<.... (0.008) |
21 (0.007) ....>T:..43_:[..G]G-----C[..C]:..27_:T<.... (0.005) |
8 (0.006) ....>T:..49_:[..G]G-*---U[..U]:..65_:T<.... (0.005) |
9 (0.005) ....>T:..50_:[..G]G-----C[..C]:..64_:T<.... (0.002) |
10 (0.003) ....>T:..51_:[..U]U-----A[..A]:..63_:T<.... (0.001) |
11 (0.004) ....>T:..52_:[..G]G-----C[..C]:..62_:T<.... (0.004) |
12 (0.007) ....>T:..53_:[..G]G-----C[..C]:..61_:T<.... (0.002) |
Стабилизация третичной структуры: (Данные были проврены в ручную при помощи PyMol)
13 (0.005) ....>T:..54_:[..U]U-**--A[..A]:..58_:T<.... (0.007) |
14 (0.008) ....>T:..55_:[..U]U-**+-G[..G]:..18_:T<.... (0.011) x
15 (0.011) ....>T:..36_:[..U]U-**+-U[..U]:..33_:T<.... (0.005) |
16 (0.013) ....>T:..38_:[..A]A-**--C[..C]:..32_:T<.... (0.003) |
22 (0.009) ....>T:..44_:[..A]A-**--G[..G]:..26_:T<.... (0.011) |
27 (0.003) ....>T:..14_:[..A]A-**--U[..U]:...8_:T<.... (0.007) |
28 (0.006) ....>T:..15_:[..G]G-**+-C[..C]:..48_:T<.... (0.008) x
29 (0.005) ....>T:..16_:[..G]G-**--U[..U]:..17_:T<.... (0.003) +
30 (0.008) ....>T:..19_:[..G]G-----C[..C]:..56_:T<.... (0.004) +.gif файлы моделировались в PyMol, пример кода ниже. За склеивание кучи .png в .gif благодарен сервису https://imgflip.com
import os, sys
import __main__
__main__.pymol_argv = [ 'pymol', '-cp' ]
import pymol
pymol.finish_launching()
from pymol import cmd
cmd.load('atgc-a.pdb')
cmd.do('''
hide everything
show cartoon
set antialias, 2
ray 600,600
mset 1x120
util.mroll(1,120,1)
mpng mov
mclear
''')