Далее дерево было перенесено в iTOL для выделения ортологов и паралогов.
На картинке ниже представлена визуализация дерева с выделением групп белков ATP-dependent Clp protease (CLPX - красный и CLPC - синий) и ATP-dependent zinc metalloprotease (FtsH - зеленый). Также выделены ортологи.
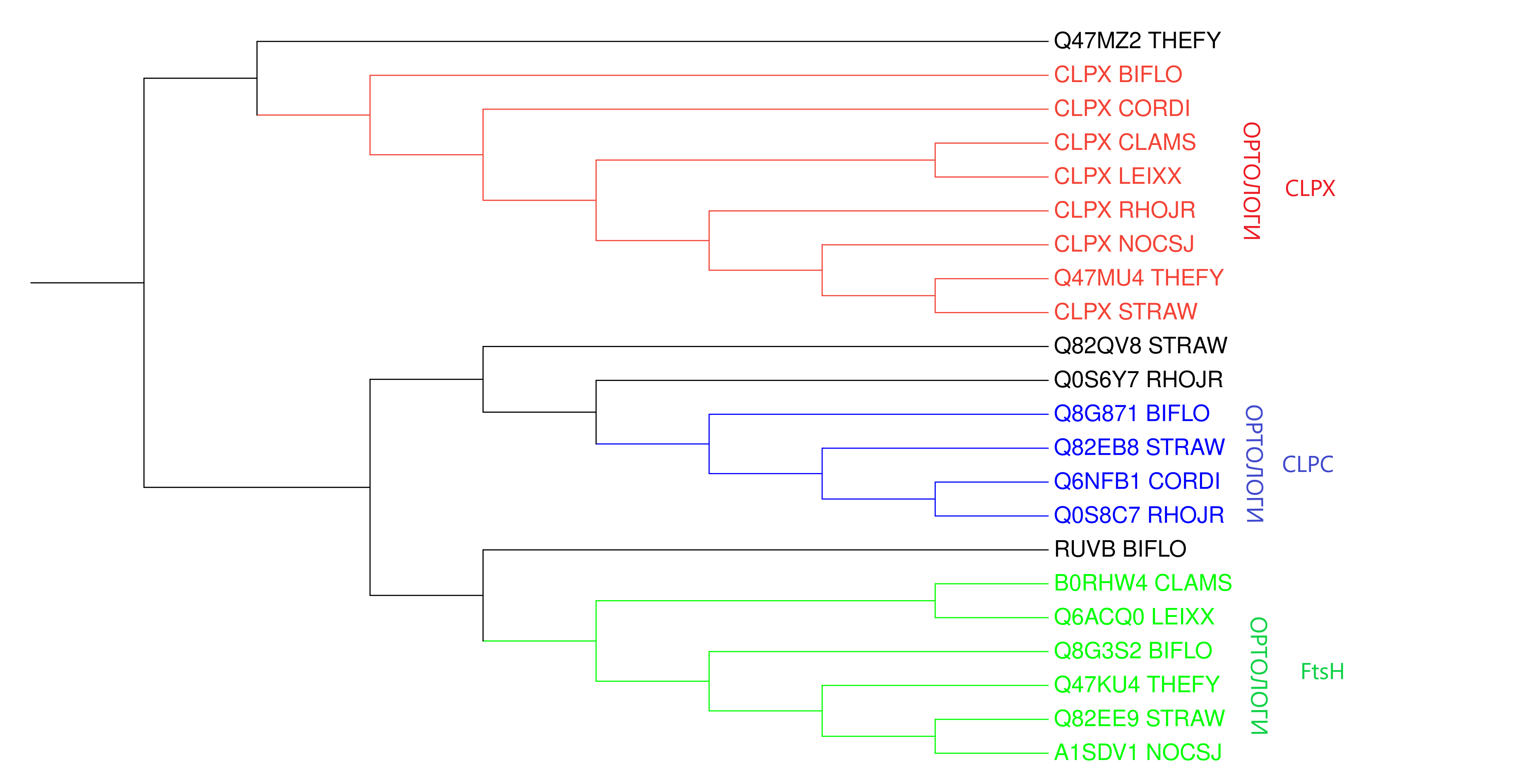
К примеру, можно указать по 3 пары ортологов и паралогов:
- Паралоги:
- Q82EB8_STRAW и CLPX_STRAW
- Q6NFB1_CORDI и CLPX_CORDI
- Q0S8C7_RHOJR и CLPX_RHOJR
- Ортологи:
- CLPX_CLAMS и CLPX_LEIXX
- Q47MU4_THEFY и CLPX_STRAW
- Q82EE9_STRAW и A1SDV1_NOCSJ
Далее представлена картинка с "схлопнутыми" ортологичными группами.
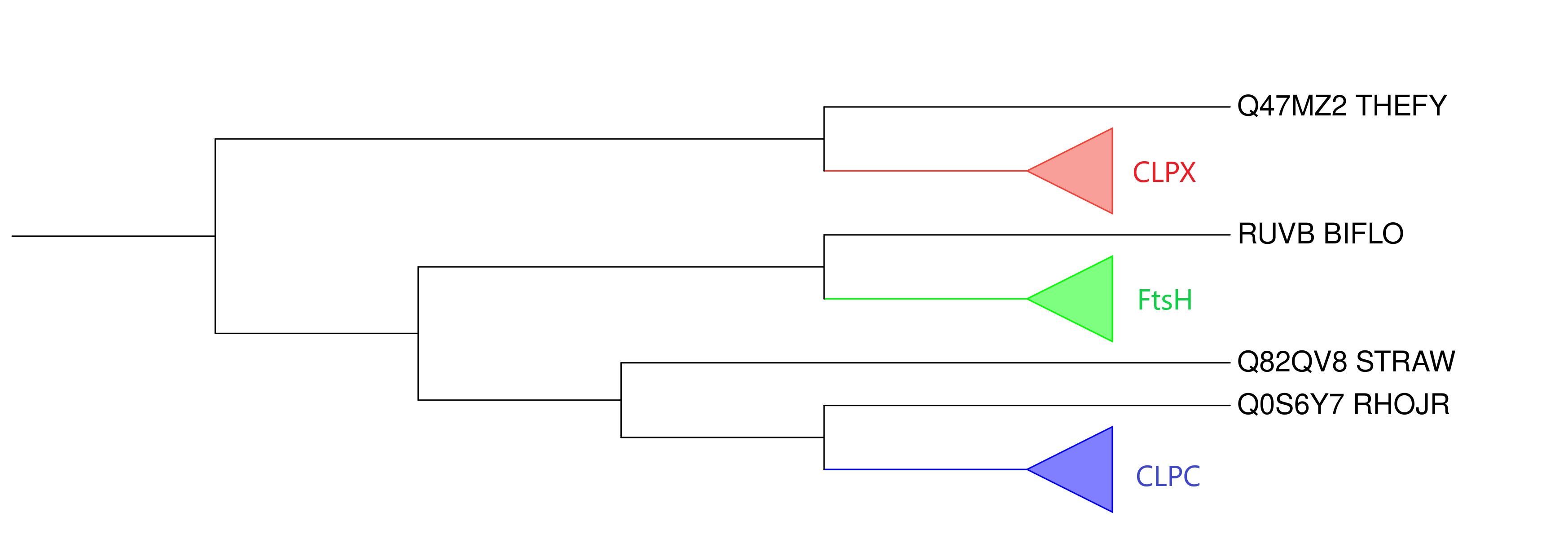
Для описания отличий реконструированных деревьев ортологических групп от дерева бактерий была составлена таблица. В первом столбце представлены нетривиальные ветви дерева бактерий, а в колонках - вхождение (+) или невхождение (-) ветви эталонного дерева в соответствующую ортологическую группу.
| Нетривиальные ветви эталона | CLPX | CLPC | FtsH |
|---|---|---|---|
| {STRAW, THEFY} vs {NOCSJ, LEIXX, CLAMS, BIFLO, RHOJR, CORDI} | + | - | - |
| {STRAW, THEFY, NOCSJ} vs {LEIXX, CLAMS, BIFLO, RHOJR, CORDI} | + | - | + |
| {LEIXX, CLAMS} vs {BIFLO, NOCSJ, STRAW, THEFY, RHOJR, CORDI} | + | - | + |
| {LEIXX, CLAMS, BIFLO} vs {NOCSJ, STRAW, THEFY, RHOJR, CORDI} | - | - | - |
| {RHOJR, CORDI} vs {BIFLO, LEIXX, CLAMS, NOCSJ, STRAW, THEFY} | - | + | - |
По таблице видно, что группа CLPX имеет большее количество нетривиальных ветвей (3 ветви из 5) исходного дерева, нежли остальные группы. Группа CLPC показала худший результат, однако это единственная группа, которая содержит ветвь (RHOJR, CORDI). Можно сказать, что топология каждого из деревьев, постороенных на основе ортологичных групп, отличается от исходной топологии.