OPM¶
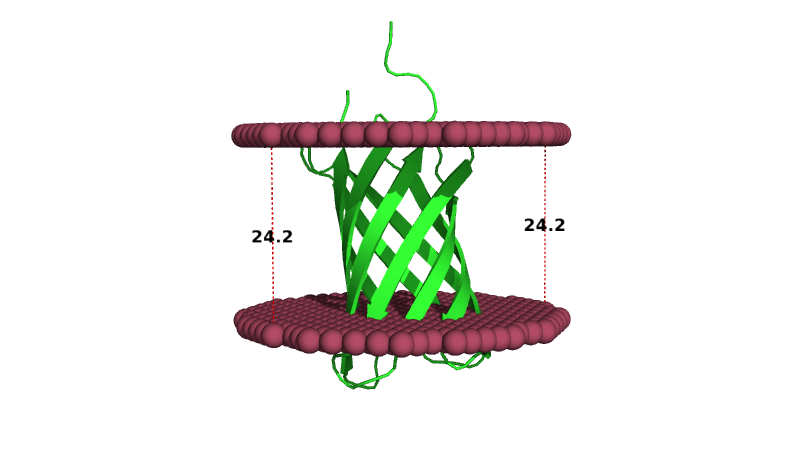
Для работы был выбран задезайненный трансмембранный β-бачонок TMB2.3. На uniprot ссылка отсутсвует. Индефикатор pdb 6x1k
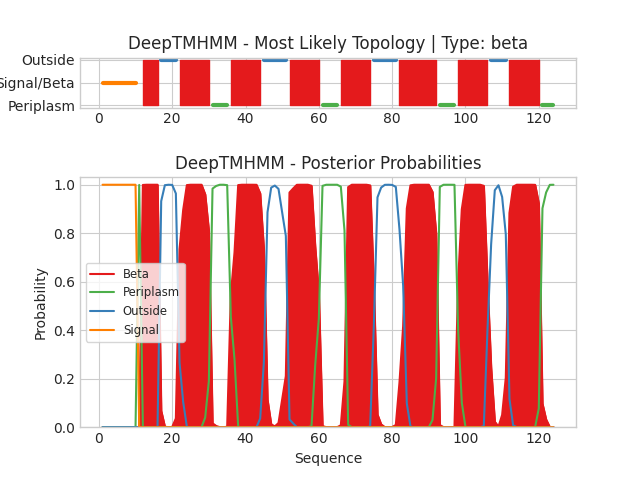
Толщина гидрофобной части была измерена в программе PyMol и составила 24.2 Å. В среднем, один β-тяж состоит из 8 аминокислотных остатков. Координаты трасмембранных участков вторичных структур представлены ниже:
1( 9- 15), 2( 23- 31), 3( 38- 43), 4( 50- 60), 5( 66- 75), 6( 82- 93), 7( 100- 106), 8( 114- 122)Толщина гидрофобной части 24.2 Å
Угол изгиба 14°
ΔG -28.5 ккал/моль
Топология субъединица А (внеклеточный N-конец)
Качество ЯМР
PDB Индефикатор 6x1k
Количство вторичных ТМ структур 8
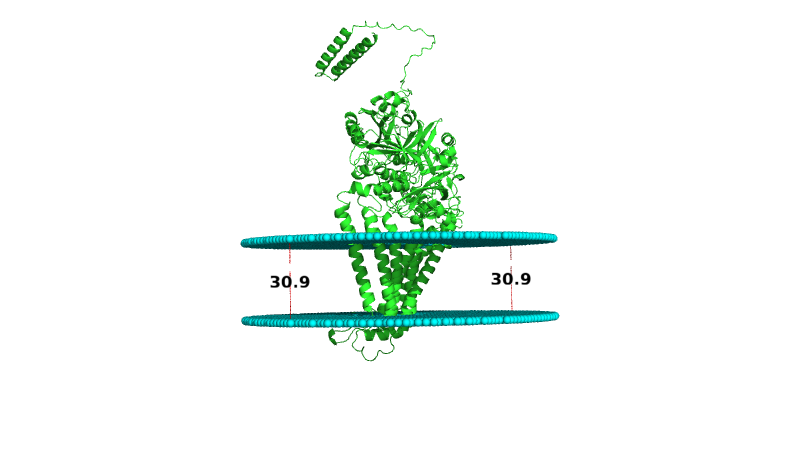
Uniprot :(На картинке представлены измерения трансмембранного участка. p-сторона направлена вниз.