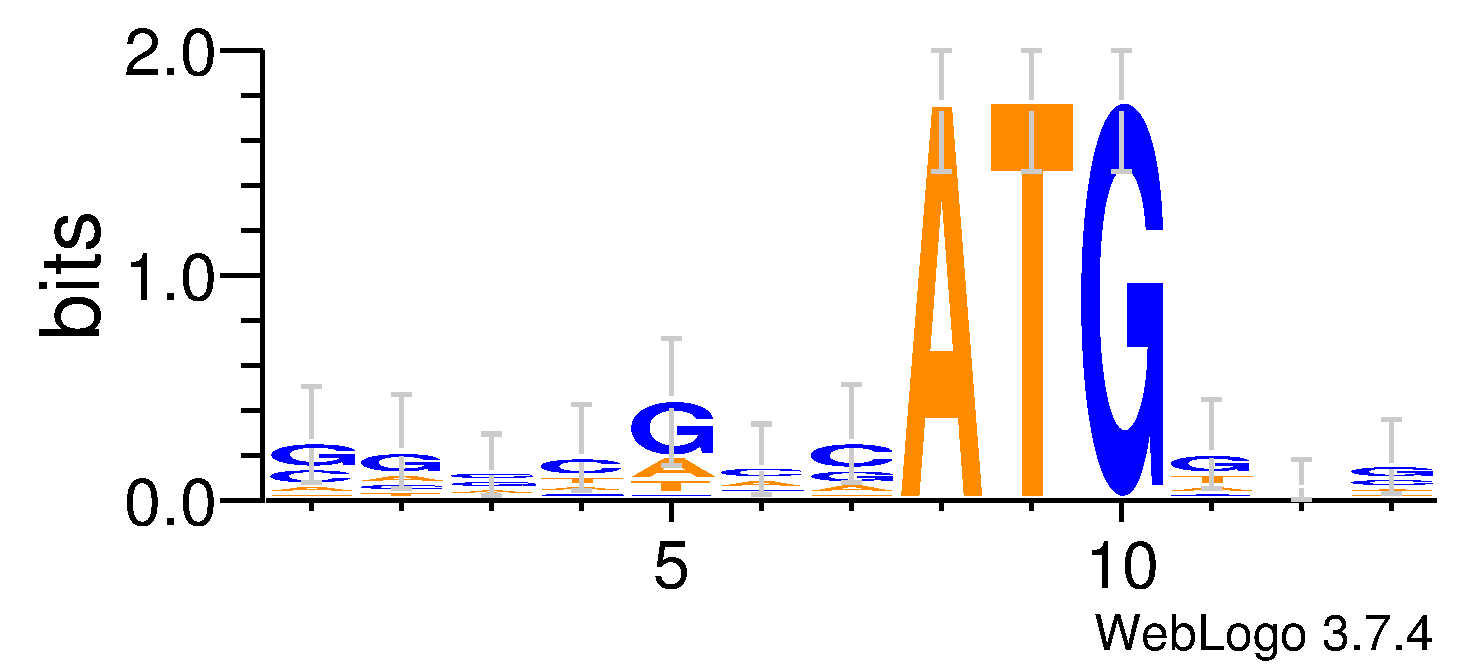
С помощью сервиса WebLogo была получена следующая визуализация:
NER - Nucleotide Excision Repair (эксцизионная репарация нуклеотидов) - один из механизмов репарации ДНК. Он способен чинить одну нить ДНК на матрице комплиментарной цепи. Его основная задача чинить такие повреждения, которые изменяют форму ДНК, из-за чего BER не способен распознать повреждение. Т. е. сигналом к репарации через NER служит изменение формы ДНК (к примеру, тиминовые димеры). У прокариот NER'ом занимаются белки семейтсва Uvr. Собственно изменение формы узнается белком UvrA, который в свою очередь подзывает другие белки семейства (B, C), тем самым запуская репарацию. По моим предположениям, сигнал является достаточно эффективным, потому что если бы сигнал недостаточным, то репарация происходила бы реже, из-за чего всё было бы плохо.
Источник - курс прошлого семестра по молекулярной биологии.
Удобно работать в jupyter lab, однако есть этот же код в формате py.
import pandas as pd
import requests, sys
import plotly.graph_objects as go
import plotly.io as pio
import random
from numpy import transpose as tp
from numpy import log, log2
from scipy.stats import mannwhitneyu
from Bio import SeqIO
pio.renderers.default = 'iframe'
Были установлены следующие параметры скрипта:
# PARAMS
seed = 27032022
gc = 0.404
psevd = 0.1
proba = {'A': (1-gc)/2, 'G': gc/2, 'T': (1-gc)/2, 'C': gc/2}
PATH_SRC = './src/'
PATH_OUT = './result/'
proba # ОЖИДАЕМЫЕ ЧАСТОТЫ
{'A': 0.298, 'G': 0.202, 'T': 0.298, 'C': 0.202}
data = pd.read_csv(PATH_SRC+'data.tsv', sep = '\t')
# RECORDS COLLECTION
random.seed(seed)
records = []
counter = 0
while True:
i = random.choice(data.index)
chrom = data.loc[i, 'chrom']
ATG = data.loc[i, 'thickStart']
ATG_revers = data.loc[i, 'thickEnd']
strand = data.loc[i, 'strand']
server = 'https://rest.ensembl.org'
if strand == '+':
ext = f'/sequence/region/human/{chrom}:{ATG-6}..{ATG+6}:1?expand_3prime=0;expand_5prime=0'
elif strand == '-':
ext = f'/sequence/region/human/{chrom}:{ATG_revers-5}..{ATG_revers+7}:-1?expand_3prime=0;expand_5prime=0'
r = requests.get(server+ext, headers={ "Content-Type" : "text/x-fasta"})
if not r.ok:
r.raise_for_status()
sys.exit()
seq = r.text.split('\n')[1]
if seq[7:10] == 'ATG':
counter += 1
records.append(seq)
if counter % 10 == 0:
print(f'Collected {counter} seqs')
if counter == 100:
break
Collected 10 seqs Collected 20 seqs Collected 30 seqs Collected 40 seqs Collected 50 seqs Collected 60 seqs Collected 70 seqs Collected 70 seqs Collected 80 seqs Collected 80 seqs Collected 90 seqs Collected 100 seqs
(Некоторые строки выше повторяются. Это не баг. Это фича :D)
На данном этапе были получаены 100 последовательностей, состоящих из 7 нуклеотидов до ATG + ATG + 3 нуклеотида после ATG. Последовательности, которые не содержат ATG в нужной позиции игнорировались.
После этого набор последовательностей был разбит на обучающие (n=40) и тестовые (n=60).
# TRAIN TEST SPLIT
train, test = records[60:], records[:60]
N = len(train)
len(records), len(train), len(test)
(100, 40, 60)
Ниже приведен код, который строит матрицу PWM по тренировочную набору, с псевдокаунтами равными 0.1 и GC-составом 40.4%.
# PWM
pwm_dict = {'A': [], 'G': [], 'T': [], 'C': []}
transposed = tp(list(map(lambda x: [i for i in x], train)))
df = pd.DataFrame(transposed)
for i in df.index:
values = df.iloc[i, :].value_counts()
for k, v in proba.items():
try:
pwm_dict[k].append(round(log((values.loc[k] + psevd)/(N + 4*psevd)/v), 3))
except KeyError:
pwm_dict[k].append(round(log((0 + psevd)/(N + 4*psevd)/v), 2))
pwm = tp(pd.DataFrame(pwm_dict))
pwm
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.680 | -0.396 | -0.176 | -0.528 | -0.280 | 0.005 | -0.528 | 1.203 | -4.790 | -4.790 | -0.528 | -0.176 | -0.528 |
| G | 0.797 | 0.850 | 0.394 | -0.291 | 1.040 | -0.139 | 0.213 | -4.400 | -4.400 | 1.592 | 0.797 | 0.109 | 0.679 |
| T | -1.077 | -0.680 | -0.859 | -0.280 | -0.680 | -0.680 | -1.077 | -4.790 | 1.203 | -4.790 | -0.176 | -0.280 | -0.680 |
| C | 0.394 | -0.139 | 0.473 | 0.797 | -1.357 | 0.615 | 0.850 | -4.400 | -4.400 | -4.400 | -0.470 | 0.394 | 0.308 |
После построения матрицы был проведен позитивный и негативный контроль.
Для положительного котроля были взяты тестовые последовательности.
# POSITIVE CONTROL
test_list = list(map(lambda x: [i for i in x], test))
positive = []
for i in test_list:
positive.append(sum([pwm.loc[j].iloc[k] for k, j in enumerate(i)]))
Для отрицательного контроля были взяты последовательности вокруг ATG (7+3+3) из генома sars-CoV-2, которые не являются инициаторными кодонами.
# NEGATIVE CONTORL
neg_df = pd.read_csv(PATH_SRC+'sarsatg.tsv', sep = '\t')
random.seed(seed)
clean_negative = neg_df[(neg_df.Strand == '+') & (neg_df.Pattern != 'start-tr')].reset_index(drop = True)
negative_random = random.choices(clean_negative.index, k = 60)
coords_atg = clean_negative.iloc[negative_random, [0, 1]].reset_index(drop = True)
coords_atg.Start = coords_atg.Start - 8
coords_atg.End = coords_atg.End + 3
iterator = SeqIO.parse(PATH_SRC+'sars.fasta', 'fasta')
genome = next(iterator)
negative_seqs = []
for i in coords_atg.index:
negative_seqs.append(genome.seq[int(coords_atg.iloc[i, 0]):int(coords_atg.iloc[i, 1])])
negative = []
for i in negative_seqs:
negative.append(sum([pwm.loc[j].iloc[k] for k, j in enumerate(i)]))
По критерию Манна-Уинти распредления весов последовательностей по PWM для позитивного и негативного контролей различны (p<0.001).
mannwhitneyu(positive, negative)
MannwhitneyuResult(statistic=3008.0, pvalue=2.331396194321208e-10)
Ниже представлена визуализация весов, взависимости от типа контроля. Действительно видно, что распределение положительного контроля находится заметно выше, чем отрицательного. (картинка итерактивна)
# BOXPLOTS
fig = go.Figure()
fig.add_trace(go.Box(y = positive, name = 'positive'))
fig.add_trace(go.Box(y = negative, name = 'negative'))
fig.update_layout(
width = 800, height=500,
showlegend=False,
margin = dict(l = 20, t = 20, r = 20, b = 20),
font=dict(
family="Futura",
size=14
)
)
fig.show()
Далее была построена матрица информационного содержания (IC) по обучающим последовательностям.
# IC
ic_dict = {'A': [], 'G': [], 'T': [], 'C': []}
transposed = tp(list(map(lambda x: [i for i in x.__str__()], train)))
df = pd.DataFrame(transposed)
for i in df.index:
values = df.iloc[i, :].value_counts()
for k, v in proba.items():
try:
ic_dict[k].append(round((values.loc[k]/N)*log2(values.loc[k]/(N*v)), 3))
except KeyError:
ic_dict[k].append(0)
ic = tp(pd.DataFrame(ic_dict))
ic
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.149 | -0.115 | -0.063 | -0.134 | -0.091 | 0.003 | -0.134 | 1.747 | 0.000 | 0.000 | -0.134 | -0.063 | -0.134 |
| G | 0.520 | 0.586 | 0.171 | -0.064 | 0.868 | -0.036 | 0.077 | 0.000 | 0.000 | 2.308 | 0.520 | 0.035 | 0.394 |
| T | -0.158 | -0.149 | -0.157 | -0.091 | -0.149 | -0.149 | -0.158 | 0.000 | 1.747 | 0.000 | -0.063 | -0.091 | -0.149 |
| C | 0.171 | -0.036 | 0.223 | 0.520 | -0.101 | 0.335 | 0.586 | 0.000 | 0.000 | 0.000 | -0.087 | 0.171 | 0.122 |
print(f'Суммарный сигнал равен {round(ic.sum().sum(), 3)}')
Суммарный сигнал равен 8.449
С помощью сервиса WebLogo была получена следующая визуализация:

По картинке можно сделать вывод, что последовательность Козак богата G и C нуклеотидами.
!jupyter nbconvert --to html pwm.ipynb
[NbConvertApp] Converting notebook pwm.ipynb to html [NbConvertApp] Writing 616735 bytes to pwm.html