Enzymes Classification (EC)
Задание 1. Сбор информации о ферменте PPOX_HUMAN
| ЕС код | 1.3.3.4 |
| PDB ID | 1SEZ |
| Название | Protoporphyrinogen oxidase (Оксидаза протопорфириногена) |
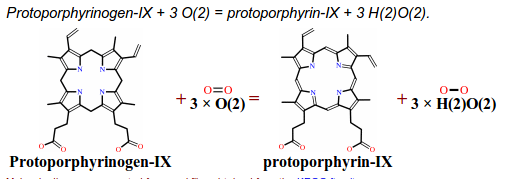
| Катализируемая реакция | протопорфироген-IX + 3хO2 = протопорфирин-IX + 3хH2O2 |
| ЕС 1 | Оксидоредуктазы |
| ЕС 1.3 | Воздействуют на CH-CH группу доноров |
| EC 1.3.3 | С кислородом в качестве акцептора |
Схема катализируемой реакции (источник):
Задание 2. Поиск других белков человека со сходными функциями (в Swiss-Prot)
С помощью SRS выясним, сколько белков человека являются ферментами с тем же классом по EC, сколько — с тем же классом и подклассом, сколько имеют общие с PPOX_HUMAN три уровня классификации и сколько — все четыре.
| ЕС код | Число записей в SW |
| 1.*.*.* | 549 |
| 1.3.*.* | 49 |
| 1.3.3.* | 5 |
| 1.3.3.4 | 1 |
Задание 3. Насколько сохраняется функция PPOX_HUMAN у белков со сходными последовательностями
С помощью SRS получим список всех белков мыши (Mus musculus) из Swiss-Prot, у которых класс (т.е., первое число) классификации EC совпадает с таковым для PPOX_HUMAN (EC 1). Всего нашлось 549 таких белков(таблица ЕС кодами, файл с fasta-последоватлеьностями). Среди них 46 белков имеют такой же подкласс (ЕС 1.3), 5 - такой же подподкласс (ЕС 1.3.3), и 1 такую же классификацию (1.3.3.4).
Пользуясь программой blastp, найдем среди этих белков сходные по последовательности с PPOX_HUMAN. Порог по e-value поставим с запасом (1):
makeblastdb -in seqs.fasta -dbtype prot -out db blastp -query 1SEZ.fasta -db db -out al.txt -evalue 1
Файл с выравниванием. Всего нашлось 17 белков, и, я думаю, очень достоверным гомологом можно считать только PPOX_MOUSE, т.к. e-value для него достаточно маленький и, кроме того, он выполняет такую же функцию. Однако, есть еще девять оксидаз, e-value для которых не превышает 3e-04. Вероятно, они менее родственны, но имеют сходное происхождение. Создайте таблицу, которая бы отражала, насколько сохраняется функция (т.е. подкласс, подподкласс и порядковый номер EC) в зависимости от позиции находки в выдаче blastp. Таблицу можно сформировать в HTML-формате или сделать в Excel и прикрепить к отчёту.
Таблица, отражающая, насколько "сохраняется код EC" в зависимости от позиции в выдаче blastp:
| Находка | E-value | EC |
| PPOX_MOUSE | 6e-20 | 1.3.3.4 |
| KDM1A_MOUSE | 2e-08 | 1.-.-.- |
| AOFA_MOUSE | 6e-07 | 1.4.3.4 |
| AOFB_MOUSE | 6e-06 | 1.4.3.4 |
| RETST_MOUSE | 2e-05 | 1.3.99.23 |
| PCYXL_MOUSE | 3e-05 | 1.8.3.- |
| PAOX_MOUSE | 8e-05 | 1.5.3.13 |
| FMO3_MOUSE | 3e-04 | 1.14.13.8, 1.14.13.148 |
| FMO4_MOUSE | 3e-04 | 1.14.13.8 |
| FMO1_MOUSE | 3e-04 | 1.14.13.8 |
| PCYOX_MOUSE | 0.001 | 1.8.3.5 |
| FMO5_MOUSE | 0.003 | 1.14.13.8 |
| FMO2_MOUSE | 0.004 | 1.14.13.8 |
| AIFM3_MOUSE | 0.023 | 1.-.-.- |
| PYRD2_MOUSE | 0.037 | 1.-.-.- |
| KMO_MOUSE | 0.29 | 1.14.13.9 |
| KDM1B_MOUSE | 0.46 | 1.-.-.- |
Как видно, находка с наименьшими e-value (PPOX_MOUSE) совпадает с ферментом PPOX_HUMAN по всему коду. При этом эти ферменты оба являются оксидазами протопорфириногена. Другие находки различаются уже в подклассе. Интересно, что большинство из них относятся в 1.14 подклассу - воздействующие на парные доноры с расщеплением молекулярного кислорода.
Наверх