Семестр VII. Рентгеноструктурный анализ (часть 1)
Электронная плотность. Рассеяние электрона
Выбор структуры
Для дальнейшей работы структура белка была выбрана по нескольким критериям:REMARK 2 RESOLUTION. 2.40 ANGSTROMS.
В итоге была выбрана симпатичная глицеральдегид-3-фосфатдегидрогеназа кролика (GAPDH, PDB ID - 1J0X). Это гомотетрамер с большим количеством гомологов во всех организмах, где есть гликолиз.
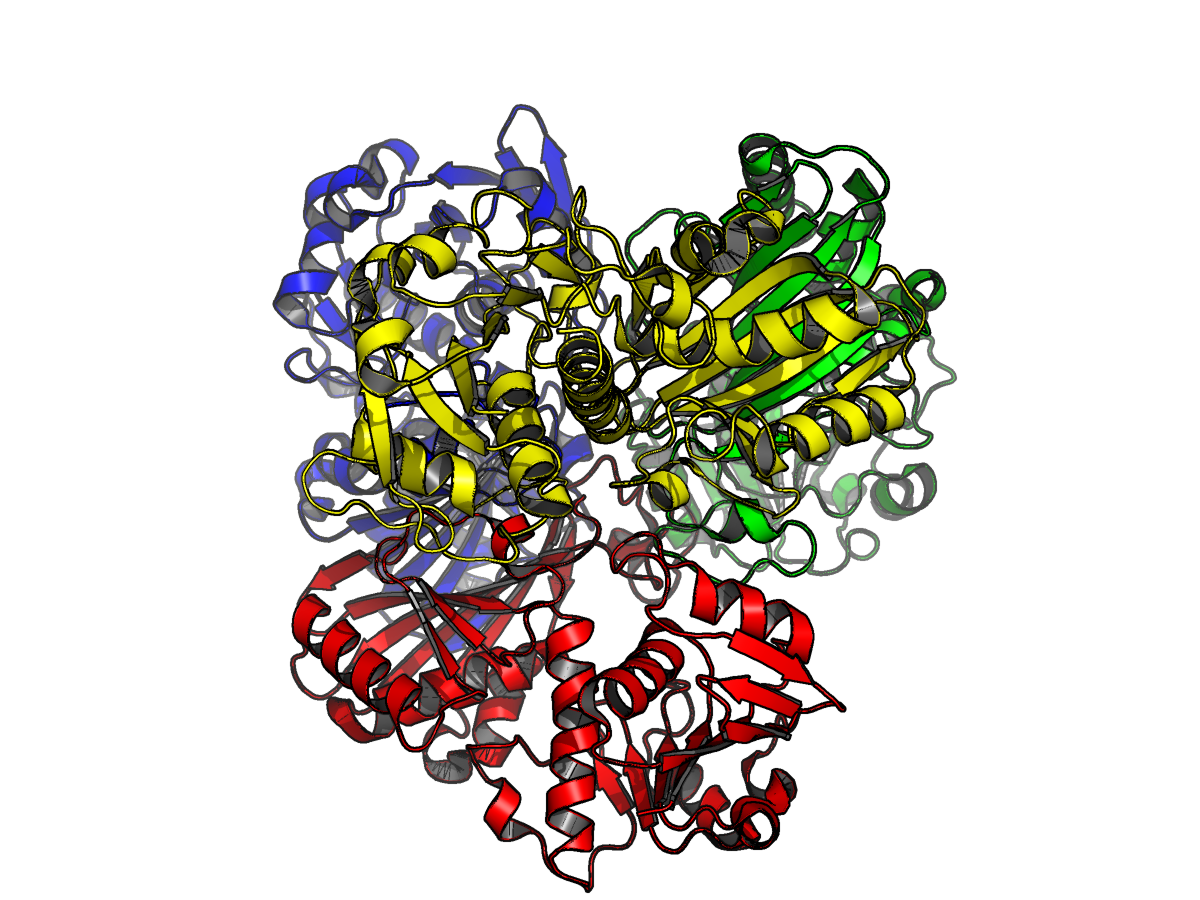
Изображение гомотетрамера ГАФД.
Модель и файл структурных факторов
Для выбранной структуры были скачаны модель 1J0X структуры, файл mmCIF и файл структурных факторов (.cif - crystallographic information file). В файле структурных факторов описано, какая информация в нем содержится:_refln.crystal_id _refln.wavelength_id _refln.scale_group_code _refln.index_h _refln.index_k _refln.index_l _refln.status _refln.F_meas_au _refln.F_meas_sigma_au 1 1 1 6 0 0 o 220.8520 5.72873Каждая строчка _refln. поясняет столбец данных. Каждый структурный фактор расположен в отдельной строке, представляющей собой последовательность чисел, которые заполняют поля mmCIF формата. Определим число атомов в модели по файлам .pdb и .cif:
grep -Eh ^ATOM\|^HETATM 1J0X.pdb | wc -l # 10605 = number of atoms
grep -Eh '[0-9]{3}' 1j0x-sf.cif | wc -l # 56197 = number of reflections
Таким образом, число записей в файле структурных факторов не соответствует количеству атомов в PDB-структуре.
Построение изображений электронной плотности
Файл с электронной плотностью можно получить на сайте EDS. На странице с информацией о записи PDB в разделе Download => "Maps" => заказать карту в формате по умолчанию (O и 2mFo-DFc).Чтобы оценить качество модели пространственной структуры, представленной в PDB файле, были построены изображения электронной плотности для 3 уровней электронной плотности (ЭП) вокруг остова цепи О структуры фермента.
 Level 1 |
 Level 2 |
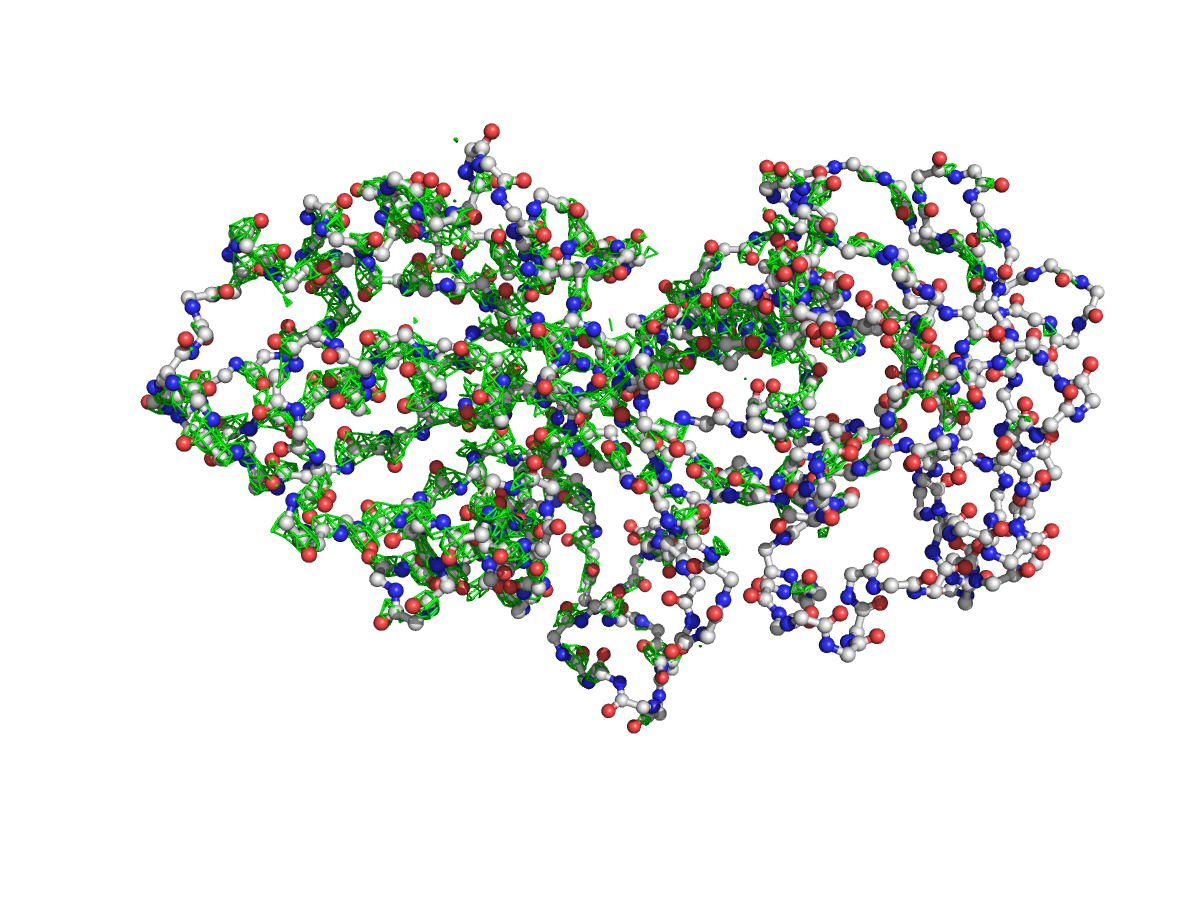 Level 3 |
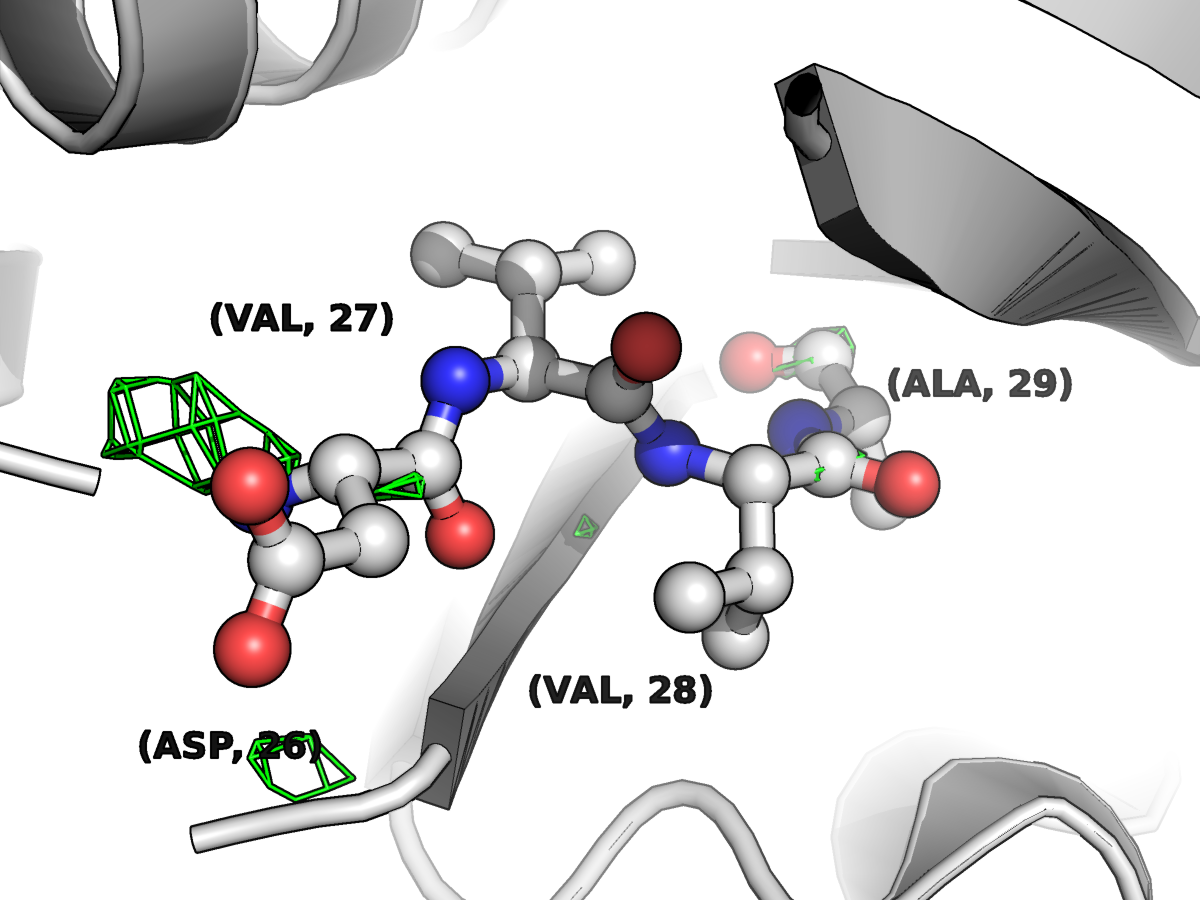
Также были построены изображения электронной плотности для 3 уровней электронной плотности (ЭП) вокруг четырех (26-29) аминокислотных остатков. Из этого представления видно, что (если словами описывать качество модели), то она не очень соотносится с распределением электронной плотности, полученной методом РСА, так кк видны непонятные ошметки паутинки ЭП на "пустых" местах модели, которые никак не могут принадлежать соседним асимметрическим единицам ячейки.
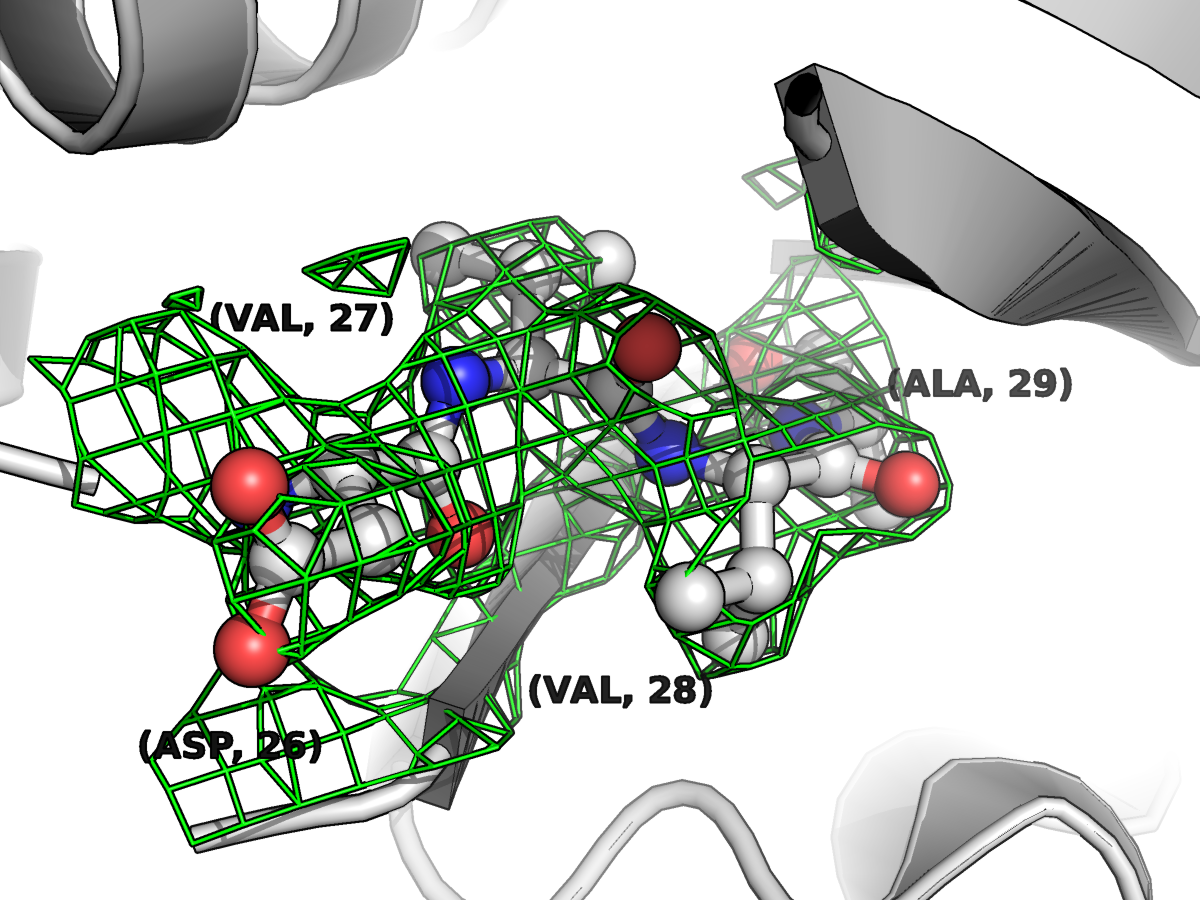
|
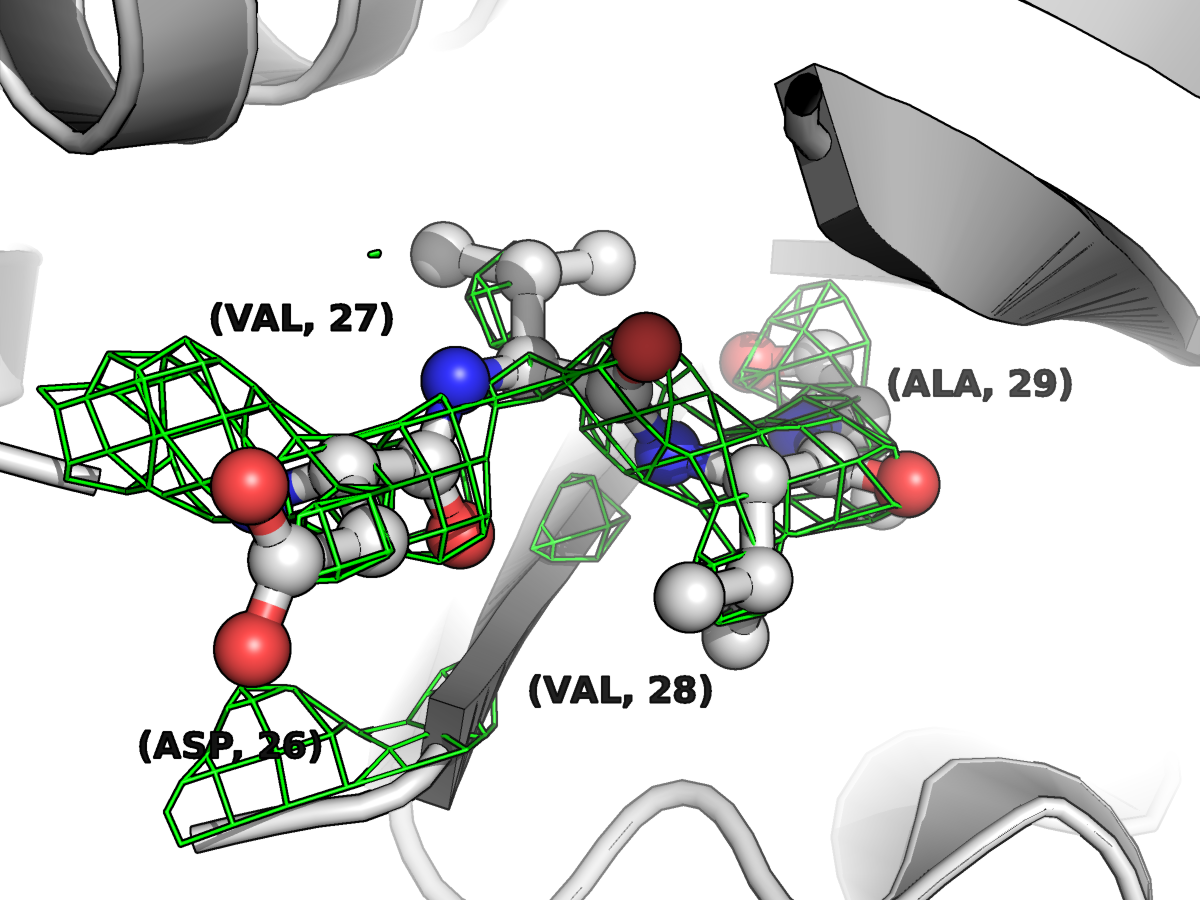
|
|
|||
| Level 1 | Level 2 | Level 3 |
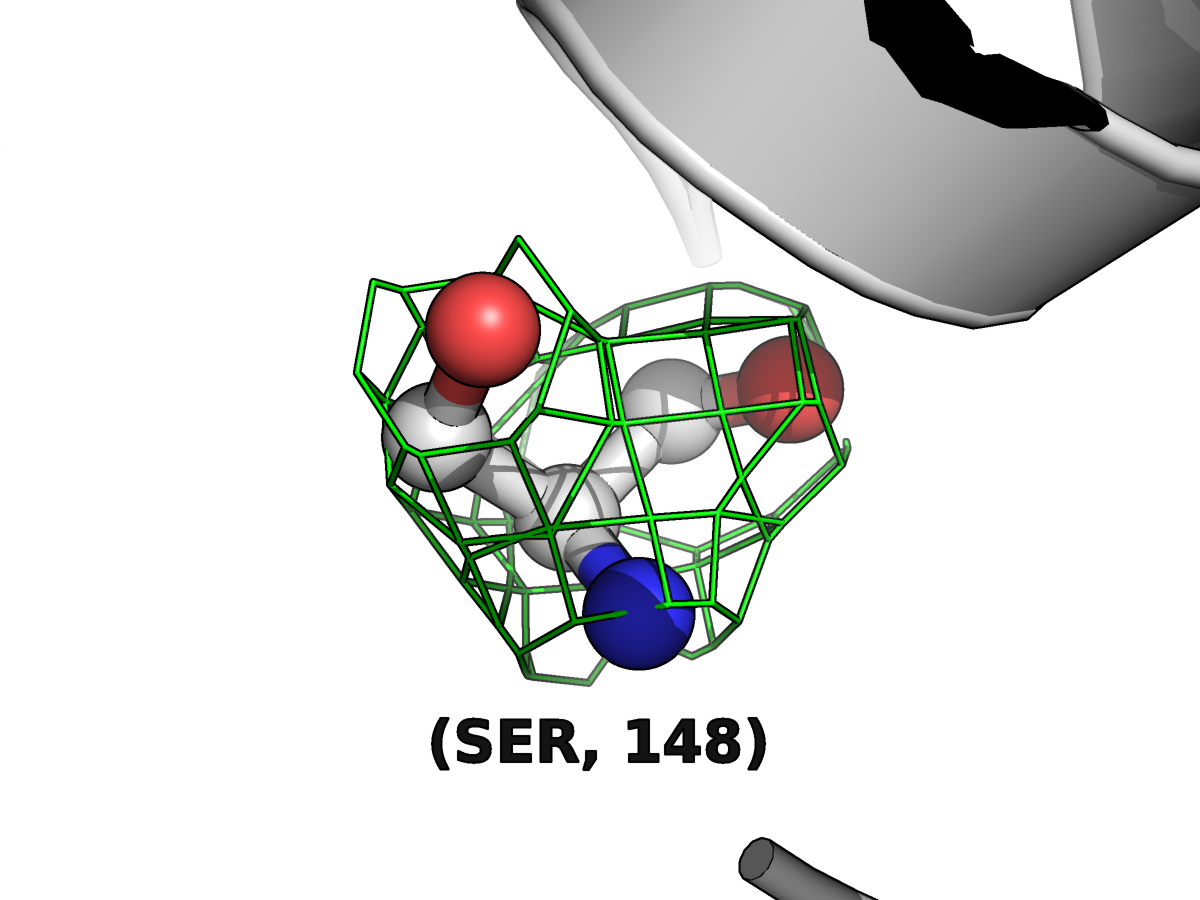
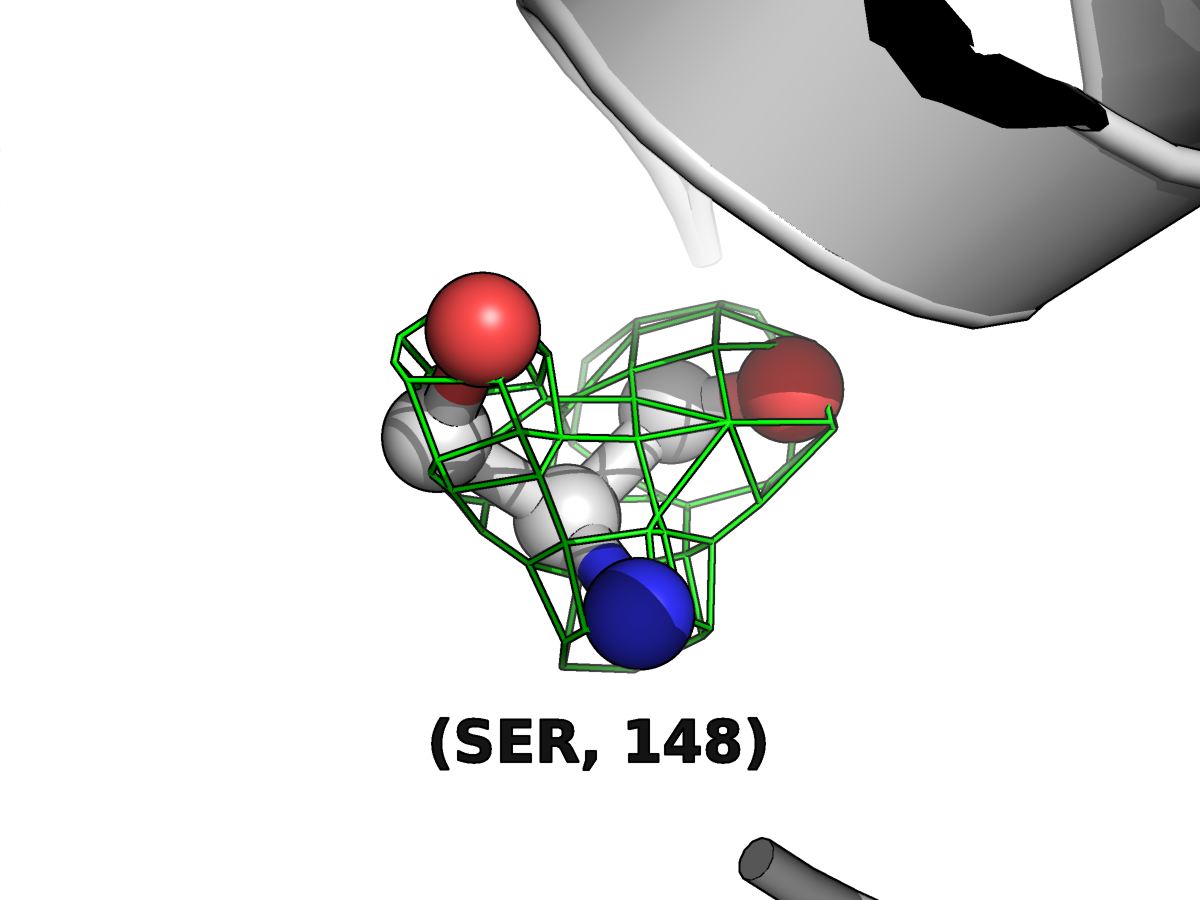
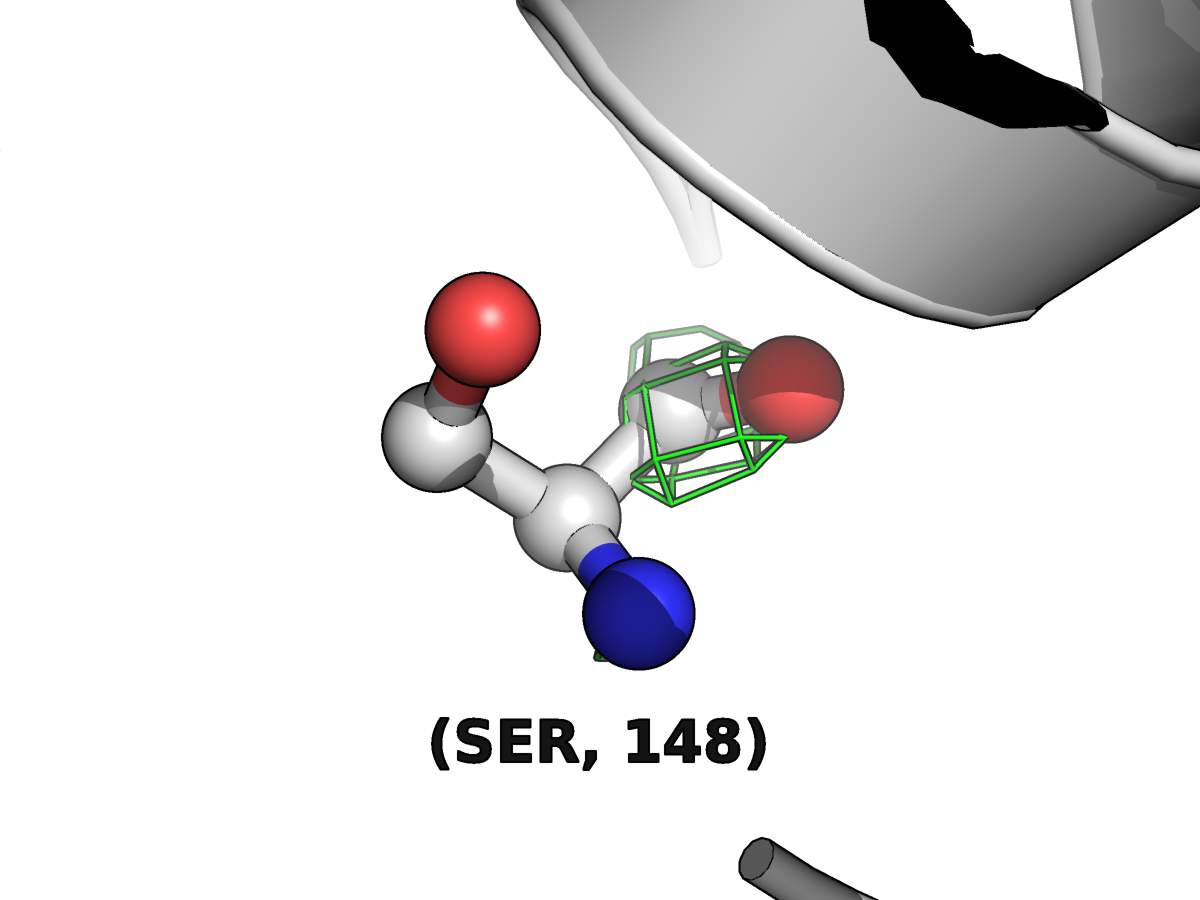
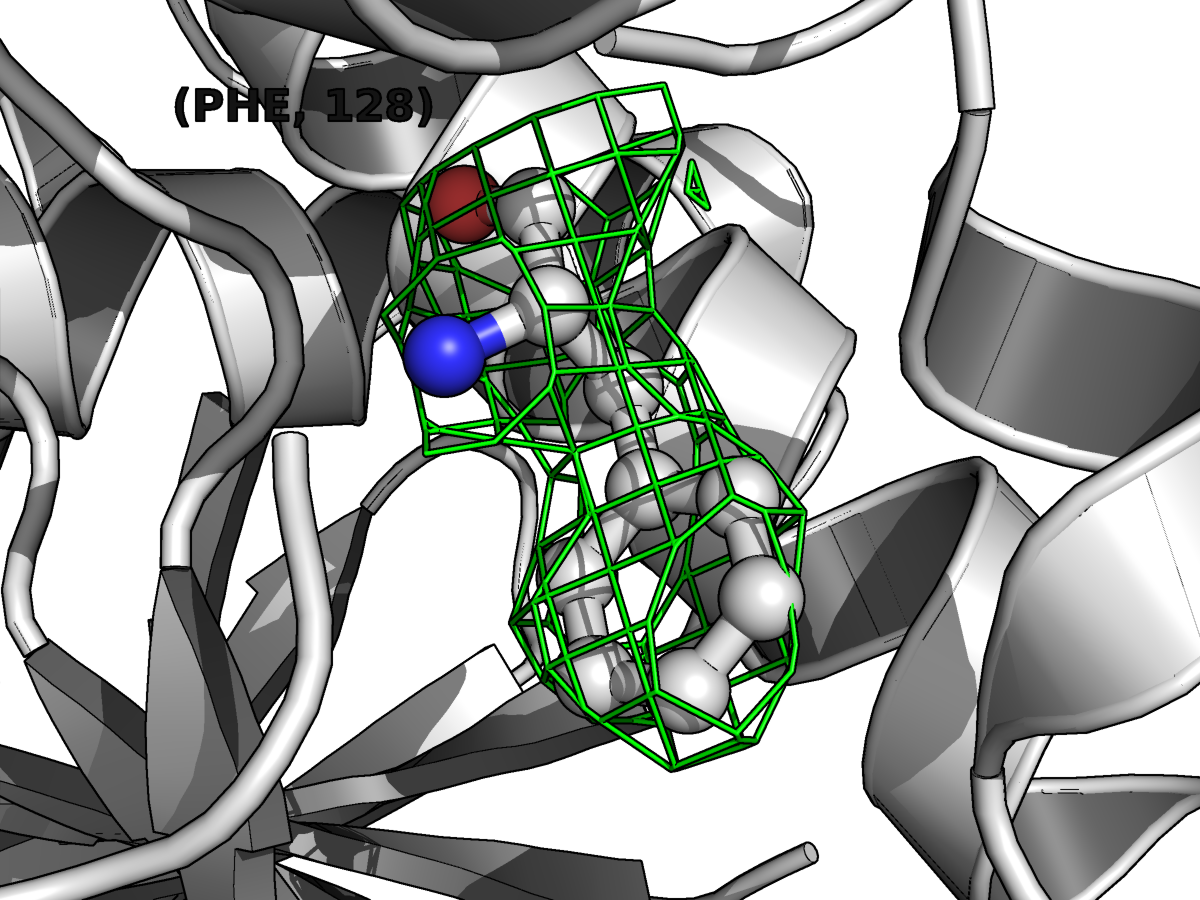
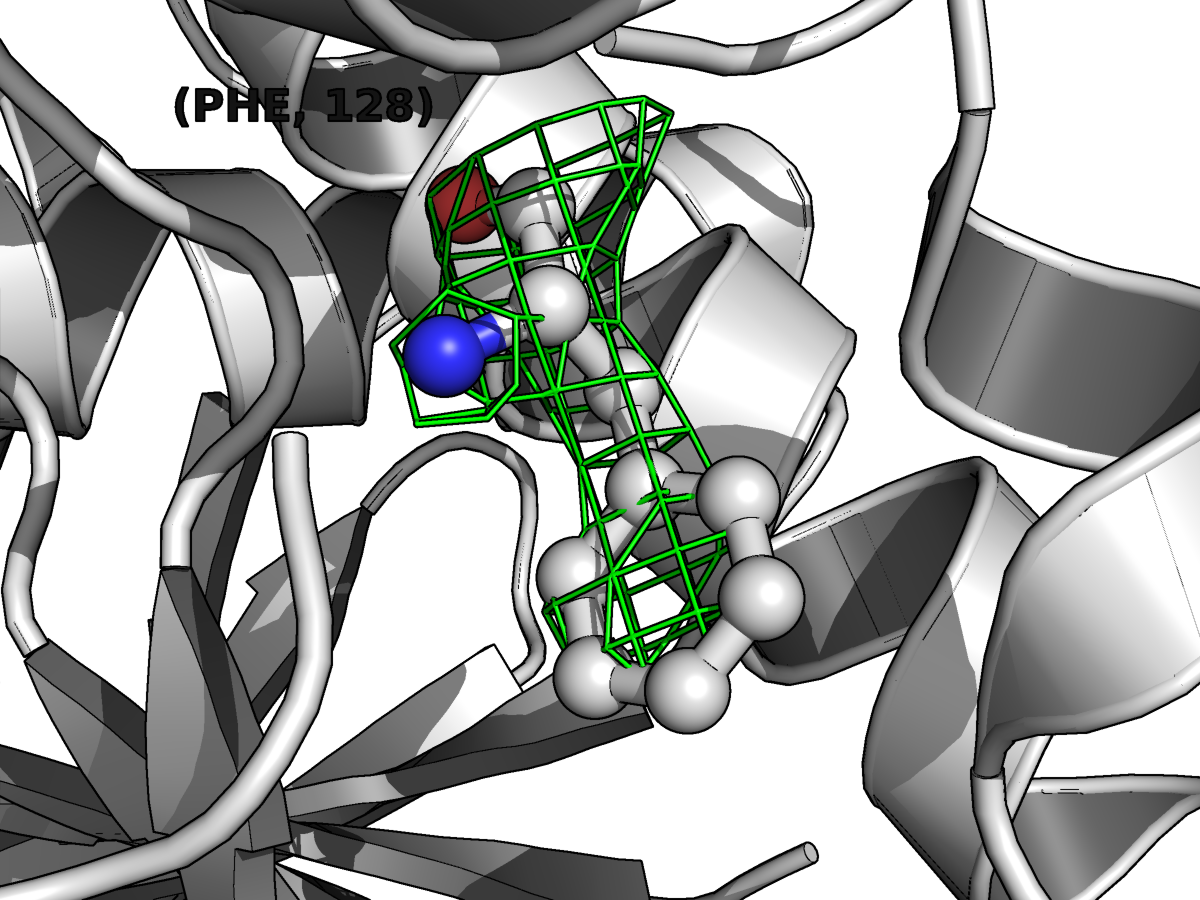
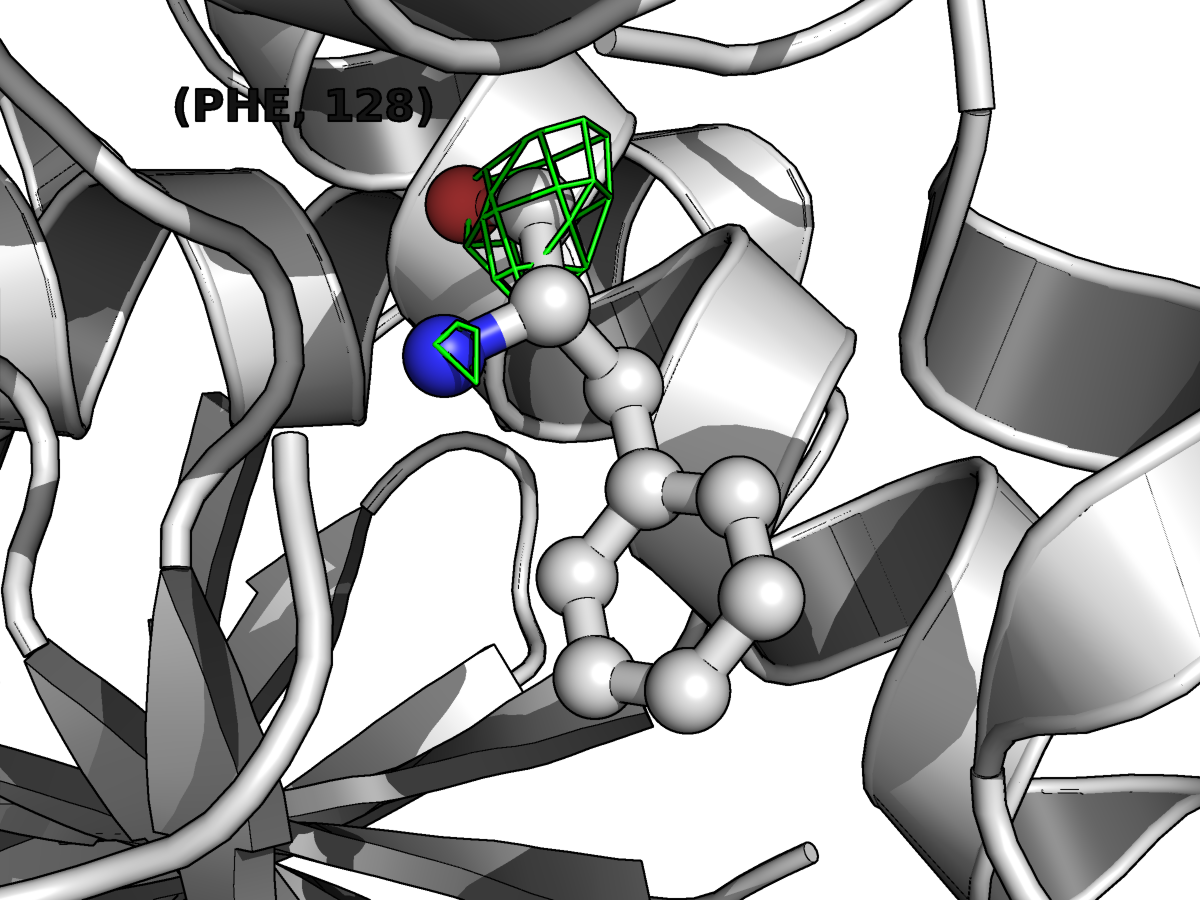
Так же приведу картинку остатков серина-148 b фенилаланина-128. Здесь можно отметить, что плотность более менее соответствует положению атомов, а при рассмотрении третьего уровня наибольшая плотнось как раз на двойной связи С=О и атоме азота N, что, вообще, логично. Хоть карта электронной плотности не очень красиво и точно повторяет модель, видимо, этого кчества достаточно для создания реконструкции структуры.
|
|
|
|||
| Level 1 | Level 2 | Level 3 | |||
|
|
|
|||
| Level 1 | Level 2 | Level 3 |
Статья о расшифровке выбранной структуры
В аннотации PDB-файла модели 1J0X указана публикация по данной структуре.JRNL AUTH S.W.COWAN-JACOB,M.KAUFMANN,A.N.ANSELMO,W.STARK, JRNL AUTH 2 M.G.GRUTTER JRNL TITL STRUCTURE OF RABBIT-MUSCLE JRNL TITL 2 GLYCERALDEHYDE-3-PHOSPHATE DEHYDROGENASE. JRNL REF ACTA CRYSTALLOGR.,SECT.D V. 59 2218 2003 JRNL REFN ISSN 0907-4449 JRNL PMID 14646080 JRNL DOI 10.1107/S0907444903020493Оналегко находится в PubMed (ссылка)