Семестр VII. Рентгеноструктурный анализ (часть 4)
|
Интерпретация особенностей РСА
Поиск ошибок в PDB-файлах
- Гликоген фосфорилаза кролика (гомотетрамер)
- 1991 г. (статья)
- Разрешение – 2.9 А
- Нарушение планарности боковых цепей некоторых аминокислот.
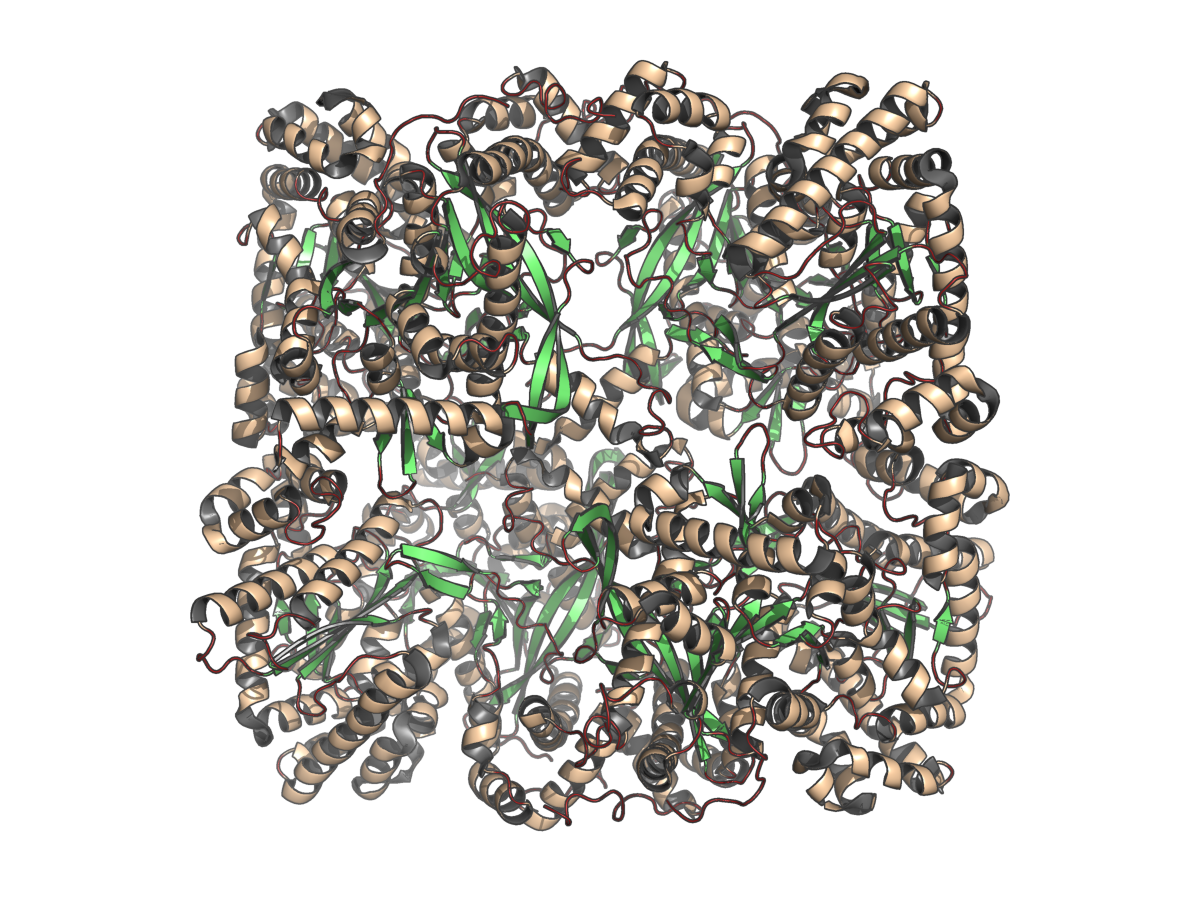 |
| Рис.1. Общий вид структуры 7GPB |
Нарушение планарности боковых цепей, в частности, наблюдается у некоторых остатков триптофанов структуры 7GPB. В норме у триптофана ароматические кольца расположены в одной плоскости, так как ЭП этих колец едина. Это позволяет ему вступать в стекинг-взаимодействия с другими подобными молекулами. На изображениях ниже показаны "нормлаьный" остаток триптофана 825 цепи D (TRP825D) и два остатка с нарушением планарности - TRP67D и TRP321B.
B-фактор для атомов бензольного кольца индола у TRP67D значимо выше, чем для TRP825D. В WHAT IF Check report базы PDBREPORT
некоторые аминокислотные остатки указаны с ошибкой о непланарности их боковых цепей (Side chain planarity problems), в которой на первой позиции и обнаруживается как раз TRP67D. В комментарияхк ошибке указано, что приведены остатки, для которых планарная группа в 4 раза отклоняется по планарности от ожидаемого значения.
grep 'TRP D 67 ' 7GPB.pdb
#Записьв PDB-файле про атомы бензольного кольца индола 67ого остатка
ATOM 20556 CG TRP D 67 77.546 64.187 24.889 1.00 33.35 C
ATOM 20557 CD1 TRP D 67 76.235 63.844 25.011 1.00 30.99 C
ATOM 20558 CD2 TRP D 67 78.292 63.928 26.025 1.00 39.56 C
ATOM 20559 NE1 TRP D 67 76.165 63.403 26.237 1.00 39.84 N
ATOM 20560 CE2 TRP D 67 77.355 63.428 26.895 1.00 42.57 C
ATOM 20561 CE3 TRP D 67 79.192 63.047 25.449 1.00 47.94 C
ATOM 20562 CZ2 TRP D 67 77.290 62.090 27.217 1.00 52.89 C
ATOM 20563 CZ3 TRP D 67 79.136 61.693 25.749 1.00 53.44 C
ATOM 20564 CH2 TRP D 67 78.194 61.211 26.633 1.00 54.15 C
#Записьв PDB-файле про атомы бензольного кольца индола 825ого остатка
ATOM 26672 CG TRP D 825 56.734 64.699 24.279 1.00 17.74 C
ATOM 26673 CD1 TRP D 825 56.251 63.645 25.047 1.00 14.42 C
ATOM 26674 CD2 TRP D 825 57.515 64.248 23.254 1.00 14.51 C
ATOM 26675 NE1 TRP D 825 56.716 62.555 24.498 1.00 3.55 N
ATOM 26676 CE2 TRP D 825 57.465 62.874 23.431 1.00 13.08 C
ATOM 26677 CE3 TRP D 825 58.290 64.790 22.257 1.00 17.33 C
ATOM 26678 CZ2 TRP D 825 58.183 62.026 22.602 1.00 18.83 C
ATOM 26679 CZ3 TRP D 825 59.018 63.935 21.423 1.00 21.80 C
ATOM 26680 CH2 TRP D 825 58.966 62.557 21.592 1.00 18.03 C
#WHAT IF Check report
Error: Side chain planarity problems
2527 TRP ( 67-) D 36.29
58 TRP ( 67-) A 12.99
1170 TRP ( 361-) B 10.86
1129 ASP ( 320-) B 8.53
1704 TRP ( 67-) C 7.28
1606 TRP ( 797-) B 7.07
1300 TRP ( 491-) B 7.06
2820 TRP ( 365-) D 6.17
1196 TRP ( 387-) B 5.98
3252 TRP ( 797-) D 5.96
180 TRP ( 189-) A 5.80
268 ARG ( 277-) A 5.52
1997 TRP ( 365-) C 5.35
2019 TRP ( 387-) C 5.26
1881 TRP ( 244-) C 5.15
1922 GLU ( 290-) C 5.00
1826 TRP ( 189-) C 4.98
881 TRP ( 67-) B 4.96
526 GLU ( 540-) A 4.89
1632 GLU ( 823-) B 4.68
3156 GLU ( 701-) D 4.64
1099 GLU ( 290-) B 4.61
996 TRP ( 182-) B 4.44
88 ASN ( 97-) A 4.37
538 GLU ( 552-) A 4.36
373 TRP ( 387-) A 4.26
477 TRP ( 491-) A 4.05
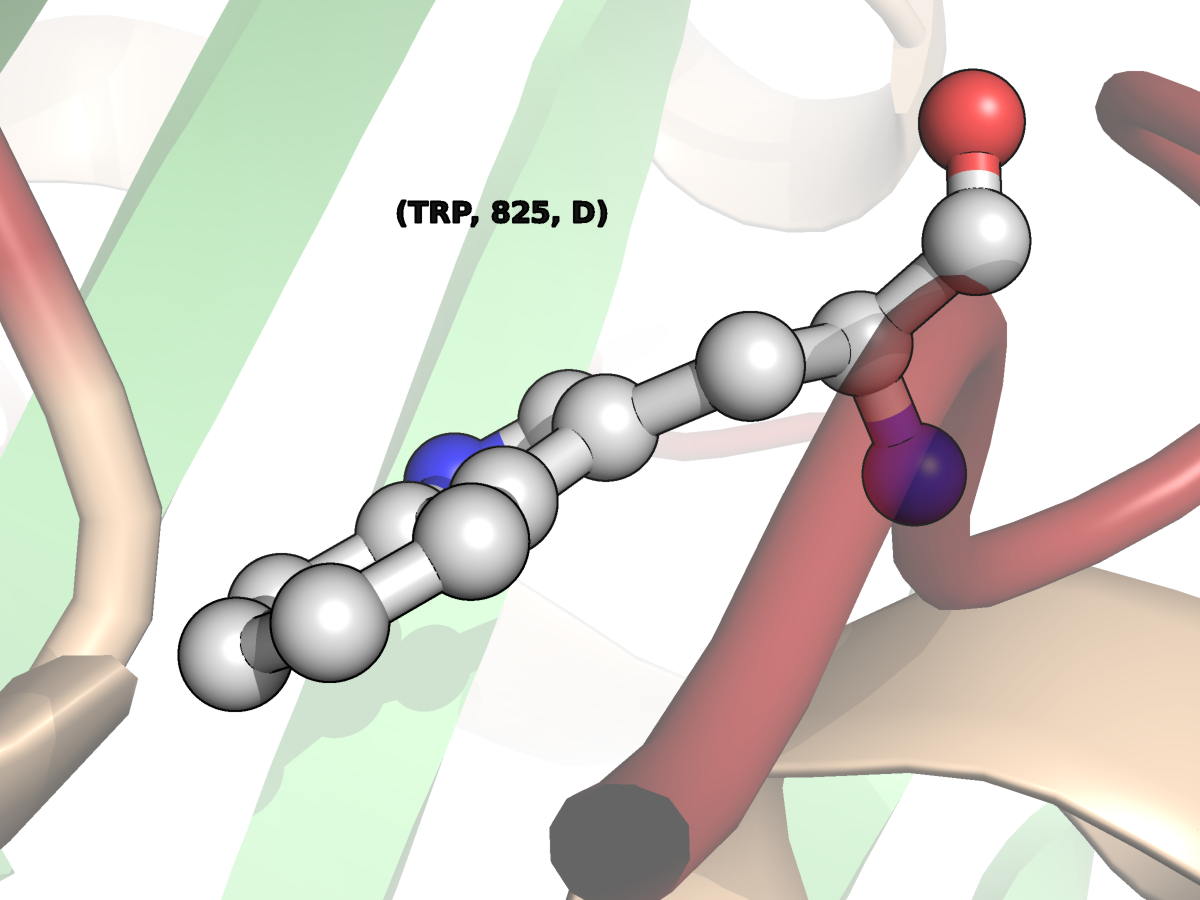 |
| Рис.2. Триптофан 825 цепи D структуры 7GPB ('нормальный') |
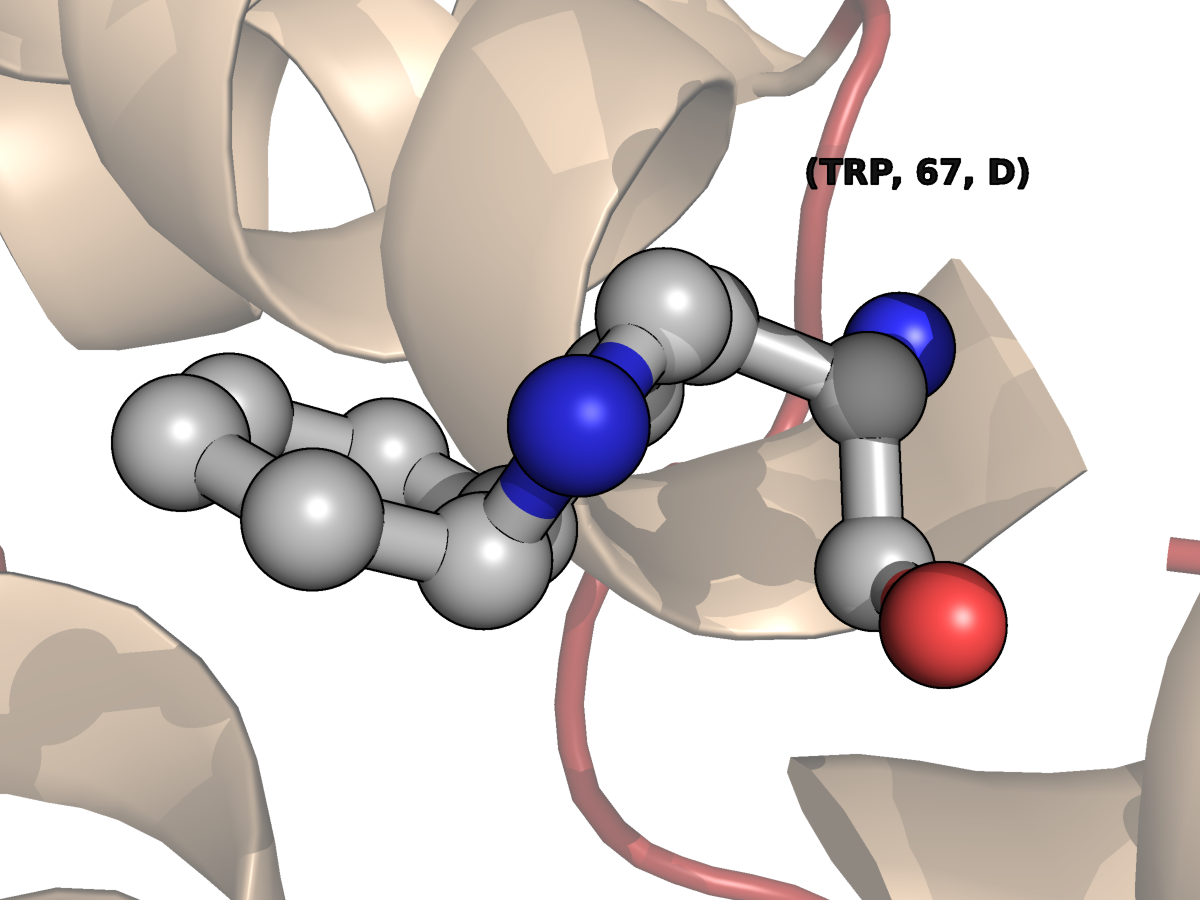 |
| Рис.3. Триптофан 67 цепи D структуры 7GPB ('дефектный') |
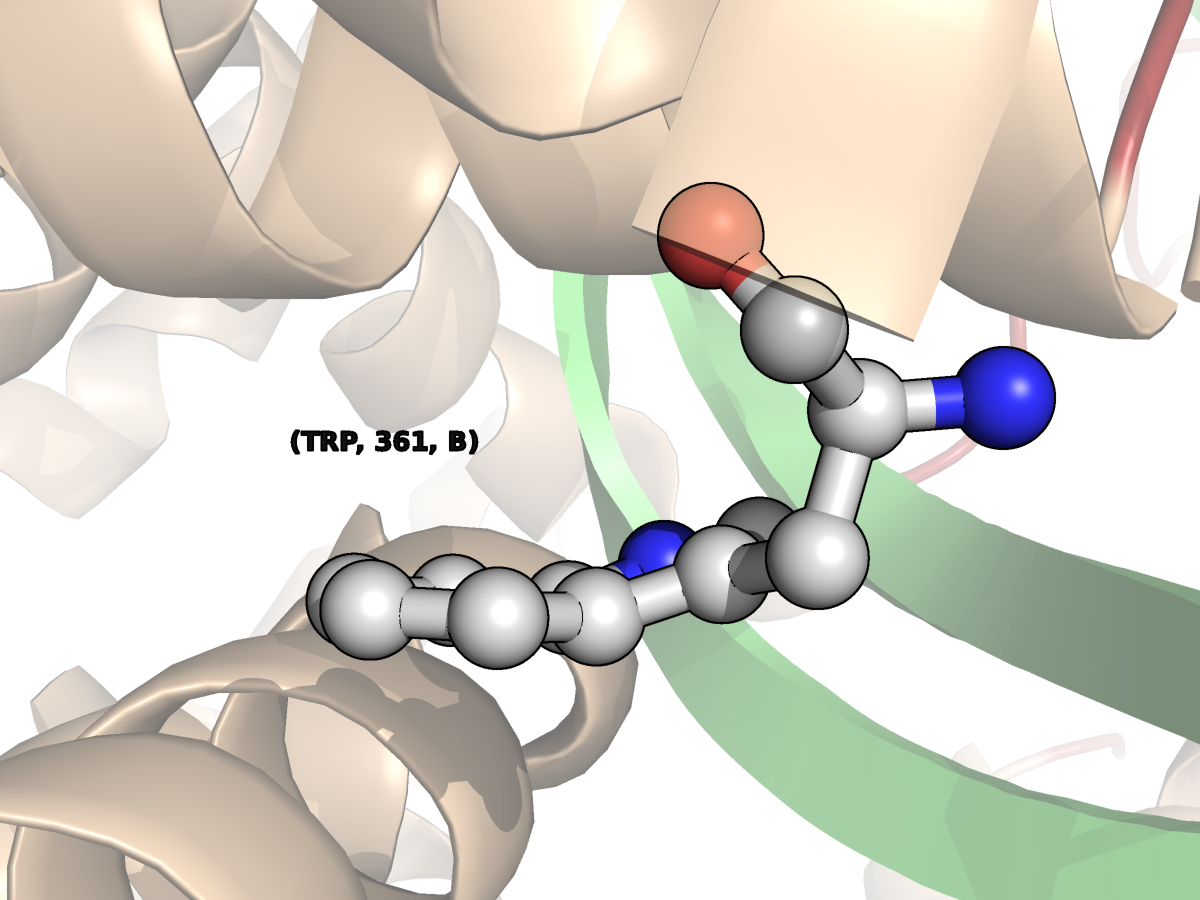 |
| Рис.4. Триптофан 361 цепи B структуры 7GPB ('дефектный') |
- Комплекс ДНК с доменами POU и HMG транскрипционных факторов
- 2003 г. (статья)
- Разрешение – 2.6 А
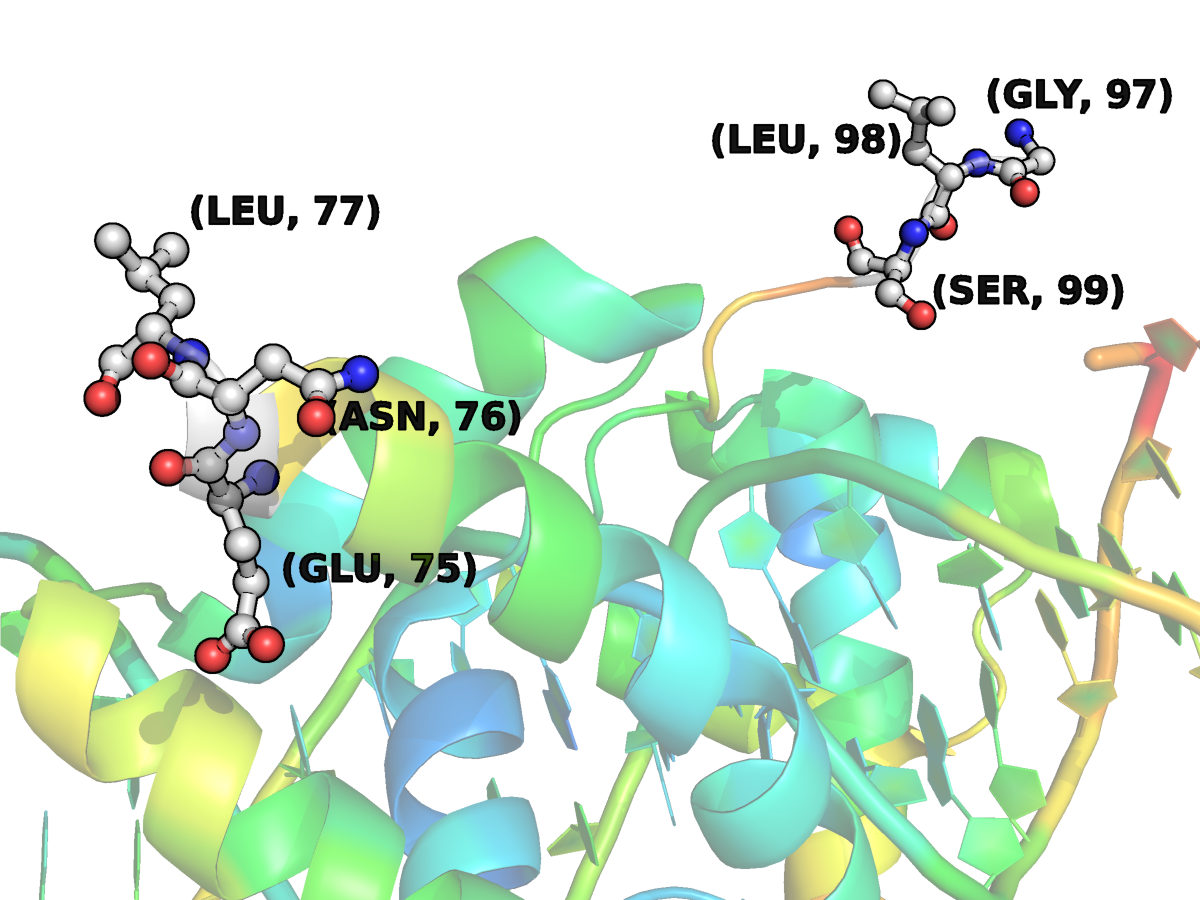
- В цепи C (домен POU) отсутствуют аминокислоты с 78 по 96.
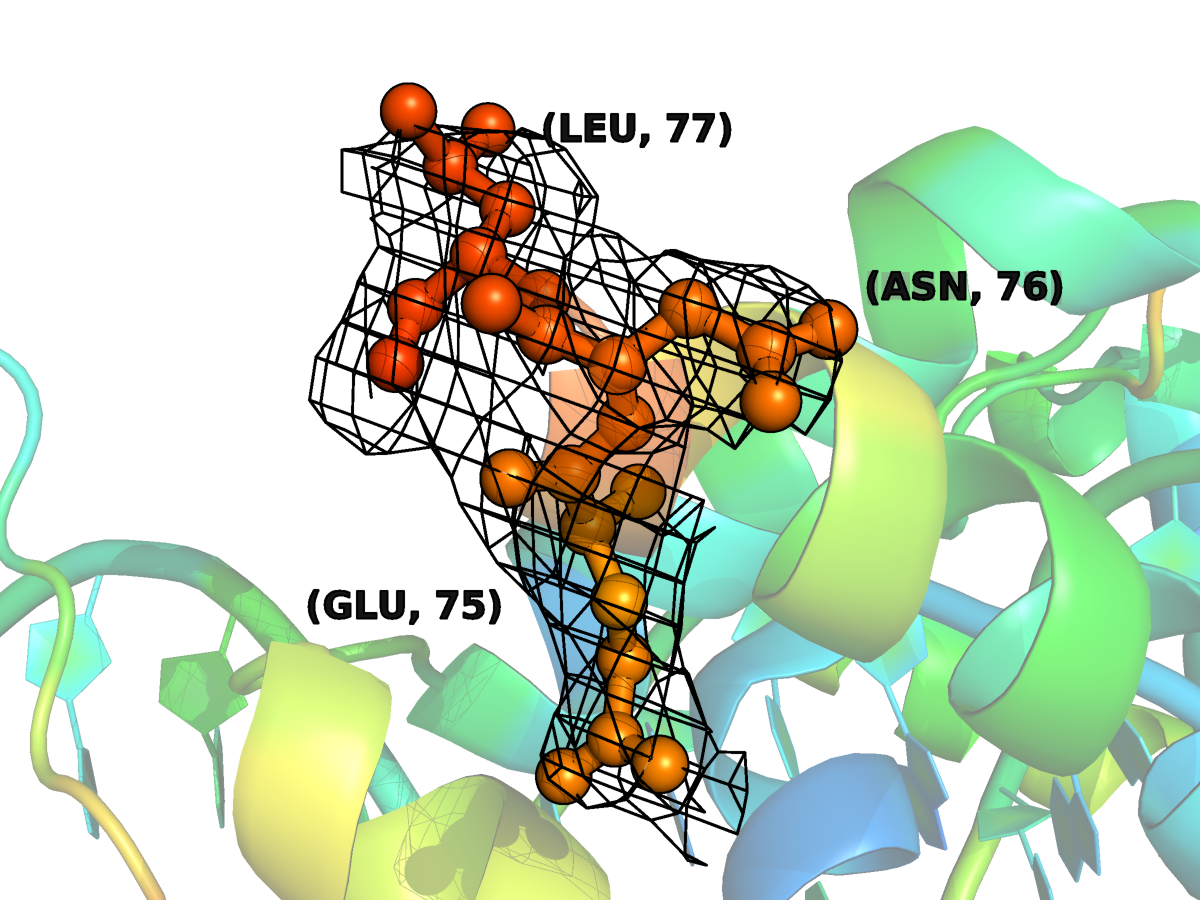
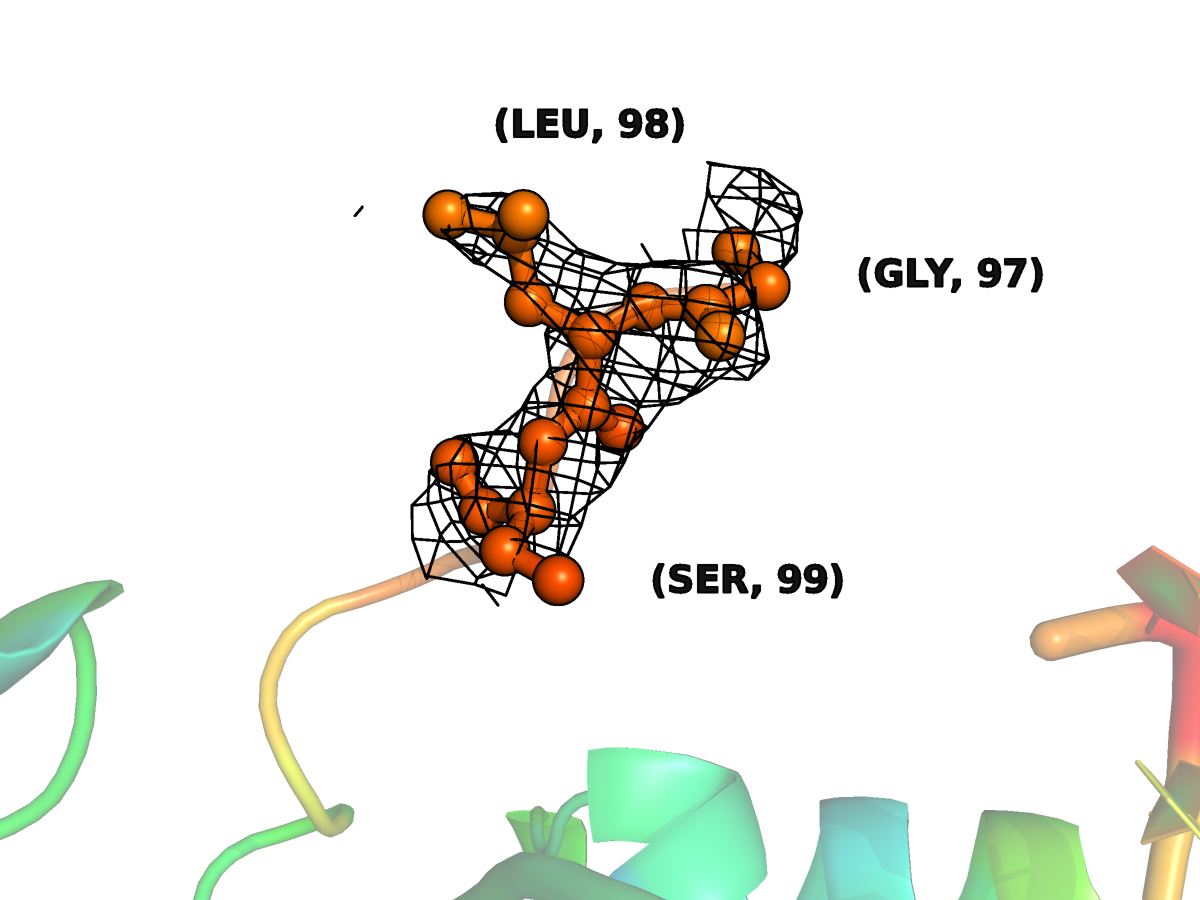
Значения температурных факторов для остатков 75 - 77 и 97 – 99 (по три с каждого края разрыва) имеют высокие значения (от 77.92 до 91.29 - на рис.5,6.1-6.3 в красную сторону радуги), то есть их расположение в различных ячейках было настолько различным, что привести состоятельную модель нельзя.
#Записьв PDB-файле про атомы остатков 75-77 и 97-99
ATOM 1558 N GLU C 75 8.467 44.875 24.155 1.00 77.92 N
ATOM 1559 CA GLU C 75 7.448 44.816 23.112 1.00 80.17 C
ATOM 1560 C GLU C 75 7.799 45.790 22.004 1.00 82.99 C
ATOM 1561 O GLU C 75 7.405 45.589 20.848 1.00 83.49 O
ATOM 1562 CB GLU C 75 6.065 45.155 23.651 1.00 78.81 C
ATOM 1563 CG GLU C 75 5.523 44.120 24.596 1.00 81.36 C
ATOM 1564 CD GLU C 75 4.011 44.024 24.555 1.00 81.89 C
ATOM 1565 OE1 GLU C 75 3.339 45.052 24.807 1.00 81.50 O
ATOM 1566 OE2 GLU C 75 3.499 42.914 24.268 1.00 82.12 O
ATOM 1567 N ASN C 76 8.538 46.844 22.356 1.00 84.44 N
ATOM 1568 CA ASN C 76 8.931 47.847 21.374 1.00 86.12 C
ATOM 1569 C ASN C 76 9.724 47.119 20.312 1.00 87.51 C
ATOM 1570 O ASN C 76 9.491 47.289 19.110 1.00 88.66 O
ATOM 1571 CB ASN C 76 9.804 48.946 22.000 1.00 84.63 C
ATOM 1572 CG ASN C 76 9.067 49.759 23.058 1.00 84.99 C
ATOM 1573 OD1 ASN C 76 7.900 50.126 22.886 1.00 83.80 O
ATOM 1574 ND2 ASN C 76 9.757 50.056 24.159 1.00 84.92 N
ATOM 1575 N LEU C 77 10.649 46.288 20.773 1.00 88.12 N
ATOM 1576 CA LEU C 77 11.499 45.516 19.882 1.00 89.76 C
ATOM 1577 C LEU C 77 10.865 44.173 19.486 1.00 90.46 C
ATOM 1578 O LEU C 77 9.655 44.076 19.239 1.00 91.29 O
ATOM 1579 CB LEU C 77 12.854 45.286 20.555 1.00 89.30 C
ATOM 1580 CG LEU C 77 13.849 44.408 19.797 1.00 90.05 C
ATOM 1581 CD1 LEU C 77 14.162 45.032 18.432 1.00 89.23 C
ATOM 1582 CD2 LEU C 77 15.112 44.249 20.643 1.00 89.10 C
ATOM 1583 N GLY C 97 30.324 49.409 51.555 1.00 84.31 N
ATOM 1584 CA GLY C 97 29.047 50.093 51.729 1.00 85.51 C
ATOM 1585 C GLY C 97 27.987 49.836 50.654 1.00 85.39 C
ATOM 1586 O GLY C 97 26.863 50.354 50.721 1.00 83.11 O
ATOM 1587 N LEU C 98 28.341 49.038 49.653 1.00 85.66 N
ATOM 1588 CA LEU C 98 27.415 48.720 48.575 1.00 86.31 C
ATOM 1589 C LEU C 98 26.543 47.531 48.961 1.00 87.06 C
ATOM 1590 O LEU C 98 26.782 46.884 49.981 1.00 88.21 O
ATOM 1591 CB LEU C 98 28.179 48.359 47.296 1.00 85.46 C
ATOM 1592 CG LEU C 98 29.058 49.376 46.576 1.00 83.94 C
ATOM 1593 CD1 LEU C 98 29.560 48.742 45.284 1.00 83.19 C
ATOM 1594 CD2 LEU C 98 28.270 50.641 46.286 1.00 83.33 C
ATOM 1595 N SER C 99 25.536 47.250 48.136 1.00 87.43 N
ATOM 1596 CA SER C 99 24.651 46.115 48.356 1.00 87.04 C
ATOM 1597 C SER C 99 25.277 44.886 47.711 1.00 87.33 C
ATOM 1598 O SER C 99 26.283 44.983 46.992 1.00 87.10 O
ATOM 1599 CB SER C 99 23.270 46.364 47.749 1.00 87.59 C
ATOM 1600 OG SER C 99 22.409 46.991 48.685 1.00 88.68 O
 |
| Рис.5. Участок цепи С с отсутствующими остатками (остатки 75-99) |
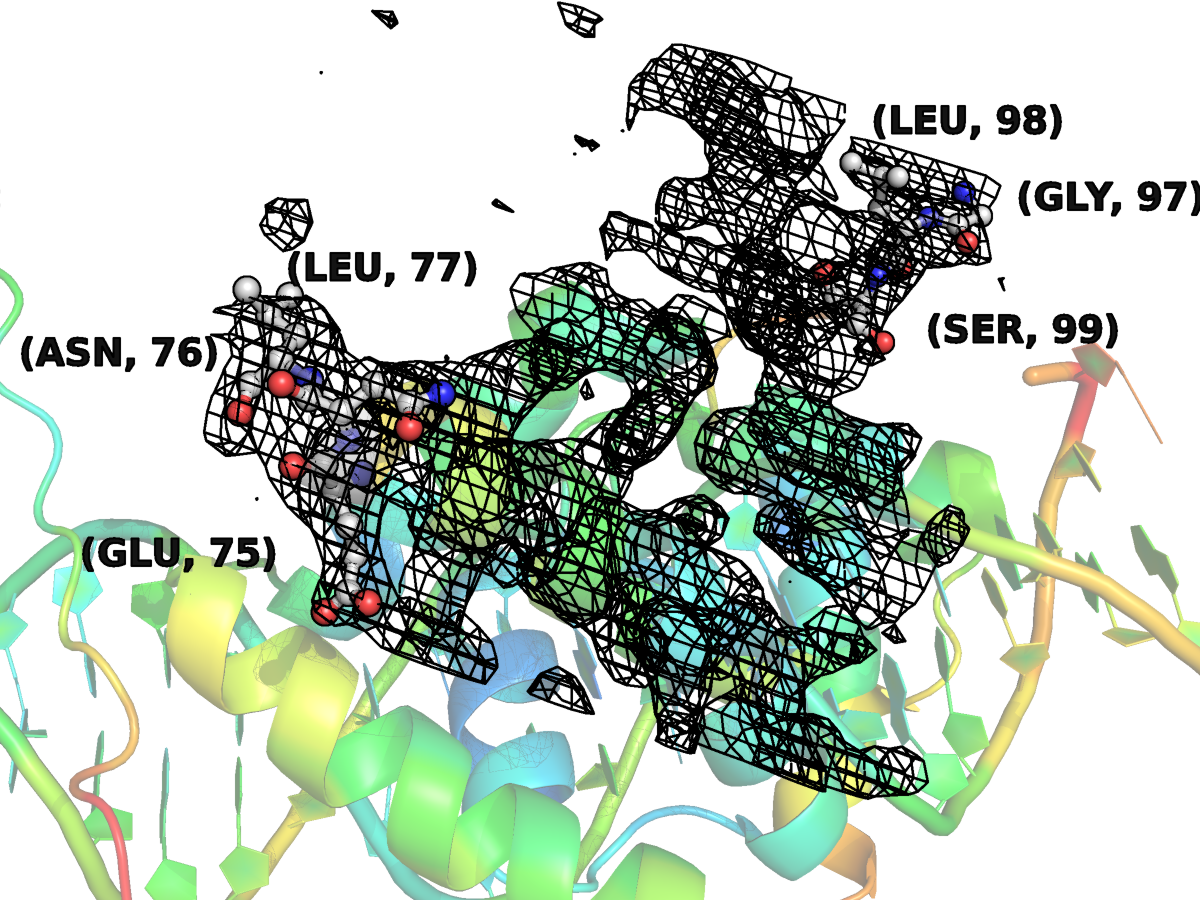 |
| Рис.6.1. Участок цепи С с отсутствующими остатками с показанной картой ЭП (уровень подрезки ЭП 1), покраска по В-фактору |
 |
| Рис.6.2. Участок цепи С с отсутствующими остатками с показанной картой ЭП (уровень подрезки ЭП 1), покраска по В-фактору |
 |
| Рис.6.3. Участок цепи С с отсутствующими остатками с показанной картой ЭП (уровень подрезки ЭП 1), покраска по В-фактору |
- Комплекс ДНК с доменами POU и HMG транскрипционных факторов
- 2005 г. (статья)
- Разрешение – 1.54 А
- Остаток Gln21С имеет две альтернативные конформации.
В PDB-файле альтернативные конформации Gln21C обозначены буквами A или B перед названием остатка:
#Описание атомов Gln21С в PDB-файле
ATOM 1404 N GLN C 21 20.209 31.249 32.802 1.00 17.96 N
ATOM 1405 CA AGLN C 21 19.364 32.425 32.992 0.50 19.11 C
ATOM 1406 CA BGLN C 21 19.326 32.397 33.057 0.50 20.53 C
ATOM 1407 C GLN C 21 19.993 33.363 34.029 1.00 16.88 C
ATOM 1408 O GLN C 21 19.948 34.602 33.860 1.00 16.48 O
ATOM 1409 CB AGLN C 21 17.931 32.039 33.379 0.50 20.64 C
ATOM 1410 CB BGLN C 21 17.973 31.950 33.643 0.50 22.06 C
ATOM 1411 CG AGLN C 21 17.034 33.216 33.781 0.50 20.74 C
ATOM 1412 CG BGLN C 21 16.993 31.438 32.609 0.50 23.71 C
ATOM 1413 CD AGLN C 21 17.055 34.359 32.789 0.50 24.73 C
ATOM 1414 CD BGLN C 21 15.641 31.013 33.167 0.50 26.46 C
ATOM 1415 OE1AGLN C 21 17.097 34.149 31.567 0.50 20.63 O
ATOM 1416 OE1BGLN C 21 15.470 30.787 34.370 0.50 32.44 O
ATOM 1417 NE2AGLN C 21 17.048 35.589 33.309 0.50 17.33 N
ATOM 1418 NE2BGLN C 21 14.666 30.890 32.270 0.50 27.89 N
В WHAT IF Check report сервиса PDBREPORT
остатки Gln21 всех четырех цепей указаны под предупреждением об их необычном пространственном окружении (an unusual packing environment). Окружение каждого остатка сравнивается со средним окружением, которое вычисляется по окружениям остатков одного типа в хороших PDB-файлах.
#WHAT IF Check report
Warning: Abnormal packing environment for some residues
21 GLN ( 21-) A -5.55
98 GLN ( 21-) B -5.53
173 GLN ( 21-) C -5.52
249 GLN ( 21-) D -5.49
122 ARG ( 45-) B -5.45
303 GLU ( 75-) D -5.05
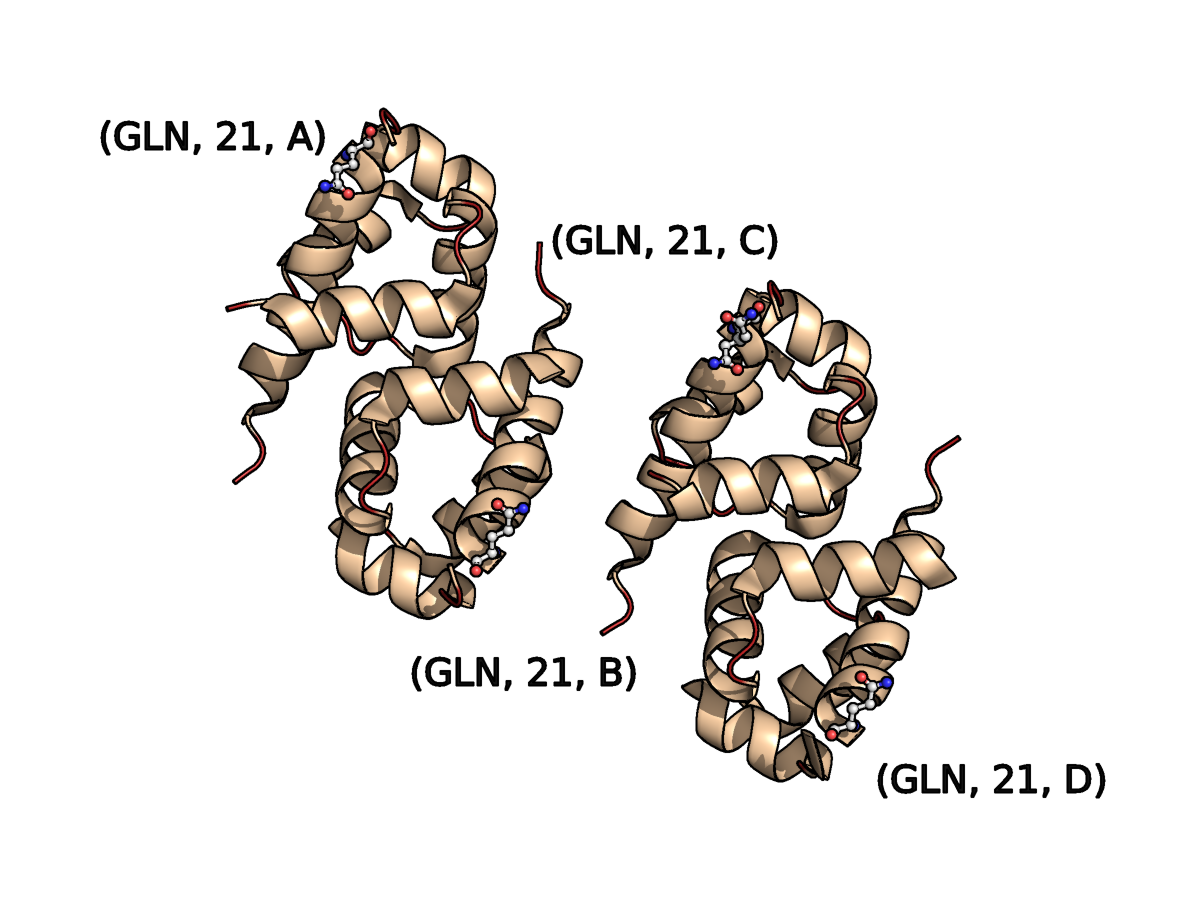 |
| Рис.7. Общий вид структуры 2B5A с обозначенными Gln21 всех цепей< |
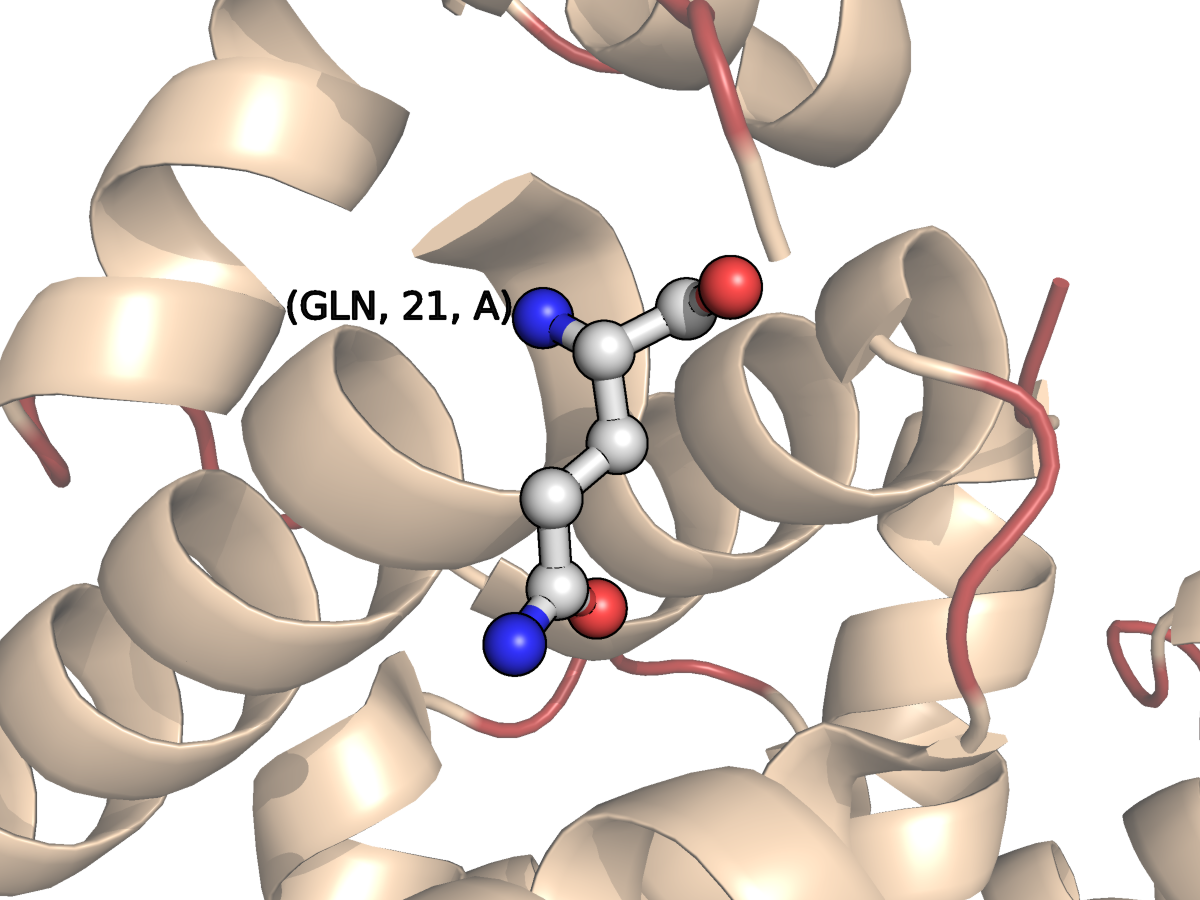 |
| Рис.8.1. Остаток Gln21 цепи A (с одним вариантом конформации) |
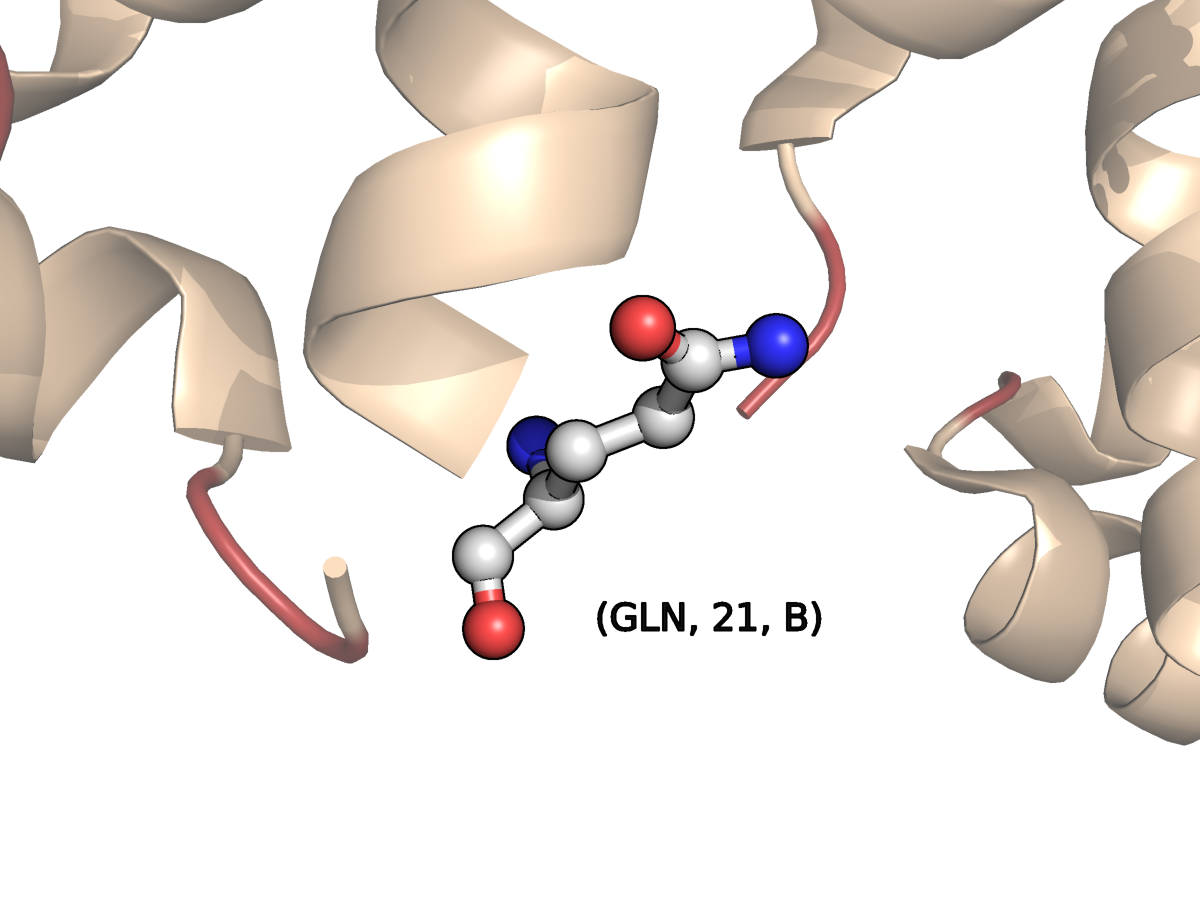 |
| Рис.8.2. Остаток Gln21 цепи B (с одним вариантом конформации) |
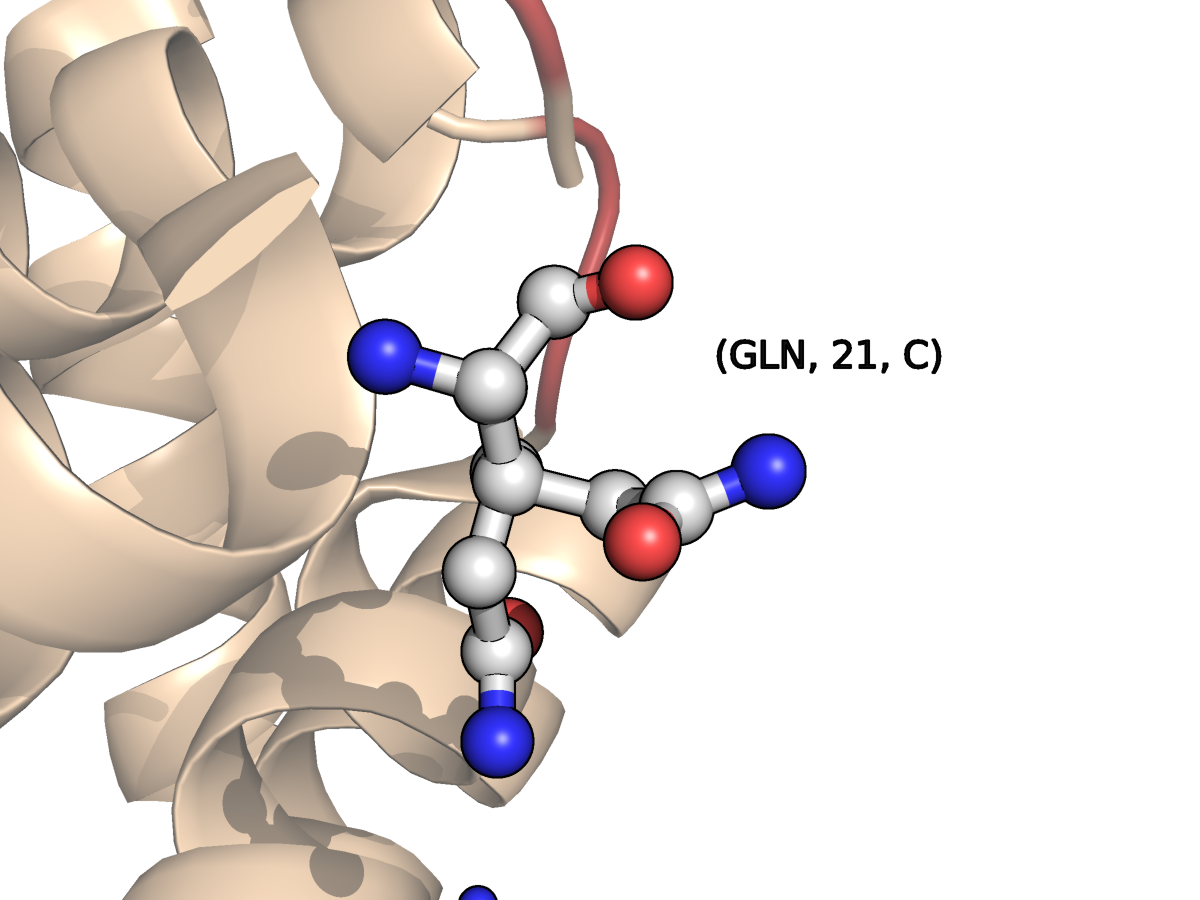 |
| Рис.8.3. Остаток Gln21 цепи С (с двумя вариантами конформации) |
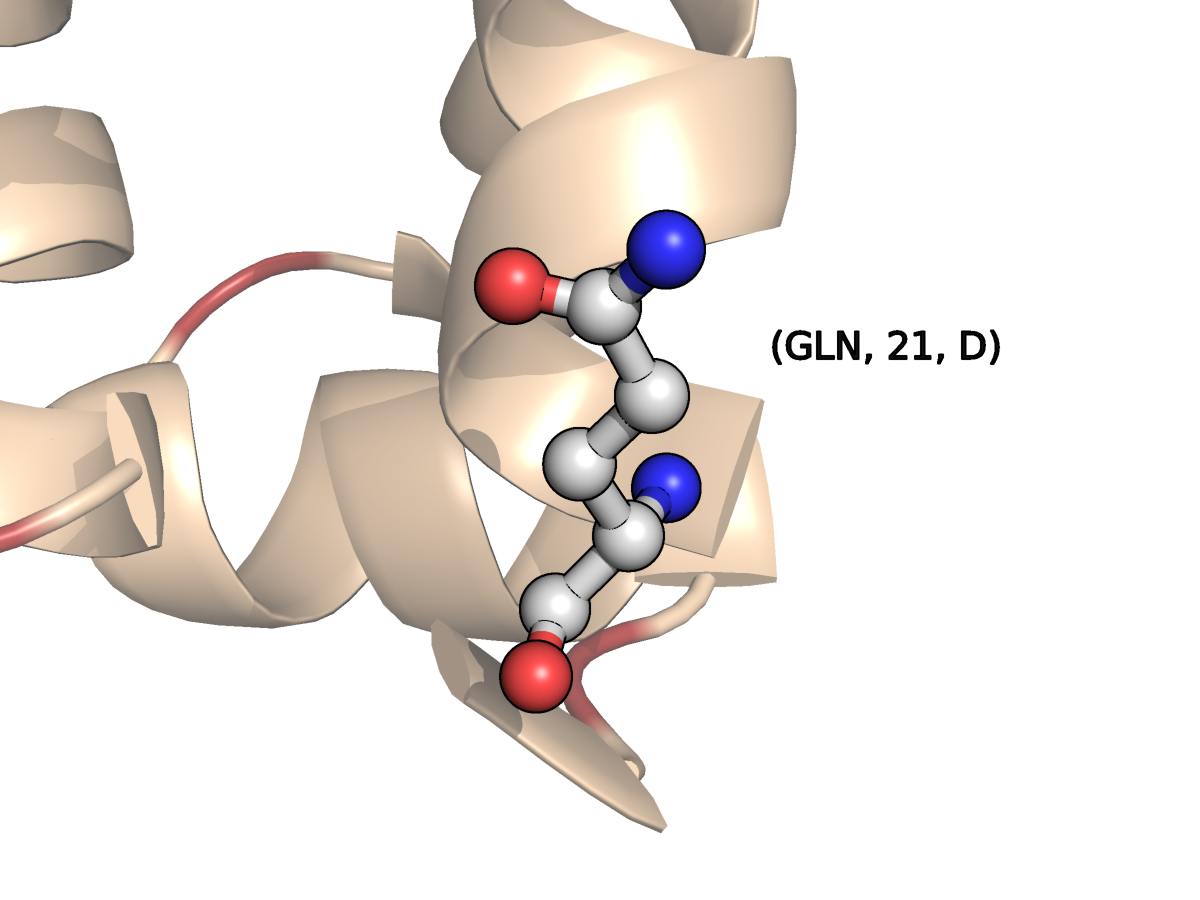 |
| Рис.8.4. Остаток Gln21 цепи D (с одним вариантом конформации)
|
Сравнение значений коэффициента заполнения (occupancy) и температурного фактора (B-factor) в структурах разного разрешения
С помощью Advanced search => Search by X-ray resolution на сайте PDB были найдены структуры разрешения до 1 А и от 2.0 А до 2.5 А. Были выбраны две модели:
- 4PSY(0.85 А) - дигидрофолатредуктаза из E.coli; мономер; 159 остатков.
- 1J78 (2.31 А) - витамин D связывающий белок; монодимер; 458 остатков.
 |
| Рис.9. Общий вид структуры 4PSY (0.85 А) |
 |
| Рис.10. Общий вид структуры 1J78 (2.31 А) |
Из PDB-файлов были получены значения температурных факторов (B-factors) и коэффициентов заполнения (occupancy) для всех атомов и представлены в виде гистограммы:
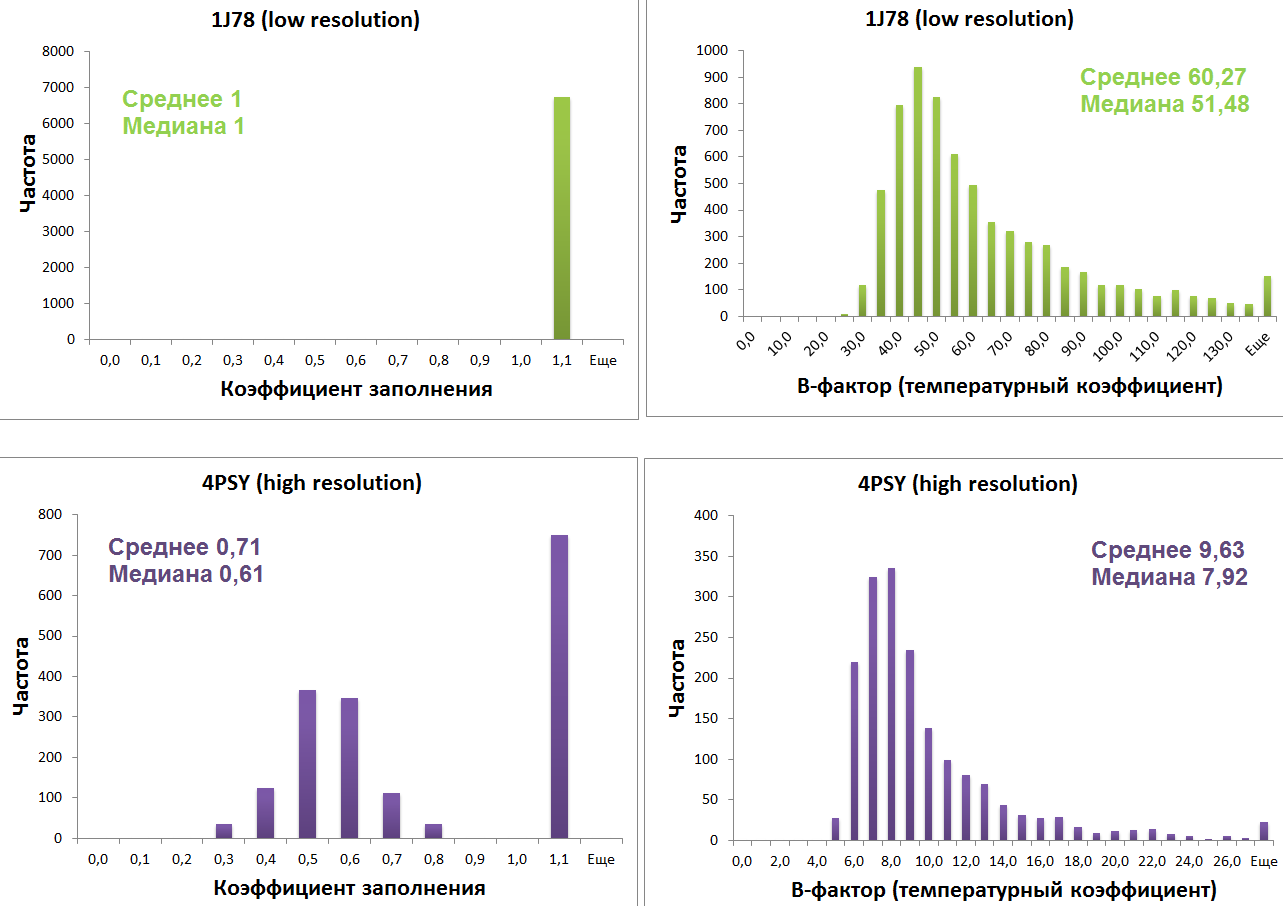 |
| Рис.12. Гистограммы частот значений коэффициентов заполнения для структур 4PSY (0.85 А) и 1J78 (2.31 А) |
Таким образом, среднее значение температурного фактора для модели с меньшим разрешением меньше (4PSY 0.85 А), чем для можели с большим разрешением (1J78 2.31 А). Температурный фактор (фактор атомного смещения) показывает, насколько восстановленная электронная плотность широко "размазана" вокруг атома. Однако, значение температурного фактора зависит не только от разрешения, но и от самой структуры, условий и качества эксперимента.
Коэффициенты заполнения всех атомов менее точной модели (1J78 2.31 А) равны единице, при этом с увеличением разрешения (4PSY 0.85 А) можно лучше различать остатки с альтернативными конформациями, т.е. положения отдельных атомов определяются более неоднозначно, и значения для большинства атомов меньше 1.
|















