Оценка возможностей Sculpting
Wizard->Demo->Sculpting
Предлагается посмотреть опцию sculpting для белка с SS-связью, визуализированными двойными связями и отображенными Ван-дер-Ваальсовыми радиусами атомов. При помощи обозначенным прямо в рабочей области действий можно "издеваться" над структурой пептида (изменять положения атомов и вращать по торсионным углам) (Рис.2)
| Рис.1 |
| Рис.2 |
Sculpting на 1LMP
Теперь попробуем это на фрагменте белка из структуры 1LMP. Мышь - в режим редактирования: Mouse > 3 Button Editing; режим скульптуры: Wizard > Sculpting.
При этом межатомные напряжения показываются в виде красных точек и дисков (Toggle Bumps) и автоматически анимированно "расслабляются" (Toggle Sculpting). Выбирем для редактирования область с лигандом: область структуры для редактирования - зеленый, "амортизатор" изменений - голубой, не рассматривается - серый (Рис.3). Можно "вытащить" лиганд из кармана для его связывания (Рис. 4).
| Рис.3 |
| Рис.4 |
Введение мутации в белок
Оценка зоны контакта
Атомы на расстоянии менее 3.5 Å показаны в модели lines (color orange). Красные и синие шарики - атомы O и N среди множества близких атомов. Желтым - остатки, содержащие близкие атомы, целиком. Желтыми линиями - возможные водородные связи между лигандом и белком (distance, порог 3.5).
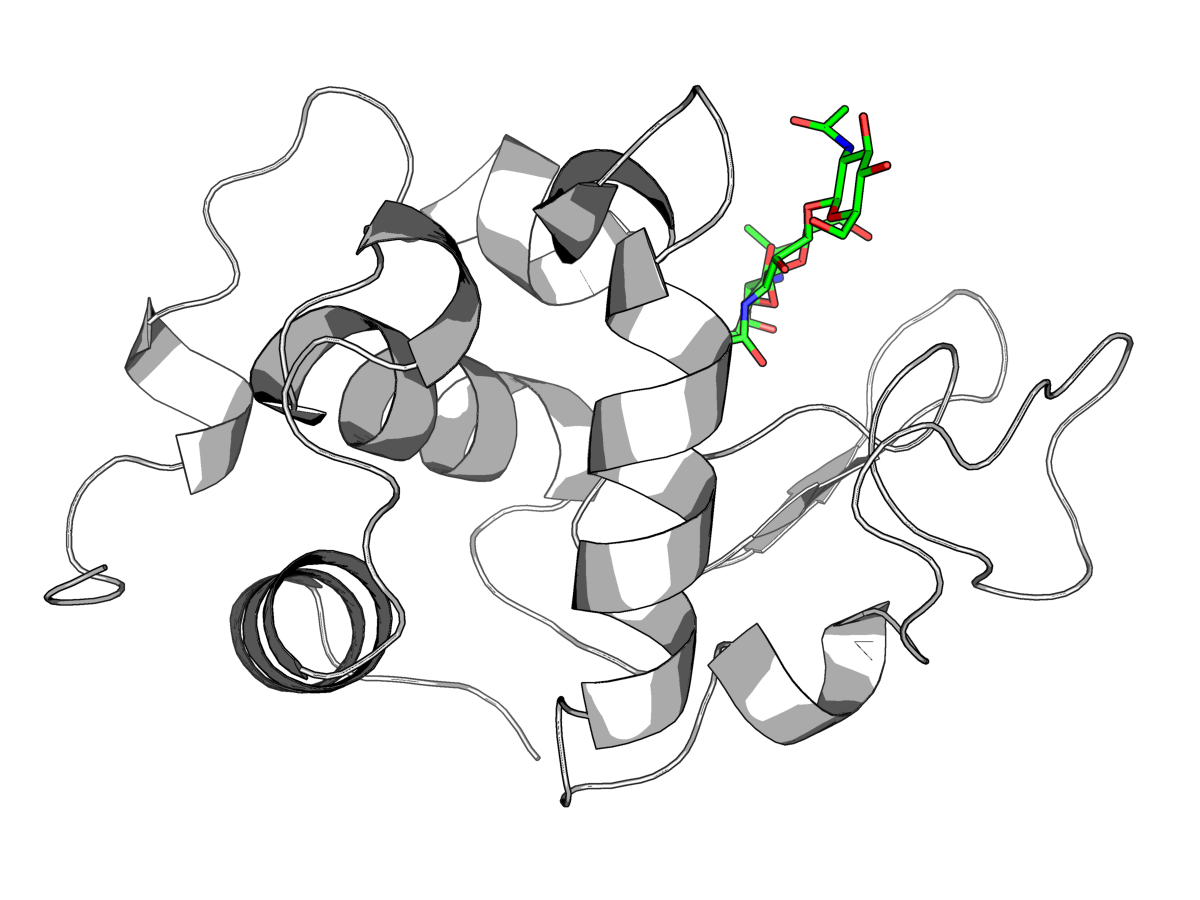 |
| Рис.5.Изображение структуры комплекса модели 1LMP |
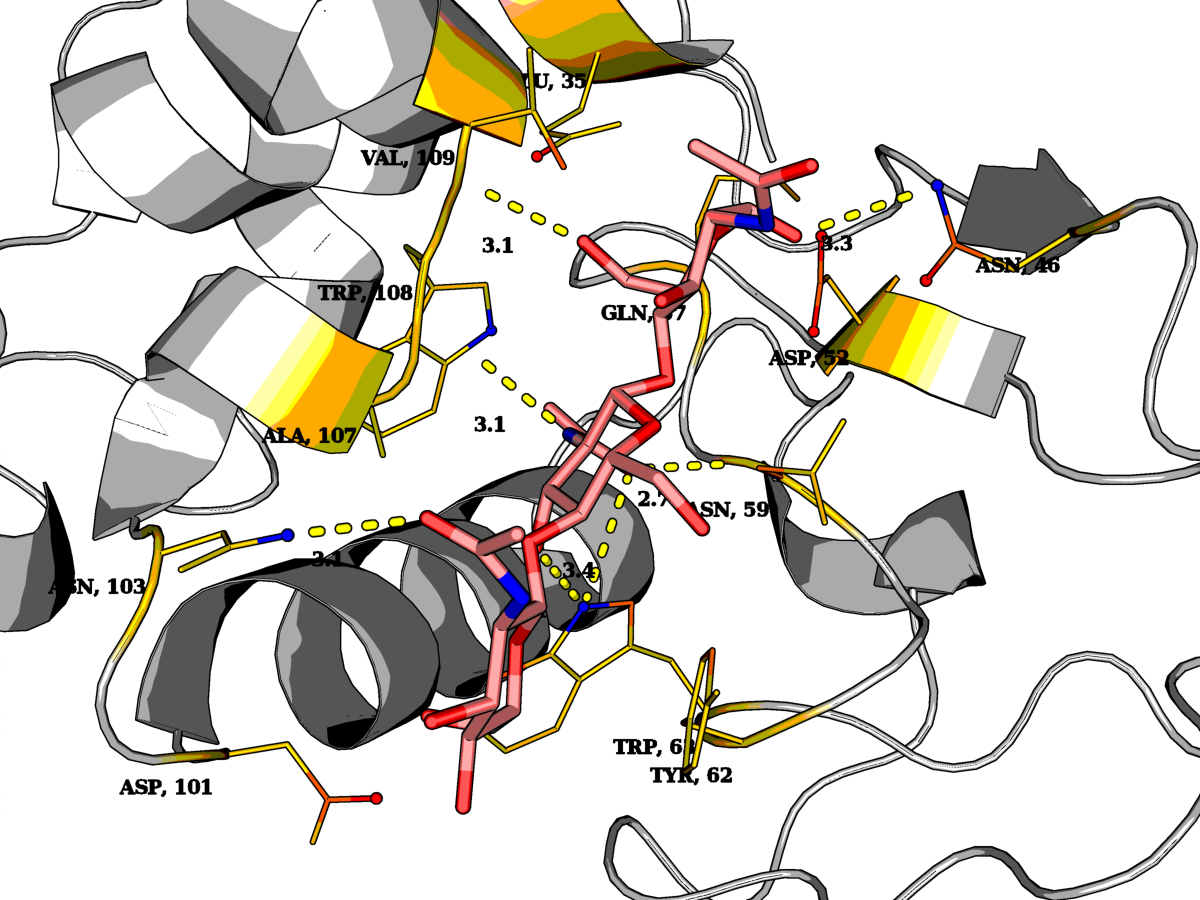 |
| Рис.6. Контакты лиганда с белком |
Wizard->Mutagenesis
Заменим мутацией Asn46 на Ala (маленький и неполярный, и вообще другой). Контакт, таким образом, совсем пропадает.
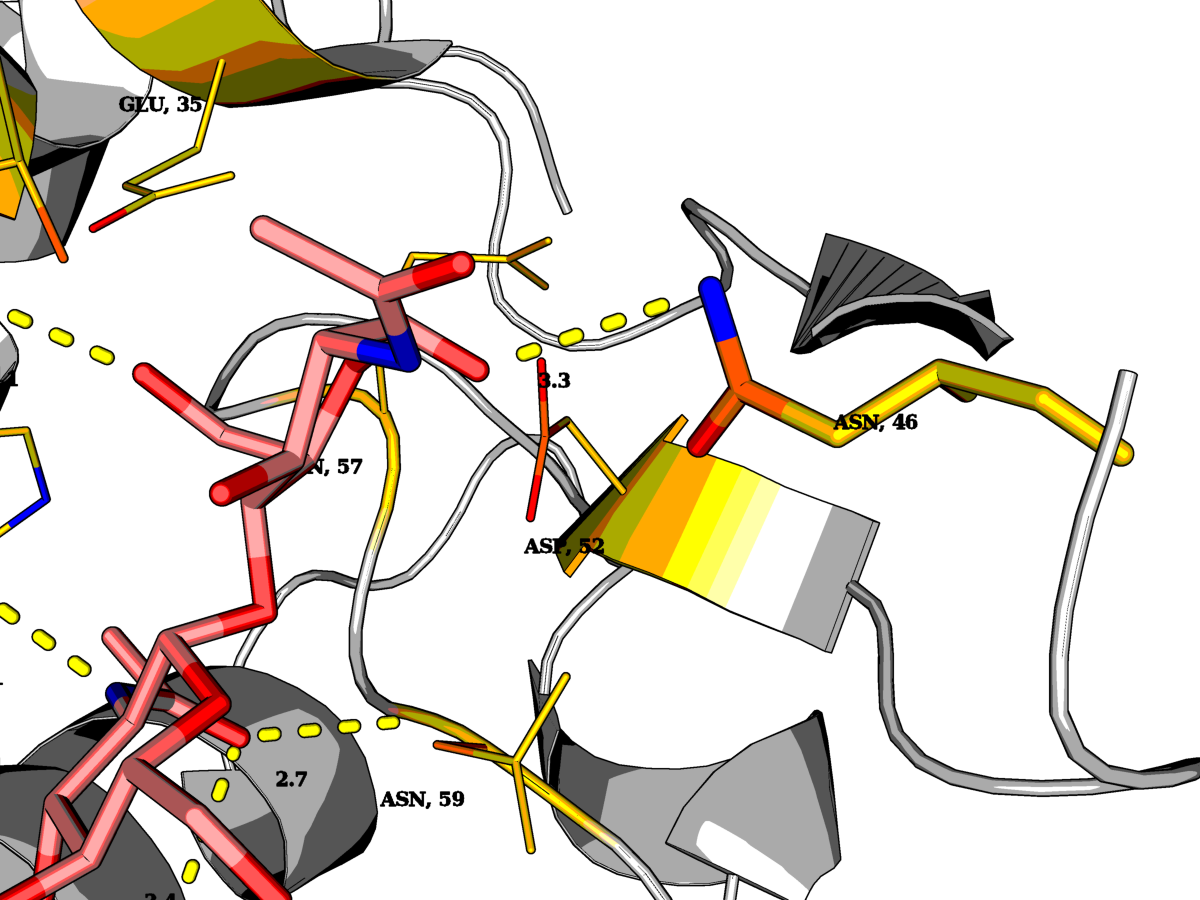 |
| Рис.7.Остаток ASN 46 модели 1LMP и его водородная связь с лигандом |
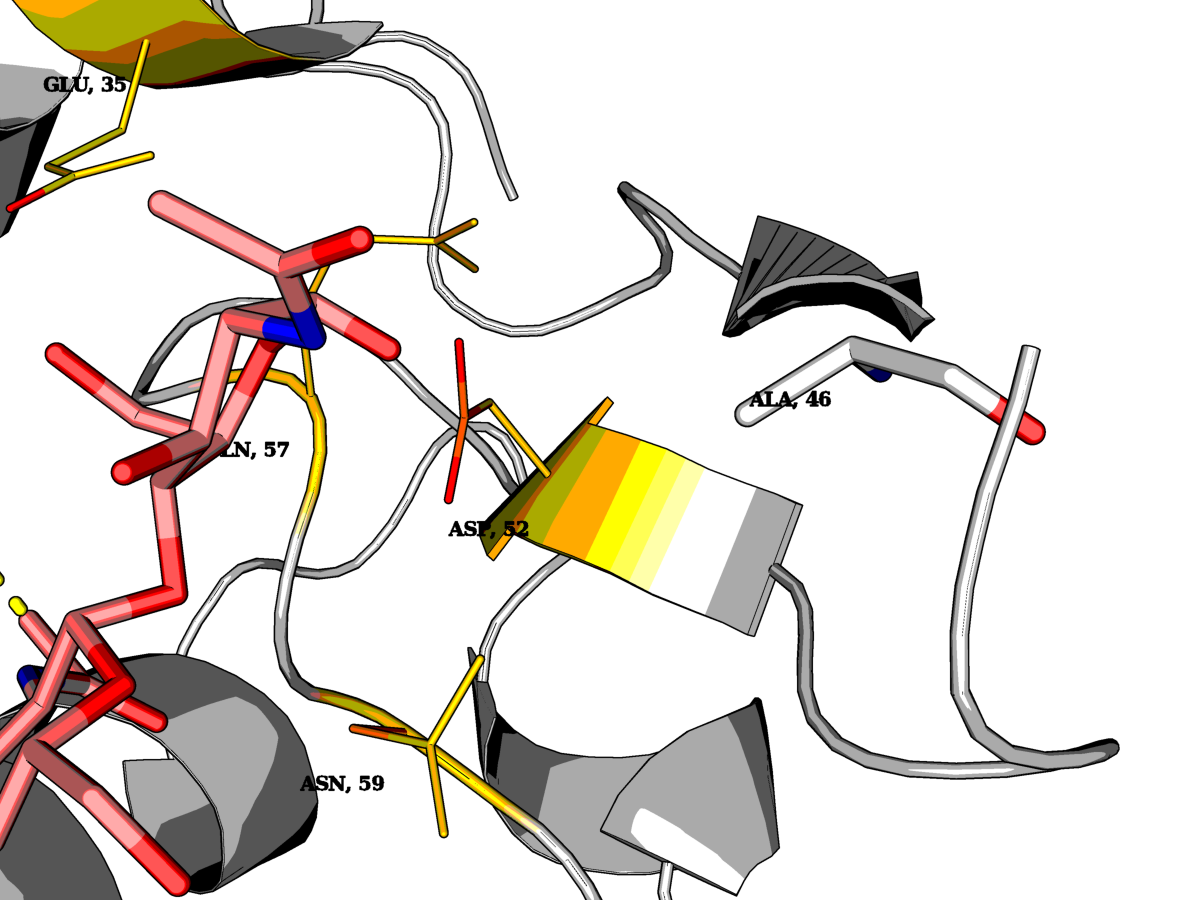 |
| Рис.8. Мутантный ALA 46 модели 1LMP. Водородная связь пропадает. |
Создание анимационного ролика
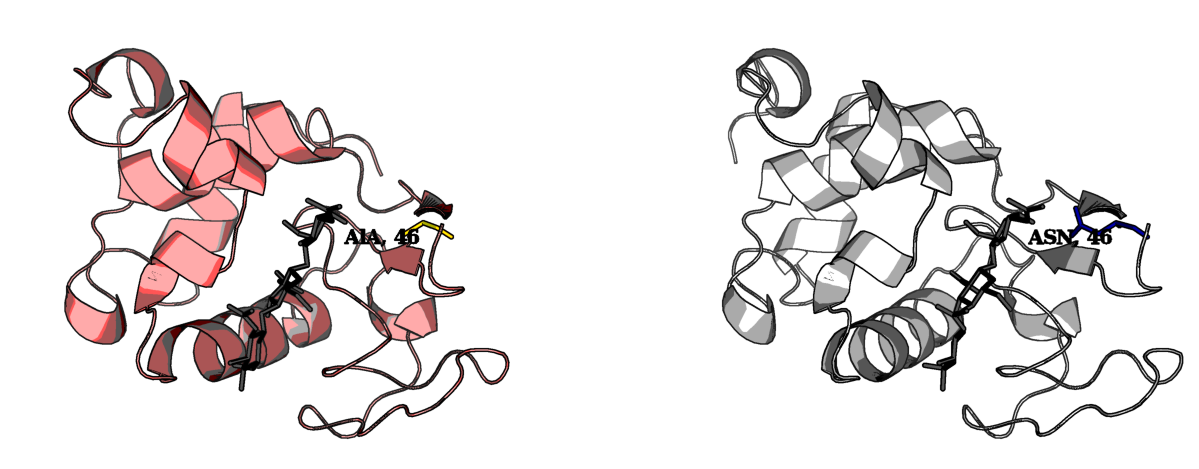 |
| Рис.9. Мутантный (слева) и нативный (справа) 1LMP |
Чтобы объединить получившиеся изображений в один видеоролик, поспользуемся командой:
ffmpeg -i 1LMP_mutation0%03d.png -b:v 5000000 -c:v libx264 movie_1lmp.mp4
Присоединение флуоресцентной метки
Флуоресцентную метку присоединим к белку через сложноэфирную связь, меняя торсионный угол так, чтобы не возникало явный структурых перекрытий. Метку скачаем отсюда.
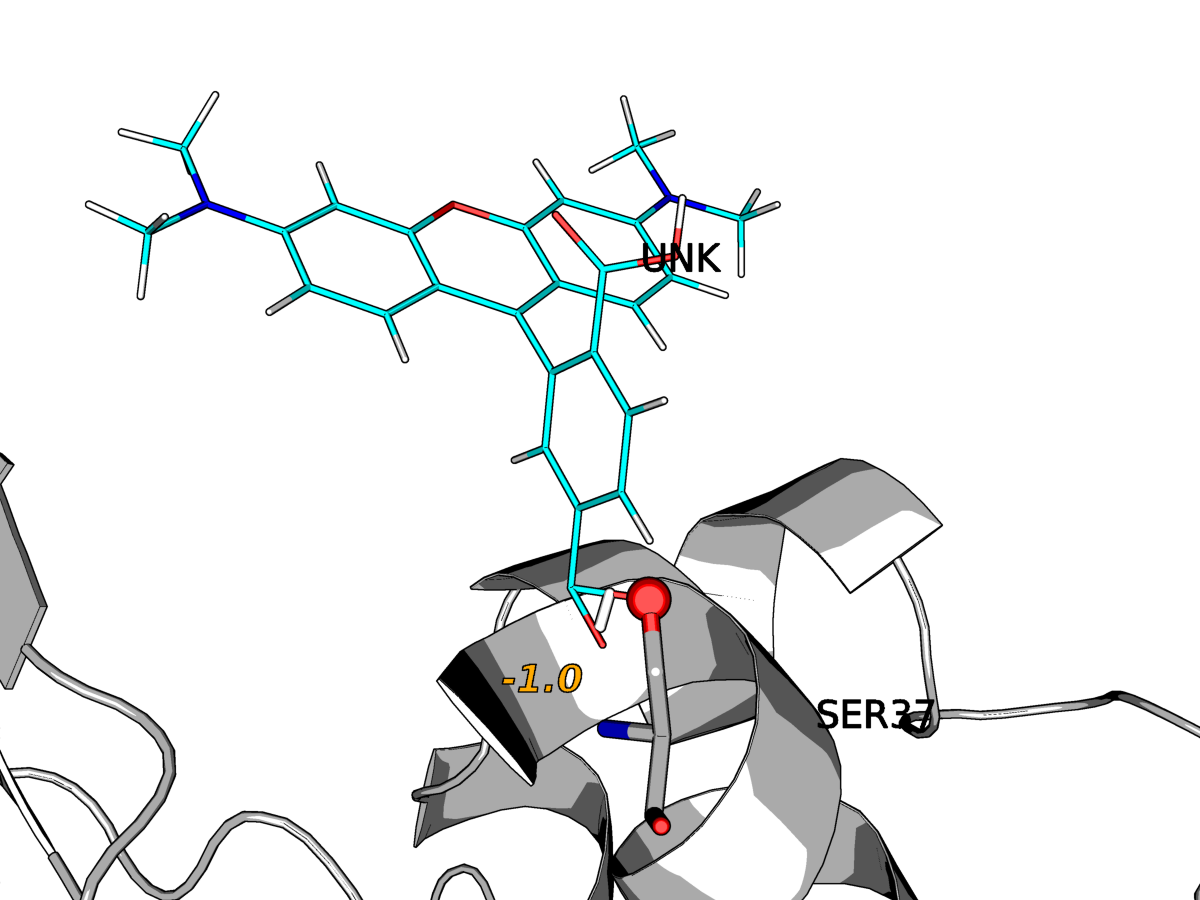 |
| Рис.10. Присоединенная к 1LMP флуоресцентная метка TAMRA |
Вся работа в PyMol была записана в скрипт: script_1.pml