Cеми-эмпирические вычисления: Mopac
Суть задания состоит в поэтапном освоении возможностей Mopac, как пакета для оптимизации структуры молекул и расчёта некоторых свойств.Оптимизация структуры порфирина
Создадим файл porphyrin.smi со описанием структуры порфирина:c1cc2cc3ccc(cc4ccc(cc5ccc(cc1n2)[nH]5)n4)[nH]3 porphyrinНа основе этого описания c помощью программы из openbabel построим 3D структуру порфирина:
obgen porphyrin.smi > porphyrin.molВ полученной стуктуре при помощи PyMol удалим лишние водороды и сохраним как .pdb.
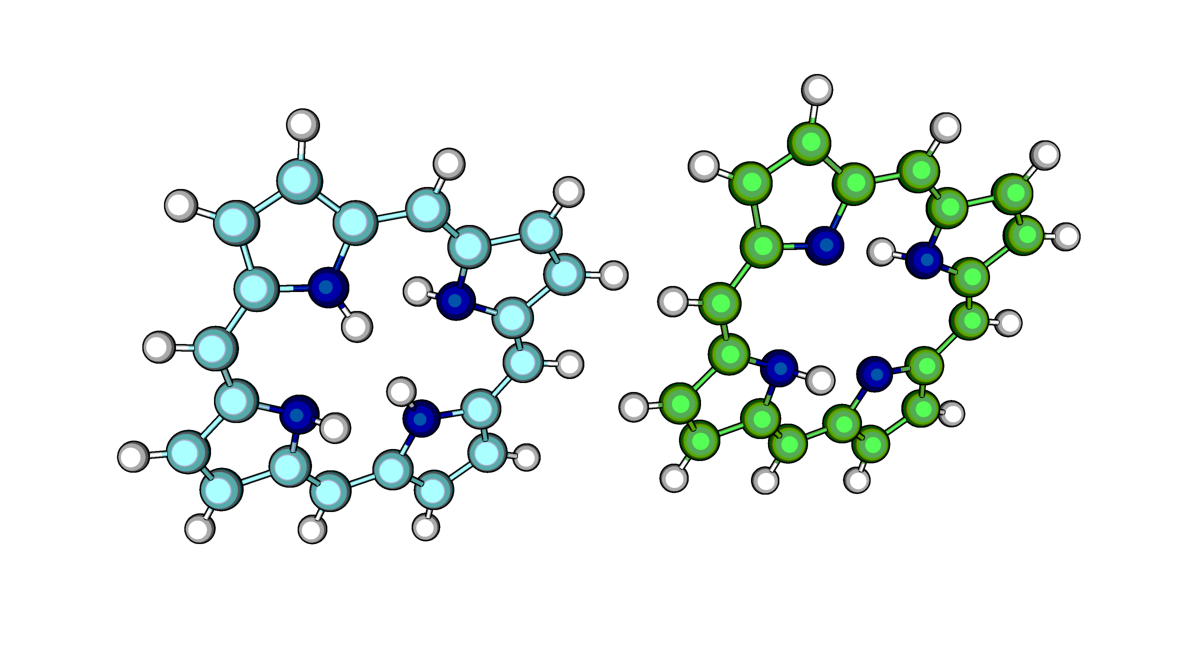 |
| Рис.1. Структура порфирина из файла porphyrin.mol (слева), с правильным количеством водородов при атомах азота (справа) |
babel -ipdb porphyrin.pdb -omop porphyrin_opt_pm6.mop -xk "PM6" babel -ipdb porphyrin.pdb -omop porphyrin_opt_am1.mop -xk "AM1"Запустим Mopac:
MOPAC2009.exe porphyrin_opt_pm6.mop babel -imopout porphyrin_opt_pm6.out -opdb porphyrin_opt_pm6.pdb MOPAC2009.exe porphyrin_opt_am1.mop babel -imopout porphyrin_opt_am1.out -opdb porphyrin_opt_am1.pdb
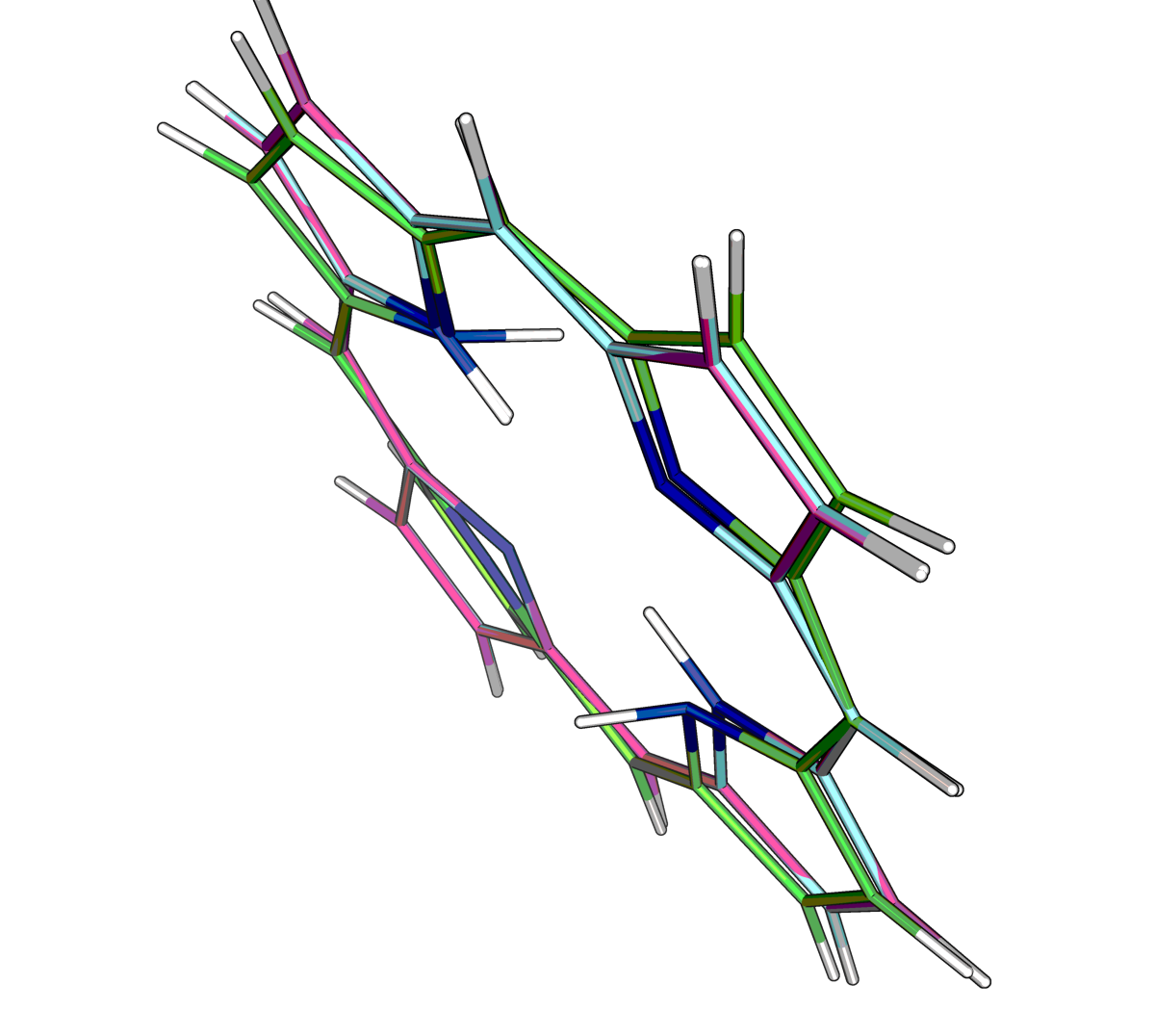
 |
| Рис.2.Сравнение структуры порфирина из файла porphyrin.pdb (зеленый) и оптимизированной при помощи MOPAC версии (голубой - PM6, розовый - AM1) |
Расчет возбужденных состояний порфирина
Создадим копию файла porphyrin_opt_pm6.mop из предыдущего задания - porphyrin_opt_pm6_spectr.mop Добавим в конец строки:cis c.i.=4 meci oldgeo some descriptionИ запустим Mopac:
MOPAC2009.exe porphyrin_opt_pm6_spectr.mopВ конце выходного файла есть таблица со значениями энергий дляэлектронных переходов:
STATE ENERGY (EV) Q.N. SPIN SYMMETRY POLARIZATION
ABSOLUTE RELATIVE X Y Z
1+ 0.000000 0.000000 1+ SINGLET ????
2 1.914378 1.914378 1 TRIPLET ????
3 2.266727 2.266727 2 SINGLET ????
4 2.464043 2.464043 2 TRIPLET ????
5 2.824240 2.824240 3 TRIPLET ????
6 3.363029 3.363029 4 TRIPLET ????
7 3.390688 3.390688 3 SINGLET ???? 0.2018 0.2355 0.0011
8 3.670048 3.670048 4 SINGLET ???? 2.3996 2.0312 0.0129
9 3.871030 3.871030 5 SINGLET ???? 1.5409 1.7989 0.0090
Для расчета длин волн переходов воспользуемся формулой:
E = h*n
Þ l = c/v = c*h/E =
()2.99792458*1014мкм/с * 4.135667517*10-15 эВ*с) / Е = 1,23984193 / Е мкм
Получаем вот такой список длин волн переходов в нм: [338 нм; 338 нм; 366 нм; 366 нм; 369 нм; 439 нм; 503 нм; 547 нм; 648 нм;]
Определение геометрии хинона и его дианиона
Определим геометрию хинона с помощью obgen и Mopac.echo "O=C1C=CC(=O)C=C1 quinone" > quinone.smi obgen quinone.smi > quinone.mol babel -ipdb quinone.pdb -omop quinone_opt_pm6.mop -xk "PM6" MOPAC2009.exe quinone_opt_pm6.mop babel -imopout quinone_opt_pm6.out -opdb quinone_opt_pm6.pdb
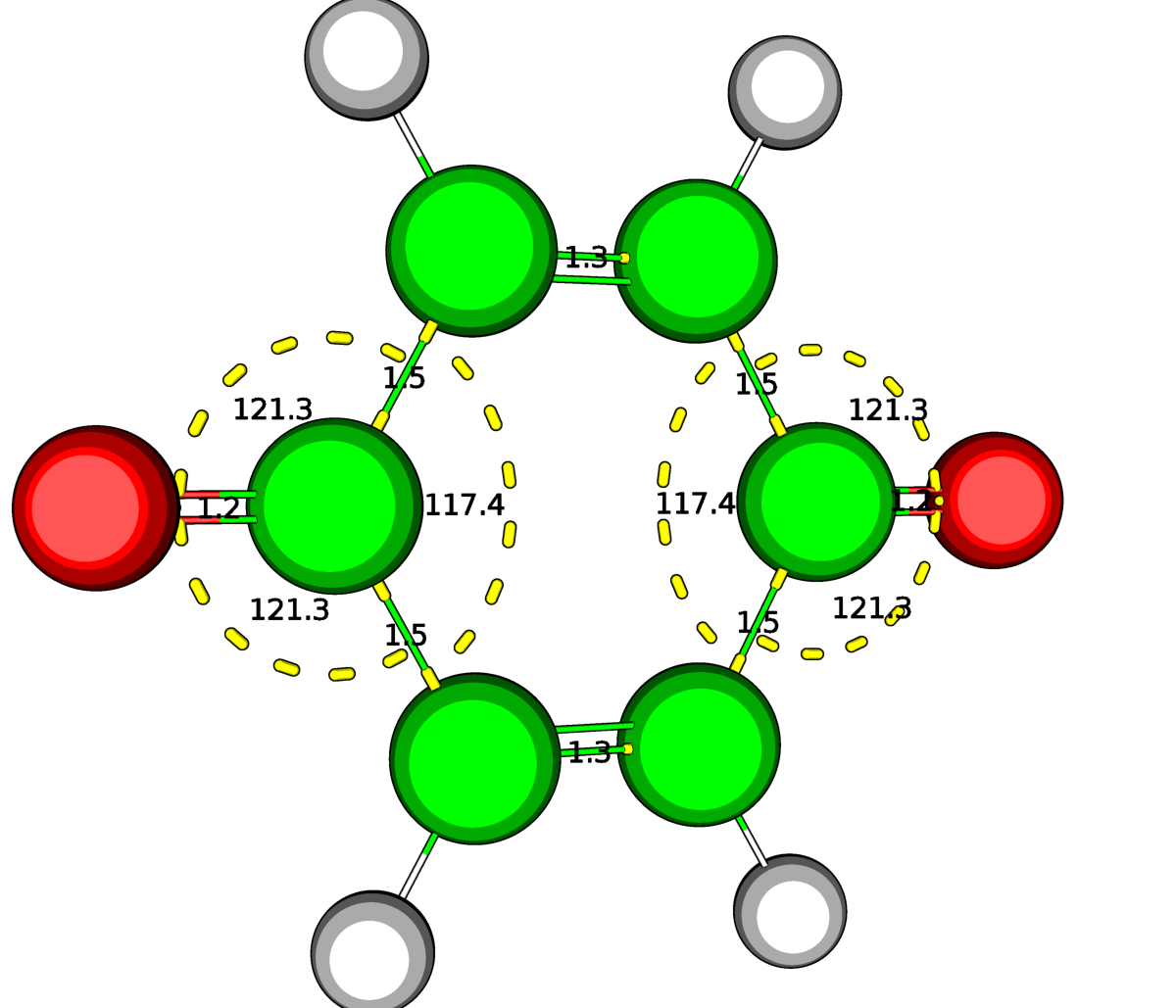 |
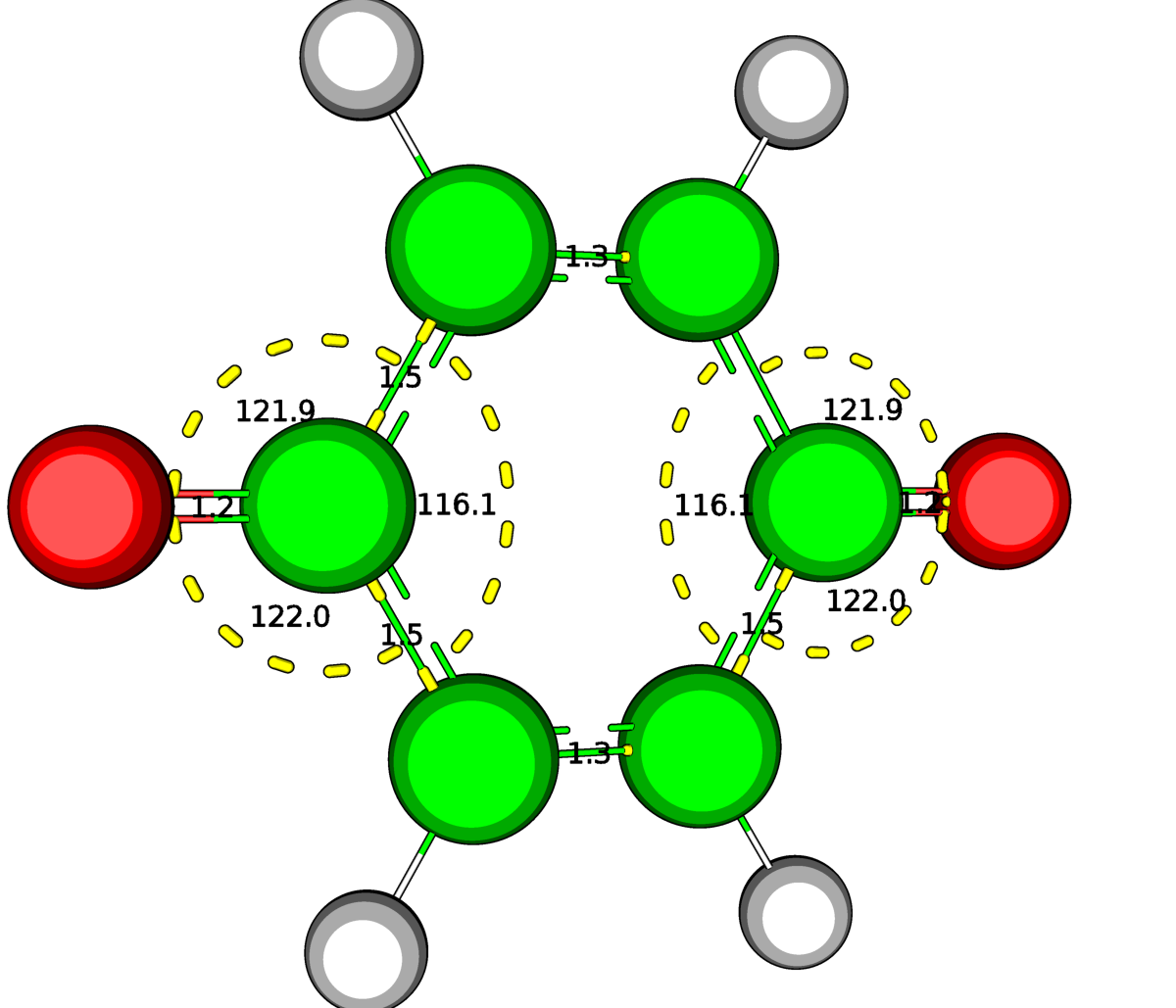 |
| Рис.3.Структура хинона, построенная по SMILES при помощи obgen | Рис.4.Структура хинона, оптимизированная при помощи Mopac (PM6) |
Для определения геометрии дианиона этой молекулы сначала в первую строчку файла .mop добавим CHARGE=-2. З атем явным способом укажем те атомы, на которых должен находиться отрицательный заряд, – атомы кислорода:
PM6 CHARGE=-2 quinone.pdb C 2.13000 1 -0.02100 1 -0.03600 1 C 2.86600 1 1.24400 1 0.15900 1 C 4.15800 1 1.25000 1 0.50100 1 C 4.90500 1 -0.00800 1 0.69900 1 C 4.16900 1 -1.27300 1 0.50300 1 C 2.87700 1 -1.27900 1 0.16100 1 O(-) 0.94800 1 -0.02600 1 -0.34900 1 O(-) 6.08700 1 -0.00200 1 1.01300 1 H 2.30400 1 2.15800 1 0.01000 1 H 4.71300 1 2.16900 1 0.64800 1 H 4.73200 1 -2.18700 1 0.65200 1 H 2.32300 1 -2.19800 1 0.01400 1
MOPAC2009.exe quinone_charge.mop babel -imopout quinone_charge.out -opdb quinone_charge.pdb
 |
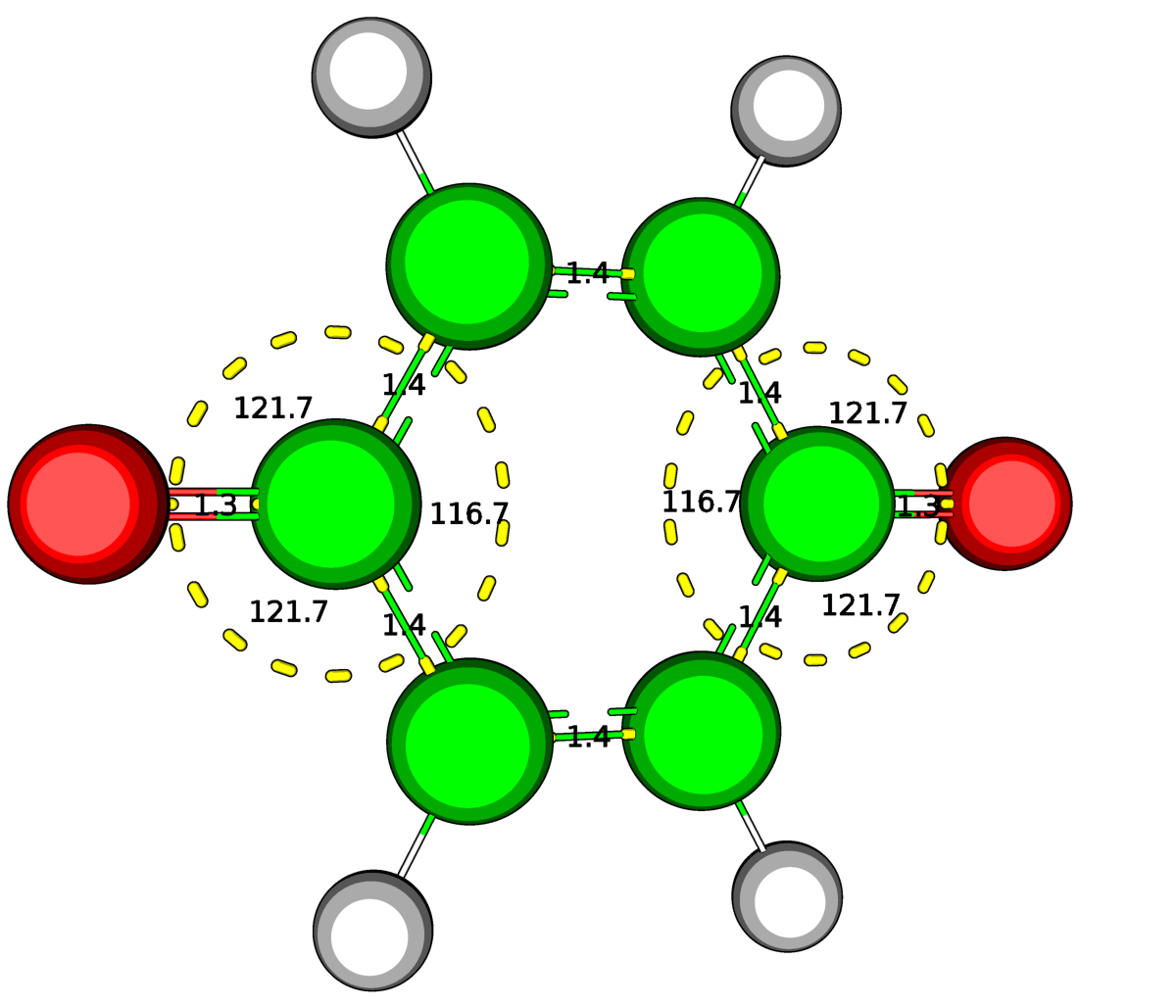 |
| Рис.5.Структура хинона, оптимизированная при помощи Mopac (PM6) | Рис.6.Структура дианиона хинона,оптимизированная при помощи Mopac (PM6) |
Вся работа в PyMol была записана в скрипт: script_1.pml
Тиминовые димеры
Известно, что ультрафиолет может превращать тимины в тиминовые димеры, так же известно, что ДНК фотолиаза при облучении ультрафиолетом востановливает основания тиминов до нормального. Наша цель увидеть переход из димера в тимины при возбуждении системы. Так как вычесления вобуждённых состояний в MOPAC затруденены, мы имитируем возбуждение ионизируя оба кольца, т.е. указывая заряд системы +2. И полученое возбуждённое состояние снова оптимизируем при заряде 0. Таким образом:- Проведем оптимизацию геометрии этого димера, при заряде системы 0.
babel -ipdb td.pdb -omop td_i.mop -xk "PM6" MOPAC2009.exe td_i.mop babel -imopout td_i.out -opdb td_i.pdb
- Проведем оптимизацию результата из пункта i. , при заряде системы +2.
babel -ipdb td_i.pdb -omop td_ii.mop -xk "PM6" sed -i 's/PM6/PM6 CHARGE=+2/' td_ii.mop MOPAC2009.exe td_ii.mop babel -imopout td_ii.out -opdb td_ii.pdb
- Проведем оптимизацию результата из пункта ii. , при заряде системы 0.
babel -ipdb td_ii.pdb -omop td_iii.mop -xk "PM6" MOPAC2009.exe td_iii.mop babel -imopout td_iii.out -opdb td_iii.pdb
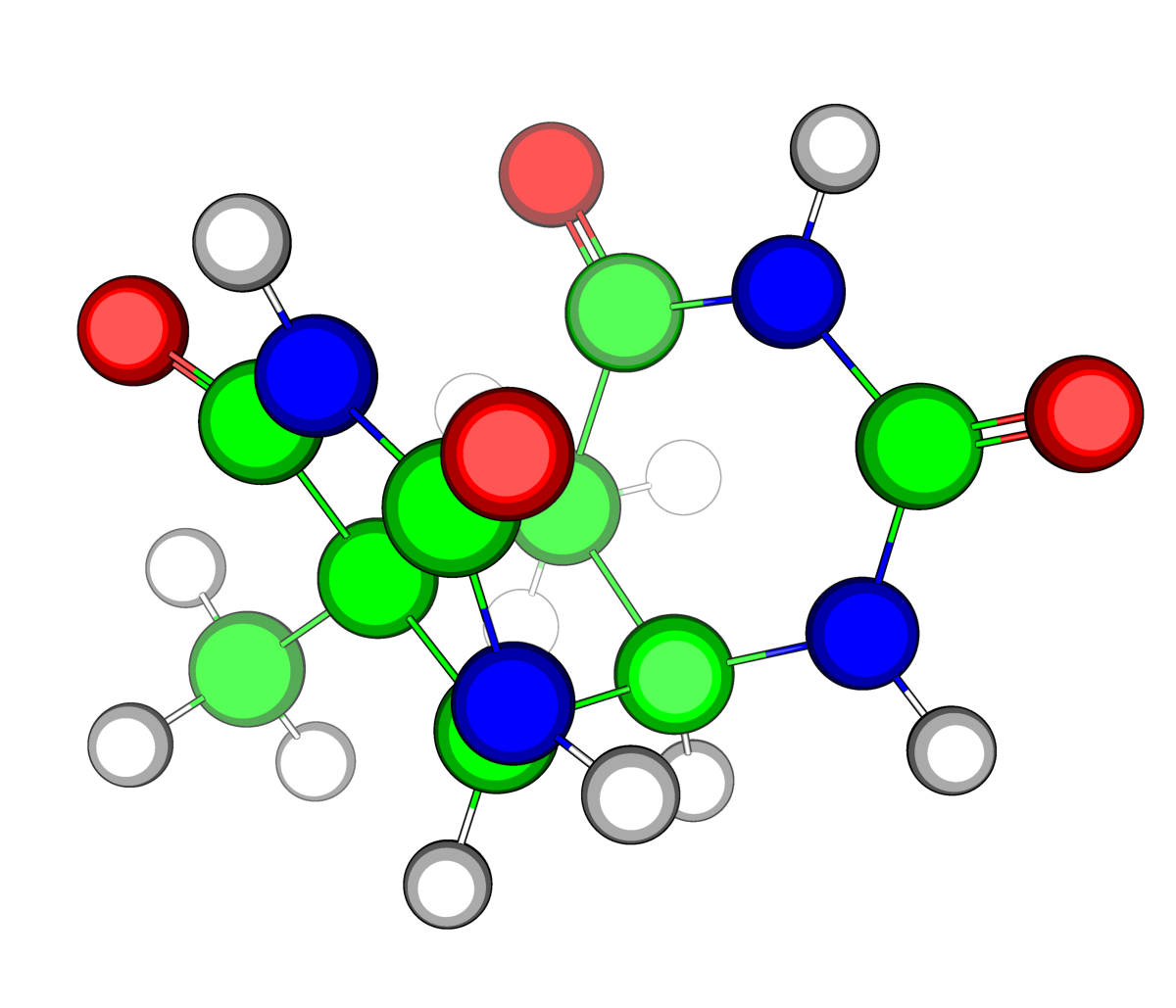 |
| Рис.7. Исходная структура тимидинового димера (заряд = 0, td_i.pdb) |
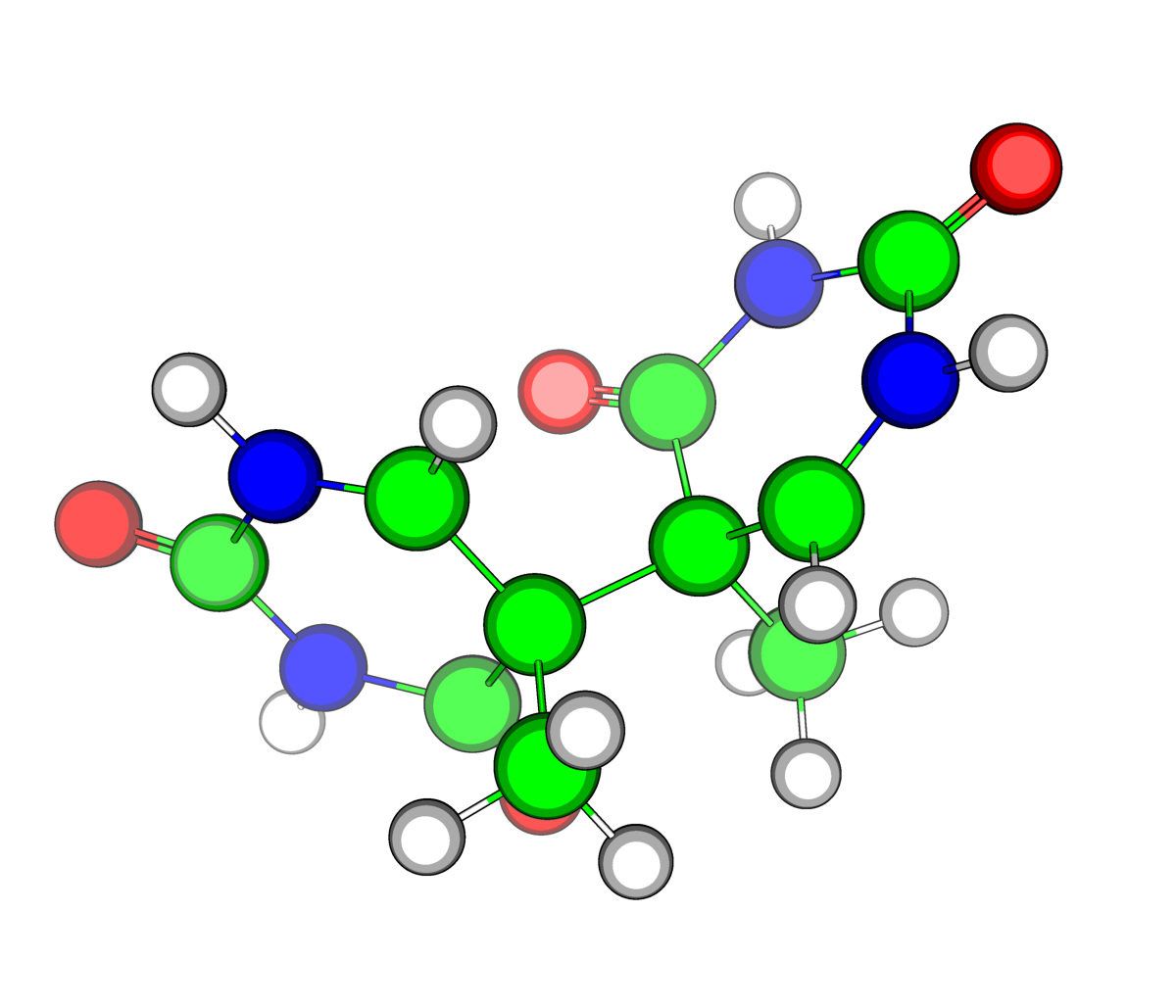 |
| Рис.8. Структура возбужденного димера (заряд = +2, td_ii.pdb) |
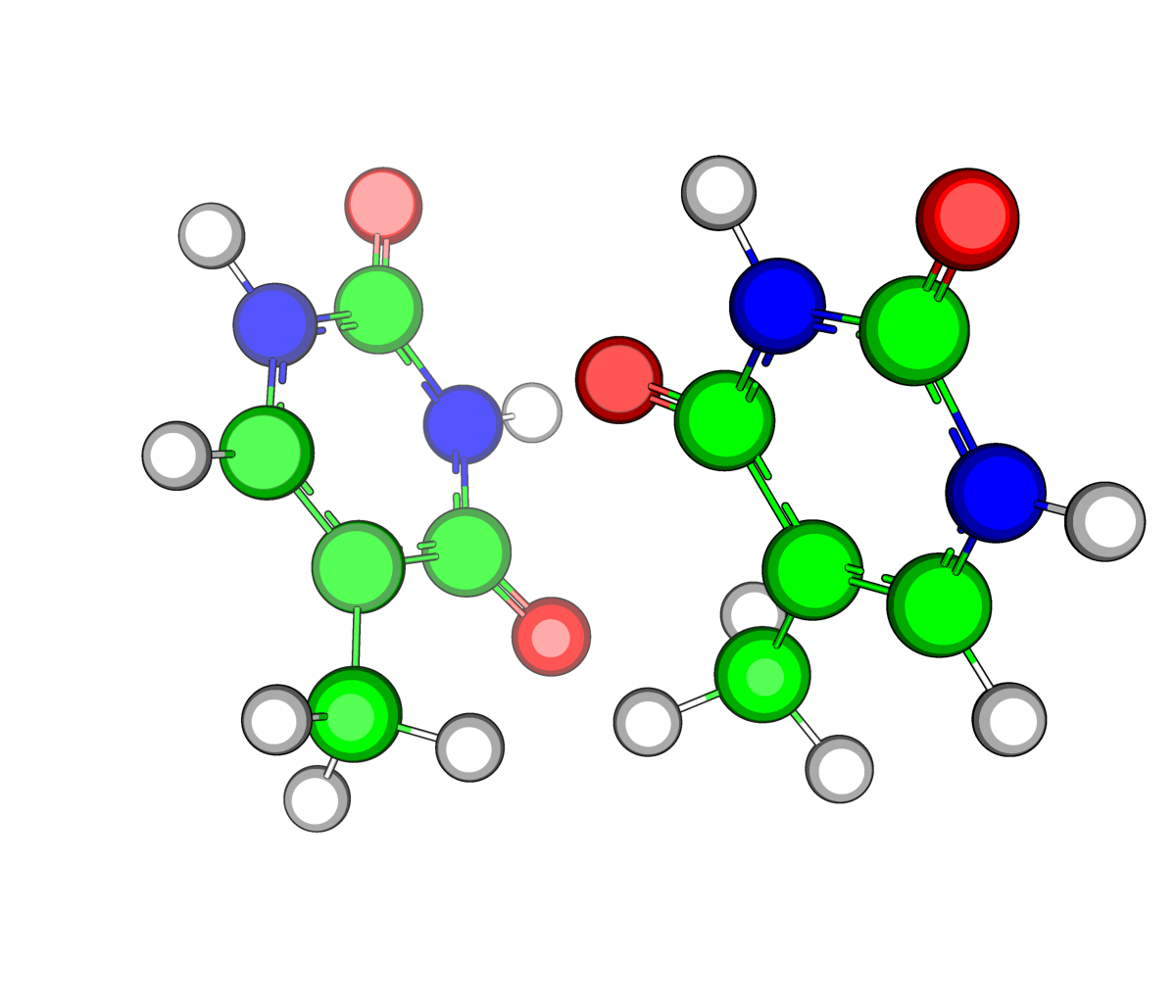 |
| Рис.9. Структура двух отдельным тиминов (заряд = 0, td_iii.pdb) |
Теперь сравним энергии всех трех состояний.
td_i.out -3273.58217 эВ td_ii.out -3253.90834 эВ td_iii.out -3273.69661 эВ
Таким образом, ионизация повысила энергию системы, которая затем скатилась к почти исходному состоянию. Почти, потому что скатилась в другой, отличный от исходного, локальный минимум.
Вся работа в PyMol была записана в скрипт: script_1.pml