Занятие 7. A- и B-формы ДНК
- С помощью программы fiber пакета 3DNA я построила A- и B-форму дуплекса ДНК,
последовательность одной из нитей которого - 4 раза повторенная
последовательность "gatc". Структура дуплекса в А-форме- в файле
gatc-a.pdb, а структура дуплекса в В-форме - в файле gatc-b.pdb.
- Я открыла pdb- файлы в Rasmol и заполнила табличку
A-форма B-форма Файл dnaN.pdb Тип спирали (правая или левая) правая правая правая Шаг спирали (Å) 26 33.75 21.96 Число оснований на виток 11; 10 15 Ширина большой бороздки 7.96 от 4-го до 23-го 18.9 от 7 до 22 18.01 от 8 до 15 Ширина малой бороздки 18.49 от 8-го до 29-го остатка 13.09 от 13 до 23 12.7 от 6 до 23
- Я открыла свой файл (dna29.pdb) в RasMol,
проанализировала изображение и заполнила последний столбец таблицы.
длинна большой бороздки у В-формы ДНК больше длины малой. По шагу спирали и ширине малой бороздки мой дуплекс ближе к А-форме, по ширине большой бороздки- к B-форме.
- С помощью программы find_pair и analyze анализируем три структуры ДНК.
С помощью команд find_pair -t gatc-a.pdb stdout | analyze find_pair -t gatc-b.pdb stdout | analyze find_pair -t dna29.pdb stdout | analyze получили такие результаты: Для gatc-a.pdb Main chain and chi torsion angles: Note: alpha: O3'(i-1)-P-O5'-C5' beta: P-O5'-C5'-C4' gamma: O5'-C5'-C4'-C3' delta: C5'-C4'-C3'-O3' epsilon: C4'-C3'-O3'-P(i+1) zeta: C3'-O3'-P(i+1)-O5'(i+1) chi for pyrimidines(Y): O4'-C1'-N1-C2 chi for purines(R): O4'-C1'-N9-C4 Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 --- --- -157.2 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.7 174.8 41.7 79.0 --- --- -157.2 2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 16 G --- 174.8 41.7 79.1 -147.7 -75.1 -157.2 Для gatc-b.pdb Main chain and chi torsion angles: Note: alpha: O3'(i-1)-P-O5'-C5' beta: P-O5'-C5'-C4' gamma: O5'-C5'-C4'-C3' delta: C5'-C4'-C3'-O3' epsilon: C4'-C3'-O3'-P(i+1) zeta: C3'-O3'-P(i+1)-O5'(i+1) chi for pyrimidines(Y): O4'-C1'-N1-C2 chi for purines(R): O4'-C1'-N9-C4 Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 --- --- -98.0 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -29.9 136.4 31.1 143.4 --- --- -98.0 2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 А- и B-структуры ДНК очень стабильны, это можно видеть по углам поворота относительно оси (они одинаковые). Для dna29.pdb Main chain and chi torsion angles: Note: alpha: O3'(i-1)-P-O5'-C5' beta: P-O5'-C5'-C4' gamma: O5'-C5'-C4'-C3' delta: C5'-C4'-C3'-O3' epsilon: C4'-C3'-O3'-P(i+1) zeta: C3'-O3'-P(i+1)-O5'(i+1) chi for pyrimidines(Y): O4'-C1'-N1-C2 chi for purines(R): O4'-C1'-N9-C4 Strand I base alpha beta gamma delta epsilon zeta chi 1 C --- --- -84.2 135.8 -147.3 -143.4 -115.7 2 G -17.2 148.3 12.7 151.2 -166.6 -127.1 -95.5 3 C -72.7 149.8 72.7 97.1 -148.6 -142.4 -140.4 4 I -21.3 116.8 62.4 128.5 162.1 -73.6 -115.9 5 A -69.1 -127.9 32.3 153.3 159.7 -89.0 -92.0 6 A -66.0 -159.2 53.5 132.5 159.9 -74.3 -95.1 7 T -92.1 -160.5 56.0 88.5 72.7 29.0 -117.7 8 T -164.5 -163.5 79.7 82.4 71.4 43.0 -123.6 9 A -120.3 -173.8 51.9 116.1 -174.8 -87.6 70.5 10 G -63.2 179.9 50.0 150.8 -83.3 147.4 -88.7 11 C -62.5 175.0 15.8 139.9 174.1 -60.8 -106.4 12 G -112.3 -174.9 70.3 92.2 --- --- -111.1 Strand II base alpha beta gamma delta epsilon zeta chi 1 G -30.6 164.1 16.8 79.0 --- --- -134.2 2 C -64.1 145.3 28.8 114.7 -170.4 -101.9 -121.9 3 G -28.7 159.3 37.2 151.4 -107.0 175.1 -90.2 4 A -88.8 -161.2 57.3 106.6 -174.8 -98.2 72.1 5 T -52.6 -158.1 29.4 132.6 170.2 -78.0 -103.4 6 T -58.0 141.8 77.3 74.6 177.1 -66.5 -133.7 7 A -94.7 -130.6 37.1 153.5 -162.6 -117.8 -84.0 8 A -84.1 -157.5 58.6 121.7 118.9 -31.8 -98.4 9 I -32.3 129.3 65.9 101.9 166.2 -69.0 -127.2 10 C -21.5 129.8 54.9 85.3 -155.1 -122.0 -143.5 11 G -78.3 -179.0 60.0 132.0 -174.9 -128.5 -110.2 12 C --- --- 171.7 160.3 174.1 -105.7 -93.2 Углы не так похожи как у предыдущих днк. Но может за счет этого структура будет более устойчивой. - Пользуясь программой pdb2img получила изображения моих структур
в виде стопочных моделей.
- Стопочные структуры для А-формы ДНК из файла gatc-a.pdb созданы командами
pdb2img -c gatc-a.pdb gatc-a rotate_mol -b gatc-a.pdb gatc-a2.pdb pdb2img -c gatc-a2.pdb gatc-a2
Вид сверху Вид сбоку 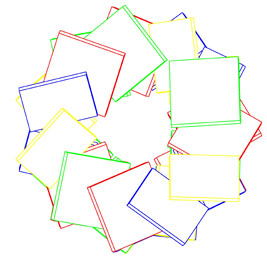
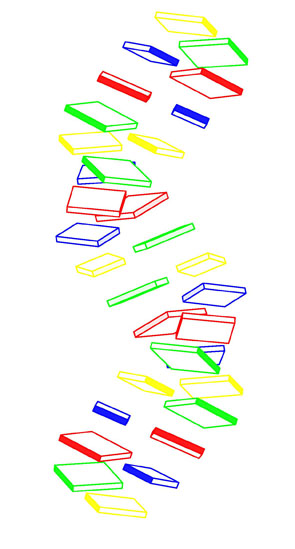
Видна характерная для А-формы ДНК полость внутри спирали.
На виде сбоку заметно, что азотистые основания наклонены. - Стопочные структуры для B-формы ДНК из файла gatc-b.pdb созданы командами
pdb2img -c gatc-b.pdb gatc-b rotate_mol -b gatc-b.pdb gatc-b2.pdb pdb2img -c gatc-b2.pdb gatc-b2
Вид сверху Вид сбоку 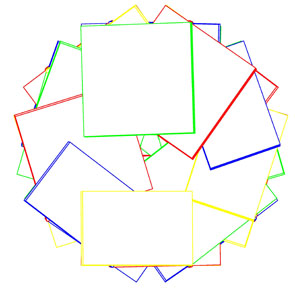
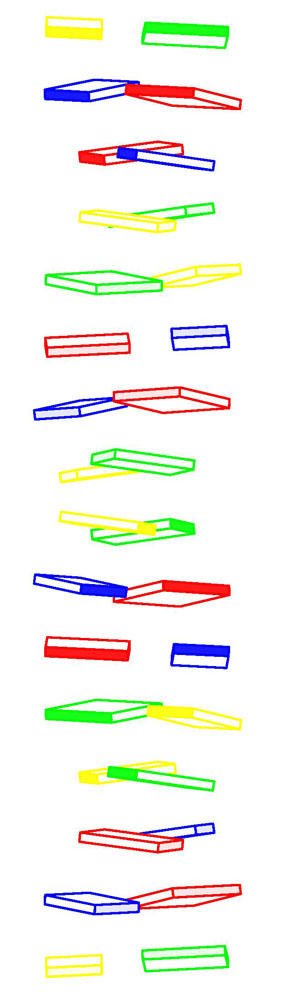
Внутри структуры отсутствует полость,
что говорит о том, что это В-форма ДНК. На виде сбоку заметно,
что азотистые основания находятся практически
перпендикулярно к оси спирали. - Стопочные структуры для РНК из файла dna29.pdb созданы командми
pdb2img -c dna29.pdb dna29 rotate_mol -b dna29.pdb dna292.pdb pdb2img -c dna292.pdb dna292
Вид сверху Вид сбоку 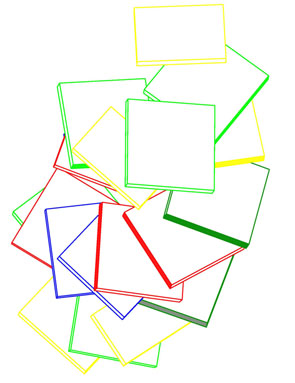
Внутри структуры не видно полости, что говорит о том, что это скорее В-форма. На виде сбоку заметно, что основания практически перпендикулярны оси спирали, что говорит в пользу В-формы.
- Стопочные структуры для А-формы ДНК из файла gatc-a.pdb созданы командами