Моделирование и реконструкция эволюции гена
1.
Визуализация дерева, заданного скобочной формулой(см. ниже). Длины ветвей пропорциональны эволюционным расстояниям.
Дерево является бинарным, не ультраметрическим (так как растояния от корня
до листьев не равные).
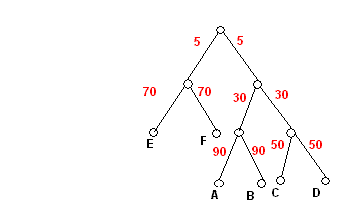
(((А:90,B:90):30,(C:50,D:50):30):5,(E:70,F:70):5);
Описание ветвей дерева как разбиения множества листьев:
ABCDEF
..****
**..**
....**
Для получения искуственных мутантных последовательностей, соответствующих листьям и
узлам дерева,приведена длина гена и формула для пересчёта расстояний в число мутаций
в моем гене и текст скрипта, которым получаются эти мутантные последовательности.
Длина гена 1323
Формула для пересчета расстояний(значения округлены,берем целое число мутаций):
5- это 1323*0.05= 66 мутаций
30- это 1323*0.3=396 мутаций
90-это 1323*0.9=1190 мутаций
50-это 1323*0.5=661 мутаций
70-это 1323*0.7=926 мутаций
(Скрипт):
msbar 2tptn.fasta abcd.fasta -point 4 -count 66 -auto
msbar abcd.fasta ab.fasta -point 4 -count 396 -auto
msbar abcd.fasta cd.fasta -point 4 -count 396 -auto
msbar ab.fasta a.fasta -point 4 -count 1190 -auto
msbar ab.fasta b.fasta -point 4 -count 1190 -auto
msbar cd.fasta c.fasta -point 4 -count 661 -auto
msbar cd.fasta d.fasta -point 4 -count 661 -auto
msbar 2tptn.fasta ef.fasta -point 4 -count 66 -auto
msbar ef.fasta e.fasta -point 4 -count 926 -auto
msbar ef.fasta f.fasta -point 4 -count 926 -auto
2.С помощью программ я получила три реконструкции моего дерева:
С помошью алгоритма максимального правдоподобия(файл):
Команда: fdnaml ali.fasta -ttratio 1 -auto
ABCDEF
****..
**..**
**....
С помощью алгоритма UPGMA(файл):
Команда для подсчета попарных расстояний между последовательностями fdnadist ali.fasta -ttratio 1 -auto
Для реконструкции: fneighbor ali.fdnadist -treetype u -auto
ABCDEF
**..**
**..*.
**....
С помощью алгоритма Neighbor-joining(файл):
Команда для подсчета попарных расстояний между последовательностями fdnadist ali.fasta -ttratio 1 -auto
Для реконструкции: fneighbor ali.fdnadist -treetype u -auto
ABCDEF
****..
**..**
**....
Итак, для сравнения деревьев, выпишем все возможные ветви(которые встретились)
ABCDEF Исходное Макс.правдоп UPGMA Neighbor
..**** + + + +
**..** + + + +
....** + + - +
**..*. - - + -
Следует отметить, что алгоритм UPGMA,который не полностью справился с задачей, в отличие от NJ и максимального правдоподобия строит укорененное дерево и использует
алгоритм "молекулярных часов"
Максимальное правдоподобие:
+----------------------------------------------B
|
| +----------------------------------F
| +--4
| | +--------------------------------------E
1---------3
| | +----------------------D
| +------------2
| +-----------------------C
|
+----------------------------------------------A
UPGMA:
+------------------------------------------------A
!
--5 +-----------------------------------------------B
! !
! ! +----------------------C
+-4 +-------------1
! +-2 +----------------------D
! ! !
+---------3 +------------------------------------F
!
+-------------------------------------E
Neighbor-joining:
+-----------------------------------------------B
!
! +-----------------------C
! +-------------1
! ! +----------------------D
2----------3
! ! +---------------------------------------E
! +-4
! +----------------------------------F
!
+-------------------------------------------------A
Назад