Исследование структуры тРНК
- Краткое описание структуры в файле
1I9V.pdb
В файле приведены координаты атомов молекулы тРНК для фенилаланина в комплексе с неомицином.
Организм Saccharomyces cerevisiae (пекарские дрожжи). Кроме тРНК этом документе представлены координаты лигандов - иона магния и собственно неомицин (антибиотик).
Для исследования была выбрана цепь А (единственная), представляющая фенилаланин-тРНК со следующей последовательностью:
[1] 5' GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAYGAUCUGGAGGUCCUGUG5MUUCGAUCCACAGAAUUCGCACCA 3' [76]
,где 1 и 76 - номера первого и последнего нуклеотида, YG [37] и 5MU [54] - модифицированные нуклеотиды: вибутозин и 5-метилурацил, соответственно (выделены в последовательности подчеркиванием).
Модифицированные основания
YG - WYBUTOSINE:Систематическое название 1H-IMIDAZO(1,2-ALPHA)PURINE-7-BUTANOIC ACID,4,9-DIHYDRO-ALPHA-((METHOXYCARBONYL)AMINO)- 4,6-DIMETHYL-9-OXO-METHYL ESTER. Практически, это сильно модифицированный гуанозин. Это основание соединяется с рибозой посредством стандартной N9-C1' связи.
5MU: 5-метилурацил, или риботимидин (обозначение 5MU, позиция 54). Фактически, это основание тимидин, но поскольку тимидина в РНК обычно не бывает, принято считать, что это модифицированный урацил. Или риботимидин, что значит - тиминовое основание, но на рибозе.
На 3'-конце есть триплет CCA, к которому присоединяется аминокислота. Этот триплет есть и в PDB файле.
- Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные
водородные связи
между азотистыми основаниями. Использовали команду
find_pair -t 1I9V.pdb pairs.inp
Получили файл pairs.inp В соответствии с полученными данными
акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 72-66.
Т-стебель - 49-53 и 65-61
D-стебель - 10-13 и 25-22
антикодоновый стебель - 40-44 и 30-26
Структуру стеблевых дуплексов поддерживают 20 канонических и 1 неканоническая пара оснований.
Неканоническая пара :
Контактирующие пары: a44.n6 - g26.o6 и a44.n1 - g26.n1 .Рис.1. Вторичная структура фенилаланин-тРНК
Скрипт для получения изображения select all wireframe off backbone 50 color black background white select 1-7 or 66-72 color red backbone 100 select 49-53 or 61-65 backbone 100 color green select 10-13 or 22-25 backbone 100 color blue select 26-30 or 40-44 color orange backbone 100
Вариабельная петля отсутствует, в Т-петле есть 5-метилурацил, или риботимидин (обозначение 5MU, позиция 54).
Дигидроуридины в D-петле отсутствуют
Антикодон в антикодоновой петле - A-A-YG (35-37 нуклеотиды)
YG - WYBUTOSINE (описание см. в пункте1).
- Исследование третичной структуры
1)Для изучения стекинг взаимодействия используем файлы, полученные с помошью команды
find pair -t 1I9V.pdb stdout | analyze
В файле 1I9V.out Находим информацию о перекрытиях оснований (поиск (F7) по слову overlap). Выбираем перекрытия .Им соответствуют структуры 11 и 12 из файла stacking.pdb.step i1-i2 i1-j2 j1-i2 j1-j2 sum 7 UC/GA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 2.93( 1.39) 2.93( 1.39) 20 AG/CG 0.00( 0.00) 0.00( 0.00) 0.71( 0.00) 0.59( 0.08) 1.30( 0.08)Чтобы получить картинки сначала вырезаем нужные фрагменты
ex_str -7 stacking.pdb step7.pdb
ex_str -12 stacking.pdb step20.pdb
Потом делаем из них картинки
stack2img -cdolt step7.pdb step10.ps
stack2img -cdolt step20.pdb step12.ps
Между концом акцепторного стебля
и началом Т-тебля
U 7 : A 66 / C 49: G 65
Площадь перекрытия 2.93( 1.39)Между антикодоновым и D-стеблем
A 44: G 26 / G 10 : C 25
Площадь перекрытия 1.30( 0.08)Выводы: стекинг-взаимодействие между парами оснований на "границе" антикодонового и D-стеблей очень мало, в этом месте общая для стеблей цепь изгибается и комплиментарные ей основания (44 и 10) сильно разобщены в пространстве. А аналогичное взаимодействие между основаниями акцепторного и Т стеблей более существенно и может иметь влияние на пространственную структуру молекулы.
2)Дополнительные водородные связи между основаниями D- и T-петель.
Взаимное расположение этих контактов в пространстве
Водородные связи образуют основания С56 и G19 D и Т петель, а также основание G15 D-петли с основанием C48 в цепи между Т-стеблем и акцепторным стеблем. На рисунке красным цветом показаны нуклеотиды D-шпильки (петля и стебель), зеленым - Т-шпильки (петля и стебель)
Реальная и предсказанная вторичная структура тРНК из файла 1I9V.pdb
Участок структуры
Позиции в структуре
(по результатам find_pair)Результаты предсказания
с помощью einvertedРезультаты предсказания
по алгоритму ЗукераАкцепторный стебель 5'1-7 3'
5' 66-72 3'
Всего 7 парни оной пары не предсказано из этого промежутка 6 из 7 пар D-стебель 10-13
22-25
4 парынуклеотиды 22-25 "спарены" с другой цепью (10-13) все 4 правильные T-стебель 49-53
65-61
5 парвсе 5 пар правильные все 5 пар правильные Антикодоновый стебель 40-44
30-26
5 парвсе 5 пар предсказаны, есть 1 лишняя стебель на 1 пару сдвинут (то есть одной не хватает, и одна лишняя) Общее число канонических пар нуклеотидов 21 всего 15, правильно 11 всего 20 пар,все правильно Изображение, полученное с помощью mfold
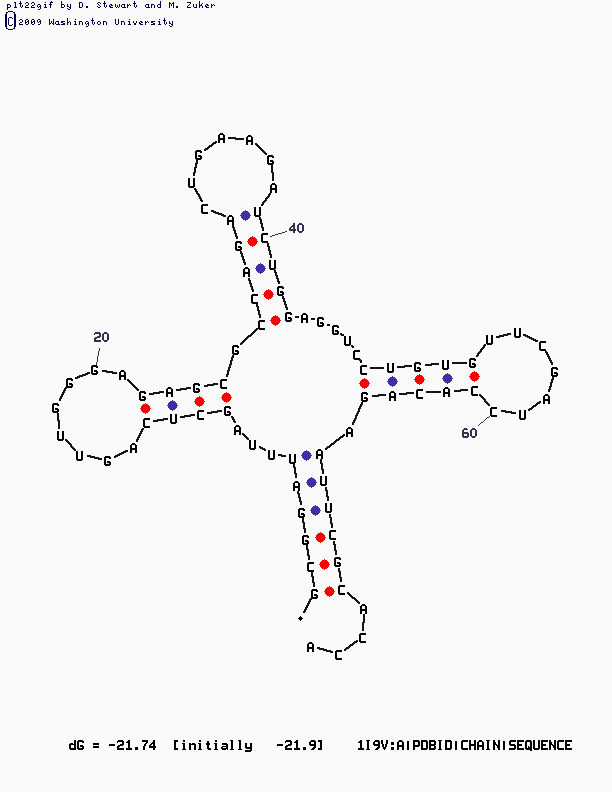
Использовались значения параметров: -для программы einverted уменьшали параметр threshold до 10, пока не появился осмысленный результат, другие параметры оставили по умолчанию. -для программы mfolt взяли P=15. Одна из полученных картинок достаточно хорошо вписывается в каноническую структуру "клеверного листа". Если увеличить Р до 20ти, вариантов значительно больше, но новых "клеверных листов" среди них мало (только один и очень "кривой").