С помощью программы emma было построено множественное выравнивание последовательностей выборки. Затем с помощью программы eprotdist была построена матрица попарных расстояний между белковыми последовательностями выборки.
Было реконструировано филогенетическое дерево по алгоритму ближайших соседей (Neighbour-Joining) с помощью программы eneighbor, принимающей на вход выходной файл, содержащий матрицу попарных расстояний, программы eprotdist.
Дерево визуализировано программой GeneMaster. Ветви, соответствующие внешней группе, сделаны потолще, а соответствующие подписи вершин подчеркнуты.
Фаил с деревом можно видеть здесь
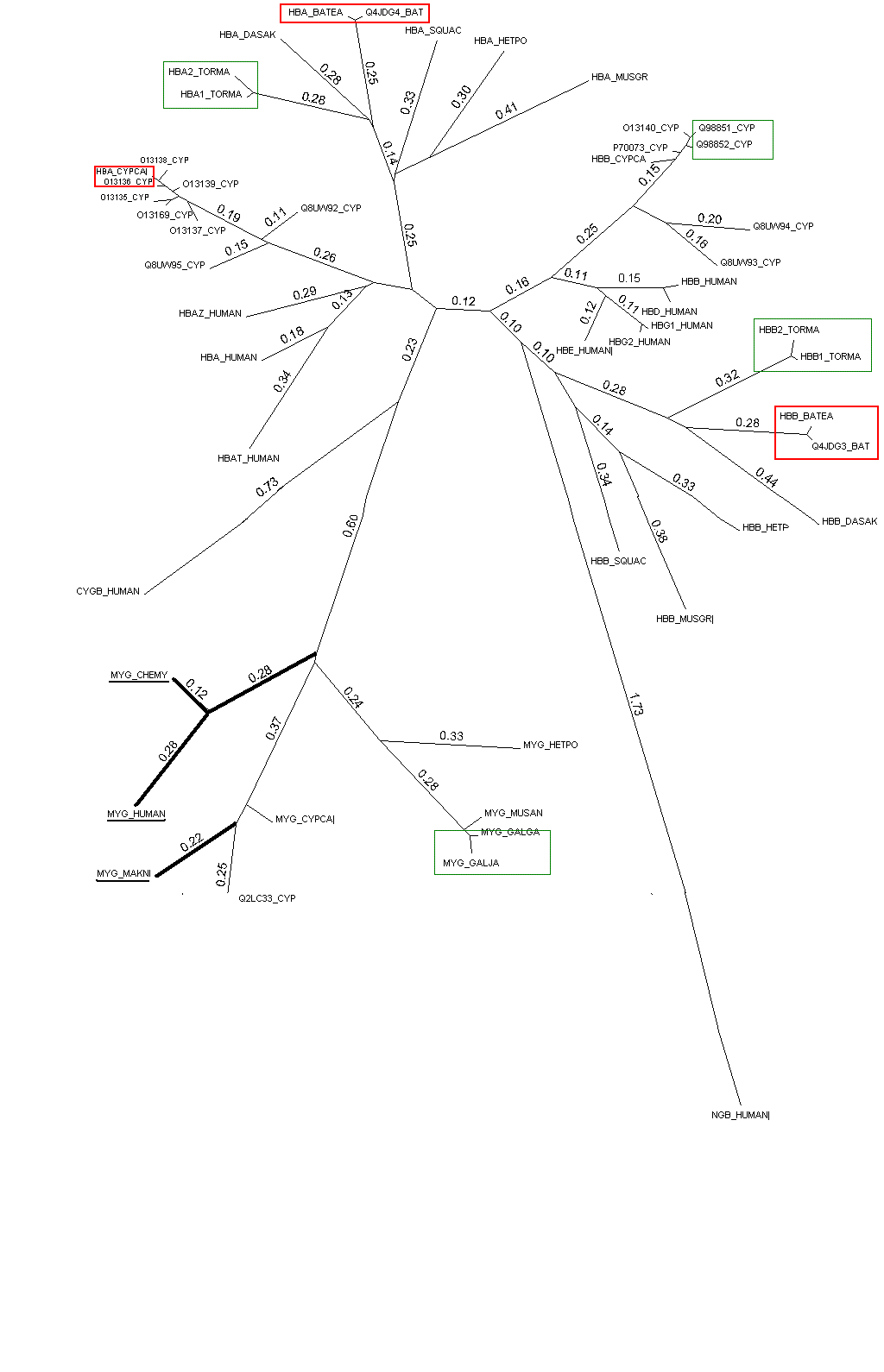
Последовательности, возникшие из одного общего предшественника в результате дупликации одного гена в одном организме называются паралогами. Как правило, они имеют разные функции. На дереве изображено три группы паралогов (зеленая рамочка).
Ортологи - белки, имеющие общее происхождение и, как правило, выполняющие схожие функции. На дереве выделено три группы ортологичных белков (красная рамочка).
© Низамутдинов Игорь, 2004