A- и В- формы ДНК. Структура РНК
Главная
Задание 1
Построила модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
С помощью программы Рutty, используя протокол ssh, подсоединилась к серверу kodomo.cmm.msu.ru. Перешла в директорию Term3/Practice2.
Ввела следующие команды, что бы указать путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
С помощью программы fiber пакета 3DNA построила A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc".
Структуру дуплекса в А-форме сохранила в файле gatc-a.pdb, структуру дуплекса в В-форме в файле gatc-b.pdb, структуру дуплекса в Z-форме в файле gatc-z.pdb.
Для этого использовала команды:
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Задание 2.
Упражнение 1
Научилась выделять разные атомы и химические группировки, используя предопределенные множества RasMol.
В этом упражнении используются файлы, созданные в Задание 1.
Для B-формы ДНК.
Сахарофосфатный остов ДНК
Все нуклеотиды
Все аденины
Атом N7 во всех гуанинах и только в первом по последовательности (синий)
Для B-формы ДНК.
Сахарофосфатный остов ДНК
Все нуклеотиды
Все аденины
Атом N7 во всех гуанинах и только в первом по последовательности (сине-зеленый)
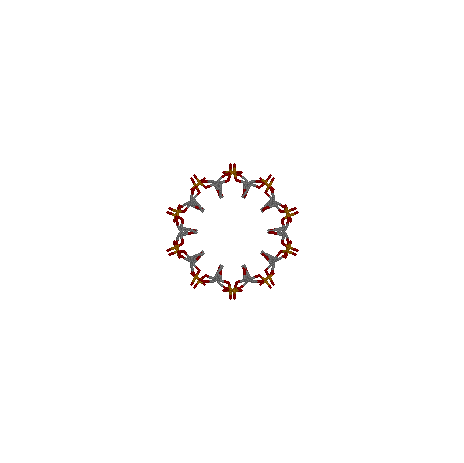
Для Z-формы ДНК.
Сахарофосфатный остов ДНК
Все нуклеотиды
Все аденины
Атом N7 во всех гуанинах и только в первом по последовательности (сине-зеленый)
Упражнение 2
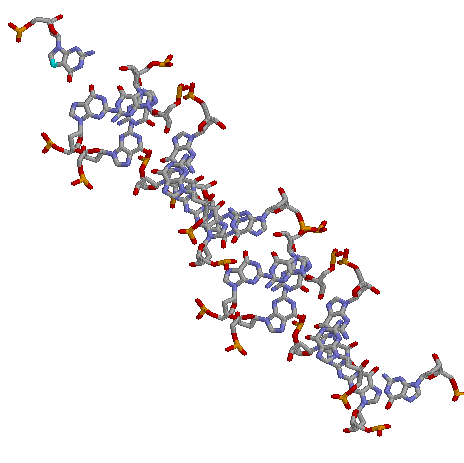
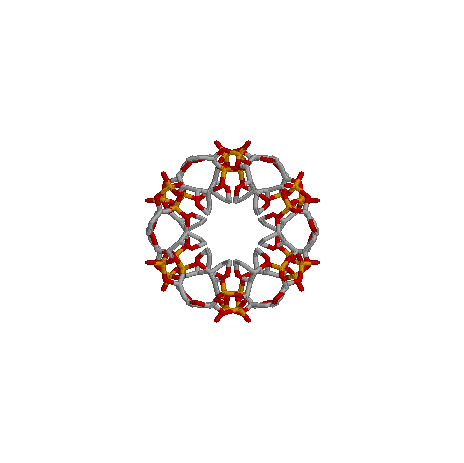
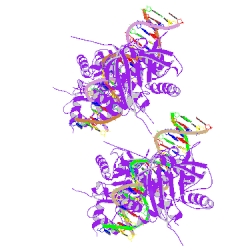
The crystal structure of yeast phenylalanine tRNA at 1.93 A resolution.
1ehz,
файл.
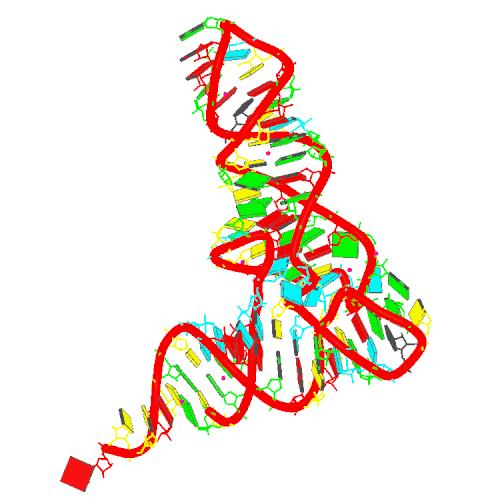
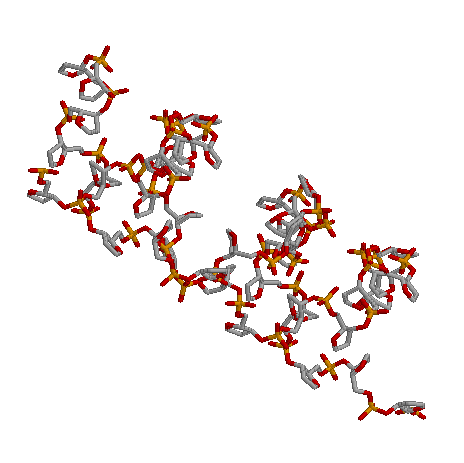
1d5y,
файл.
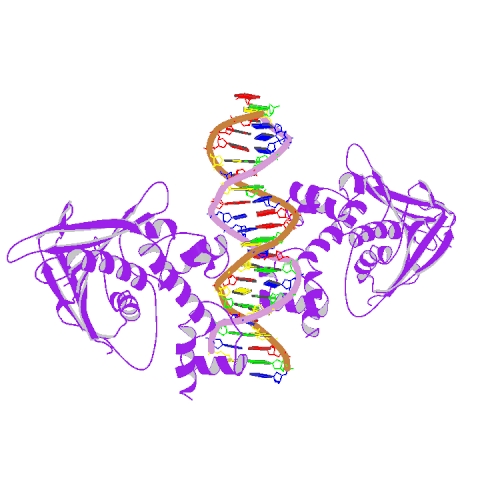
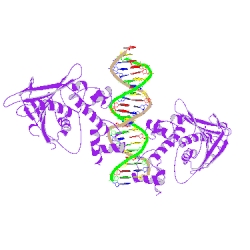
Crystal structure of the Escherichia coli Rob transcription factor in complex with DNA.
Упражнение 3
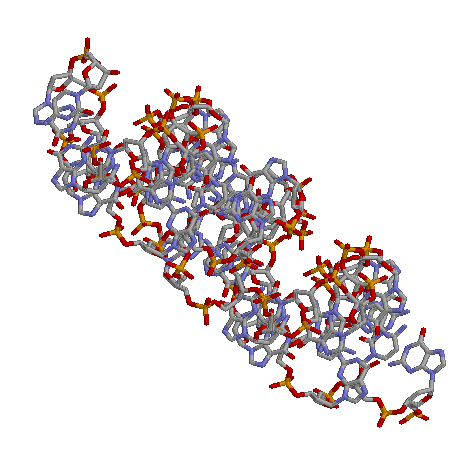
Проверка заданных структур ДНК и РНК на наличие разрывов.
В ДНК разрывов не обнаружено.
В тРНК они имеются.

ДНК
|
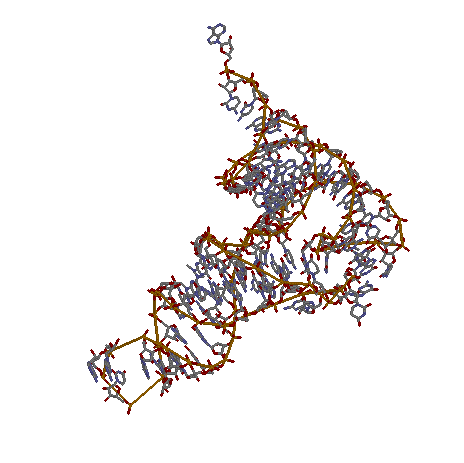
тРНК
|
Задание 3.
Упражнение 1
Открыла в RasMol файл gatc-b.pdb.
Рассмотрела структуру и визуально определила большую и малую бороздку.
Выбрала аденин 22 (А22).
Определила, какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой.
С помощью ChemSketch получила изображение основания, выделила красным цветом атомы, смотрящие в сторону большой бороздки, синим в сторону малой.
В сторону большой бороздки обращены атомы: N1,N3,N6,N7,C2,C5,C6.
В сторону малой бороздки обращены атомы: O1P, O2P, O3’, O4’, O5’, C1’, C2’,C3’,C5’.
Остальные атомы основания находятся между ними или нелья четко определить где.
Упражнение 2
Сравненние основны спиральных параметров разных форм ДНК.
|
А-форма |
B-форма |
Z-форма |
| Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
| Шаг спирали (A) |
28,03 |
33,75 |
43,5 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки |
16,81 |
17,91 |
18,3 |
| Ширина малой бороздки |
7,98 |
11,69 |
9,86 |
Упражнение 3
Сравнение торсионных углов в структурах А- и В-форм.
| угол |
α |
β |
γ |
δ |
ε |
ξ |
χ |
| A-форма (презентация) |
-62 |
173 |
52 |
88 или 3 |
178 |
-50 |
-160 |
| A-форма (RasMol) |
-51.69 |
174.81 |
41.73 |
79.07 |
-147.75 |
-75.12 |
-157.20 |
| B-форма (презентация) |
-63 |
171 |
54 |
123 или 131 |
155 |
-90 |
-117 |
| B-форма (RasMol) |
-29.90 |
136.31 |
31.19 |
143.33 |
-140.74 |
-160.49 |
-98.02 |
Задание 4.
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Упражнение 1
Для файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb была выполнена команда:
find_pair -t gatc-x.pdb stdout | analyze
В результате для каждой структуры создан ряд файлов с описанием разных её параметров. В файлах
gatc-a.out, gatc-b.out,gatc-z.out можно найти значения торсионных углов.
В наиболшей степени отлчаюся значения углов:в A- и B-формах - δ и χ
в A- и Z-формах - α
в B- и Z-формах - α, ξ и χ.
Определение значения торсионных углов в заданной структуре тРНК (1ehz)
Использовала программу remediator для перевода файла 1EHZ.pdb в старый формат:
remediator --old "1EHZ.pdb" > "1EHZ_old.pdb"
Затем применила команду:
find_pair -t 1EHZ_old.pdb stdout | analyze
Значения торсионных углов в структуре тРНК (1EHZ_old.out):
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- -128.1 67.8 82.9 -155.6 -68.6 -167.8
2 C -67.4 -178.4 53.8 83.4 -145.1 -76.8 -163.8
3 G -74.5 169.7 59.5 80.7 -148.3 -80.0 -161.9
4 G -64.4 162.2 60.7 82.2 -157.4 -68.7 -168.7
5 A -74.7 -176.5 53.4 84.9 -137.5 -81.7 -162.9
6 U -48.8 157.6 55.3 81.3 -151.0 -77.0 -160.0
7 U -59.5 -178.7 62.5 137.3 --- --- -133.1
8 c --- 168.5 42.2 84.3 -145.0 -82.1 -173.6
9 U -51.7 177.2 42.1 80.4 -150.6 -67.8 -165.3
10 G -63.9 176.8 52.8 79.4 -150.4 -71.3 -156.6
11 U -64.7 173.6 48.5 80.3 -156.5 -69.4 -164.0
12 G -56.9 171.5 56.2 83.9 -159.4 -64.9 -169.2
13 u -79.7 -172.8 57.7 77.6 -128.6 -70.7 -161.5
14 P -49.7 168.8 44.1 76.6 --- --- -147.0
15 A --- 165.7 51.3 72.2 --- --- -158.4
16 A --- -180.0 46.9 82.5 -136.8 -76.4 -169.4
17 P -47.7 160.4 53.3 79.3 -140.1 -68.6 -165.6
18 c -67.4 172.0 56.2 83.2 -154.2 -74.9 -162.6
19 U -68.2 -179.4 52.4 78.9 -137.3 -84.7 -169.0
20 G -47.9 158.7 55.6 79.8 -160.3 -70.3 -169.0
21 G -67.0 -178.3 55.6 81.6 -154.9 -76.4 -160.2
22 A -59.7 162.1 60.0 85.3 --- --- -159.4
23 g --- 147.2 60.1 89.3 -126.2 -88.7 169.6
24 C -56.1 167.9 48.2 87.2 -150.5 -69.9 -160.9
25 U -67.8 172.9 51.8 80.7 -158.5 -65.2 -158.3
26 C 166.6 -169.9 178.6 82.5 -153.1 -97.4 -168.3
27 A 83.4 -158.3 -114.6 92.0 -125.5 -57.3 -170.7
28 G -55.1 162.5 51.9 79.8 -136.3 -143.9 -164.5
29 u -6.1 91.2 76.8 96.8 --- --- -85.8
30 G --- -178.9 53.8 153.8 --- --- -80.3
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C -73.2 176.2 62.1 83.0 --- --- -161.6
2 G -78.4 173.6 60.3 80.3 -149.6 -68.4 -162.8
3 C -61.7 164.6 53.1 79.0 -158.5 -64.5 -152.0
4 U -63.8 168.1 55.1 79.1 -155.4 -85.6 -161.4
5 U -59.8 175.3 47.3 82.2 -152.9 -65.4 -160.1
6 A -62.0 164.1 54.2 83.2 -152.2 -78.3 -162.8
7 A -57.9 178.5 52.0 81.7 -151.0 -73.5 -164.9
8 G -44.0 164.2 49.9 79.8 -152.0 -73.3 -172.8
9 A -66.9 180.0 44.1 75.8 -147.5 -76.5 -161.8
10 C -62.0 167.3 50.9 80.7 -152.3 -70.7 -152.6
11 A -60.1 179.6 46.9 80.5 -145.6 -74.1 -158.7
12 C --- 179.8 38.2 83.0 -152.3 -74.5 -166.7
13 a --- -146.1 71.8 156.7 --- --- -86.3
14 G --- -159.4 59.0 150.6 --- --- -99.5
15 U -67.7 -177.0 47.0 82.1 --- --- -148.2
16 c -52.7 161.4 49.3 80.1 -145.9 -71.2 -149.9
17 A -69.9 177.8 52.3 83.7 -137.0 -75.5 -156.7
18 G -54.0 165.9 56.9 83.6 -144.7 -62.3 -171.7
19 A -66.6 174.0 55.6 81.4 -155.5 -78.3 -165.9
20 C -72.4 178.3 49.3 80.1 -152.1 -67.0 -160.6
21 C -53.0 166.9 43.6 83.4 -148.5 -73.4 -168.2
22 g -53.8 170.8 47.7 86.0 -136.3 -76.9 -163.4
23 C -65.1 168.9 53.9 83.3 -145.1 -68.4 -160.3
24 G -68.8 178.2 46.8 83.6 -144.3 -72.8 -160.7
25 A -53.3 174.8 52.5 82.3 -155.3 -66.4 -158.0
26 G --- 153.5 179.3 82.0 -145.0 -80.4 -175.5
27 U --- -145.6 55.4 78.6 --- --- -161.5
28 C --- -174.3 161.5 145.6 --- --- -140.1
29 U --- -158.8 63.7 84.6 --- --- -165.6
30 C --- 171.8 53.3 83.4 --- --- -161.5
Данная структура больше всего похожа на А-форму ДНК.
Аналогично получила значения торсионных углов 1d5y ДНК(1D5Y_old.out):
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- -155.9 43.0 143.2 -172.8 -128.6 -106.1
2 A -51.0 170.9 47.1 145.1 179.5 -91.7 -107.2
3 C -56.7 177.3 47.1 136.1 -147.8 -150.9 -110.3
4 A -58.9 156.5 50.2 137.7 -166.0 -100.1 -124.3
5 G -56.2 171.6 39.2 135.7 -167.6 -152.1 -109.7
6 C -27.8 143.9 46.9 141.7 -161.1 -116.9 -115.5
7 A -70.3 176.4 49.9 143.2 -163.6 -117.7 -105.2
8 C -65.9 177.3 45.1 133.7 158.0 -91.9 -98.8
9 T -50.5 -159.0 41.7 139.1 -178.9 -94.0 -103.6
10 G -58.0 -178.9 41.0 140.0 -147.9 -155.6 -103.8
11 A -38.8 142.4 38.4 132.3 176.5 -96.0 -115.4
12 A -49.3 -163.8 33.0 139.3 171.4 -96.2 -100.3
13 T -49.6 -176.4 45.0 135.8 176.1 -109.7 -106.3
14 G -53.4 -178.3 44.0 141.4 170.9 -100.4 -111.4
15 T -62.2 -157.5 41.0 140.7 -160.9 -133.4 -101.0
16 C -42.8 149.5 44.8 134.6 178.5 -96.7 -110.8
17 A -62.9 -172.0 46.8 143.3 -143.3 -176.2 -96.0
18 A -48.3 131.5 53.1 143.7 175.5 -94.1 -117.5
19 A -55.1 -173.0 42.6 141.2 178.2 -104.1 -115.1
20 G -72.7 -155.2 42.2 147.1 --- --- -93.3
21 G --- -174.1 38.8 141.2 -158.7 -147.9 -105.8
22 A -42.8 155.0 40.6 137.2 177.8 -84.3 -115.3
23 C -53.0 -177.4 42.8 131.9 -168.2 -129.7 -115.6
24 A -47.0 173.0 42.7 138.0 -167.8 -108.4 -114.1
25 G -46.6 164.9 39.0 136.3 -170.3 -139.8 -107.6
26 C -40.1 161.9 44.0 137.4 -176.0 -107.3 -116.9
27 A -64.6 -167.3 45.7 147.0 -169.9 -125.6 -98.8
28 C -49.8 162.4 48.9 135.7 175.3 -104.7 -112.0
29 T -60.7 -167.3 48.9 140.5 178.5 -95.1 -94.5
30 G -55.7 -179.8 43.0 139.9 -150.1 -149.2 -102.2
31 A -45.4 149.5 40.8 134.6 160.6 -86.4 -110.4
32 A -69.5 -149.1 44.4 141.6 175.2 -108.3 -102.5
33 T -48.9 -179.2 45.1 139.9 -171.9 -116.0 -106.0
34 G -54.8 170.0 48.1 138.2 164.3 -93.4 -111.9
35 T -72.2 -146.1 41.3 142.3 -161.6 -172.8 -93.5
36 C 9.7 102.6 30.6 131.2 174.6 -95.9 -96.7
37 A -56.7 -173.9 43.3 140.9 -163.7 -174.2 -101.1
38 A -36.9 132.1 54.9 143.6 -179.8 -83.4 -132.0
39 A -72.8 -164.8 43.5 135.7 166.4 -99.3 -111.2
40 G -63.3 -157.8 47.9 153.0 --- --- -99.7
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C -82.5 -161.2 48.9 145.3 --- --- -102.5
2 T -51.6 158.6 59.2 137.0 176.1 -84.8 -125.5
3 G -78.1 -157.1 41.4 142.4 -172.3 -143.0 -99.6
4 T -34.9 169.1 44.9 140.6 173.2 -83.1 -118.9
5 C -9.1 132.2 30.1 136.1 175.8 -124.4 -106.6
6 G -39.4 157.4 39.5 135.0 -161.2 -153.7 -117.7
7 T -52.8 147.1 48.4 135.9 -165.9 -134.8 -90.3
8 G -74.7 -165.9 39.6 146.0 -150.7 -139.7 -88.7
9 A -46.2 144.1 53.6 144.1 -172.0 -90.2 -123.0
10 C -45.3 154.3 45.6 137.5 -155.7 -157.7 -102.8
11 T -60.2 -152.4 42.3 138.1 -164.5 -113.6 -102.2
12 T -65.1 -163.4 39.9 134.3 161.5 -91.9 -98.6
13 A -46.1 166.3 51.5 143.8 175.6 -90.0 -114.2
14 C -63.5 -168.2 39.0 142.5 -177.0 -142.4 -105.4
15 A -53.7 157.1 47.9 142.6 177.3 -83.3 -121.8
16 G -44.8 147.7 45.4 139.8 -155.5 -151.5 -109.6
17 T -69.3 -159.1 44.9 141.9 -161.3 -137.5 -93.3
18 T -68.9 -154.6 52.1 143.0 171.1 -87.2 -107.9
19 T -23.1 130.4 52.0 140.9 165.6 -101.3 -115.5
20 C --- -128.7 53.4 142.7 -172.1 -162.2 -91.3
21 C -73.3 177.8 48.0 144.8 --- --- -104.4
22 T -53.5 163.6 62.0 138.3 -163.0 -104.2 -116.6
23 G -75.3 -168.8 41.0 140.5 -177.4 -131.6 -101.7
24 T -31.2 132.9 50.9 134.4 -170.0 -95.4 -128.6
25 C -39.2 169.9 36.1 137.1 -158.8 -149.5 -111.1
26 G -40.3 -166.1 37.5 138.4 179.7 -110.1 -102.2
27 T -92.8 147.9 27.8 133.6 162.5 -99.4 -85.9
28 G -95.6 -164.2 47.0 149.2 -68.8 162.4 -75.5
29 A -36.1 137.9 49.9 140.7 -167.7 -84.4 -126.5
30 C -45.5 162.1 40.7 139.8 -158.7 -157.1 -103.4
31 T -46.9 -175.0 45.8 134.1 -165.3 -108.3 -114.3
32 T -67.3 -169.4 37.9 134.9 169.8 -110.9 -96.8
33 A -48.1 162.3 51.0 140.3 -175.7 -90.1 -117.5
34 C -78.0 -151.1 43.3 142.8 -175.2 -141.5 -97.7
35 A -50.4 158.5 46.5 145.8 165.2 -73.4 -112.6
36 G -46.9 146.8 49.2 139.7 -158.2 -146.5 -112.5
37 T -70.1 -150.2 48.9 147.4 -165.2 -142.5 -91.1
38 T -62.5 -153.7 43.5 139.5 160.7 -87.5 -104.2
39 T -47.7 143.9 55.4 136.2 167.4 -94.9 -118.1
40 C --- -134.3 48.2 142.9 -160.1 -144.7 -98.1
Упражнение 2
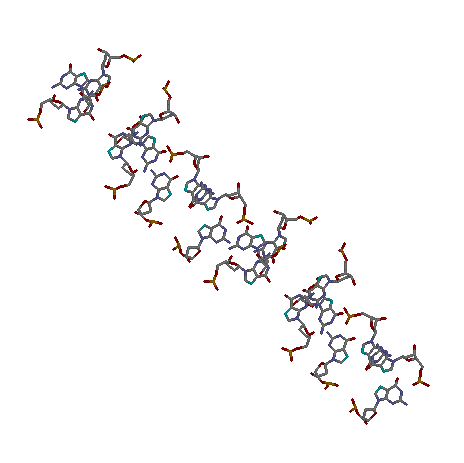
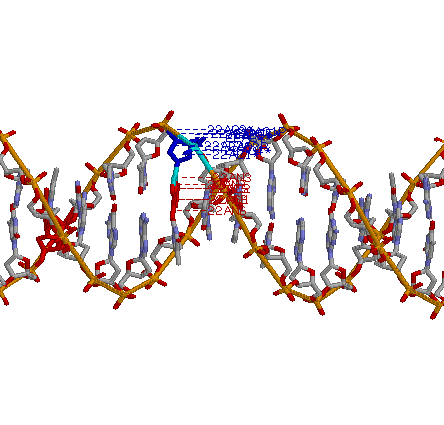
Определение структуры водородных связей
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями
(1EHZ_old.out)
Акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 72-66.
Т-стебель состоит из участка 49-53 и комплементарного ему участка 65-61.
D-стебель состоит из участка 10-13 и комплементарного ему участка 25-22.
Антикодоновый стебель состоит из участка 26-32 и комплементарного ему участка 44-38.
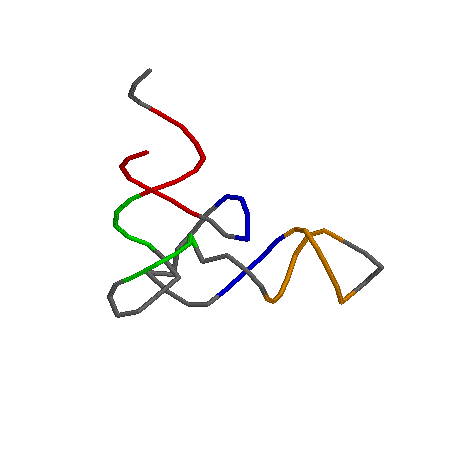
Акцепторный стебель выделен красным,
Т-стебель - зеленым,
D-стебель - синим,
антикодоновый - оранжевым.
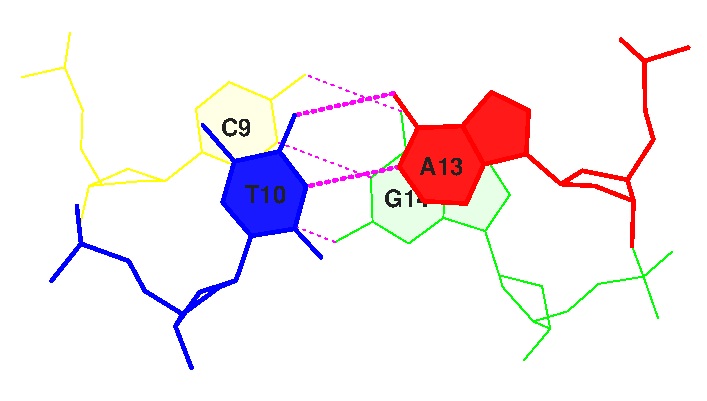
Неканонические пары нуклеотидов:
G4-U69;
A38-C32;
A44-G26.
Пары нуклеотидов 54-58 и 55-18 дополнительно стабилизируют Т-петлю.
Упражнение 3
Нахождение возможных стекинг-взаимодействий.
Данные о величине площади "перекрывании" 2-х последовательных пар азотистых оснований
(1EHZ_old.out):
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GC/GC 5.19( 2.33) 0.00( 0.00) 0.00( 0.00) 6.59( 3.41) 11.78( 5.74)
2 CG/CG 0.51( 0.00) 0.00( 0.00) 2.98( 0.46) 0.71( 0.00) 4.20( 0.46)
3 GG/UC 2.74( 1.28) 0.00( 0.00) 0.01( 0.00) 0.30( 0.00) 3.05( 1.28)
4 GA/UU 4.55( 2.72) 0.00( 0.00) 0.00( 0.00) 2.78( 0.92) 7.34( 3.64)
5 AU/AU 4.86( 3.47) 0.00( 0.00) 0.00( 0.00) 3.00( 1.72) 7.86( 5.18)
6 UU/AA 0.50( 0.00) 0.00( 0.00) 0.00( 0.00) 3.45( 3.11) 3.94( 3.11)
7 Uc/GA 0.98( 0.00) 0.00( 0.00) 0.00( 0.00) 3.08( 1.54) 4.06( 1.54)
8 cU/AG 0.00( 0.00) 0.00( 0.00) 0.40( 0.00) 3.43( 2.87) 3.83( 2.87)
9 UG/CA 0.31( 0.00) 0.00( 0.00) 2.45( 1.12) 0.22( 0.00) 2.99( 1.12)
10 GU/AC 5.87( 3.10) 0.00( 0.00) 0.00( 0.00) 4.01( 2.38) 9.88( 5.48)
11 UG/CA 0.00( 0.00) 0.00( 0.00) 4.02( 1.93) 0.00( 0.00) 4.02( 1.93)
12 Gu/aC 8.58( 3.94) 0.00( 0.00) 0.00( 0.00) 3.56( 0.68) 12.14( 4.63)
13 uP/Ga 6.08( 1.82) 0.00( 0.00) 0.00( 0.00) 8.21( 3.27) 14.29( 5.10)
14 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 AA/cU 2.73( 0.95) 0.00( 0.00) 0.00( 0.00) 4.92( 2.25) 7.66( 3.20)
16 AP/Ac 5.04( 3.17) 0.00( 0.00) 0.00( 0.00) 6.30( 3.92) 11.34( 7.08)
17 Pc/GA 3.17( 0.17) 0.00( 0.00) 0.00( 0.00) 6.10( 4.17) 9.28( 4.34)
18 cU/AG 0.00( 0.00) 0.00( 0.00) 0.70( 0.00) 1.74( 1.71) 2.44( 1.71)
19 UG/CA 0.00( 0.00) 0.00( 0.00) 2.99( 1.72) 0.00( 0.00) 2.99( 1.72)
20 GG/CC 2.53( 1.04) 0.00( 0.00) 0.27( 0.00) 0.15( 0.00) 2.94( 1.04)
21 GA/gC 3.12( 0.91) 0.00( 0.00) 0.00( 0.00) 4.81( 2.13) 7.93( 3.04)
22 Ag/Cg 0.00( 0.00) 0.00( 0.00) 1.02( 0.00) 0.40( 0.07) 1.42( 0.07)
23 gC/GC 3.53( 0.73) 0.00( 0.00) 0.00( 0.00) 7.32( 4.35) 10.85( 5.08)
24 CU/AG 1.03( 0.01) 0.00( 0.00) 0.00( 0.00) 2.64( 2.33) 3.67( 2.35)
25 UC/GA 1.53( 0.43) 0.00( 0.00) 0.00( 0.00) 1.09( 0.09) 2.62( 0.52)
26 CA/UG 0.00( 0.00) 1.92( 0.00) 2.31( 0.19) 0.00( 0.00) 4.23( 0.19)
27 AG/CU 2.06( 0.54) 0.00( 0.00) 0.25( 0.00) 0.00( 0.00) 2.31( 0.54)
28 Gu/UC 3.09( 1.19) 1.91( 0.00) 0.00( 0.00) 0.00( 0.00) 5.00( 1.19)
29 uG/CU 0.00( 0.00) 0.00( 0.00) 1.39( 0.19) 0.16( 0.00) 1.55( 0.19)
По-моему, у 12-ой пары наибольшие значения площади перекрывания(13-я,наверное, не подходит).
 ________________
________________
Задание 2
Упражнение 1
Предсказание вторичной структуры заданной тРНК
При параметрах:
Minimum score threshold [50]: 10
Match score [3]: 10
Нашлось 2 стебля:акцепторный и антикодоновый. Определились также как в find_pair.
При параметрах:
Gap penalty: 15
Minimum score threshold: 25
Match score: 10
Mismatch score: -15
Нашелся T-стебель, полностью совпадает.
| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания
с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель |
5'-1-7-3'
5'-66-72-3'
Всего 7 пар (6 канонич.) |
все 6
|
все 6 канонич. |
| D-стебель |
5'-10-13-3'
5'-22-25-3'
Всего 4 пары
(все канонич.) |
Не найдено |
все 4 |
| T-стебель |
5'-49-53-3'
5'-61-65-3'
Всего 5 (все канонич.) |
все 5 |
Все 5 |
| Антикодоновый стебель |
5'-38-44-3'
5'-26-33-3'
8 пар (5 канонических)
|
все 6
|
4 канонич. +1 |
| Общее число канонических пар нуклеотидов |
20 |
17 |
19 |
Упражнение 2
Предсказание вторичной структуры тРНК по алгоритму Зукера.
Программа mfold из пакета EMBOSS реализует алгоритм Зукера.
Постаралась подобрать параметры для получения предсказания, наиболее близкого к реальной структуре.
Результаты внесла в таблицу.
Картинка с лучшим предсказанием. Было выдано при параметрах по умолчанию(p=5).
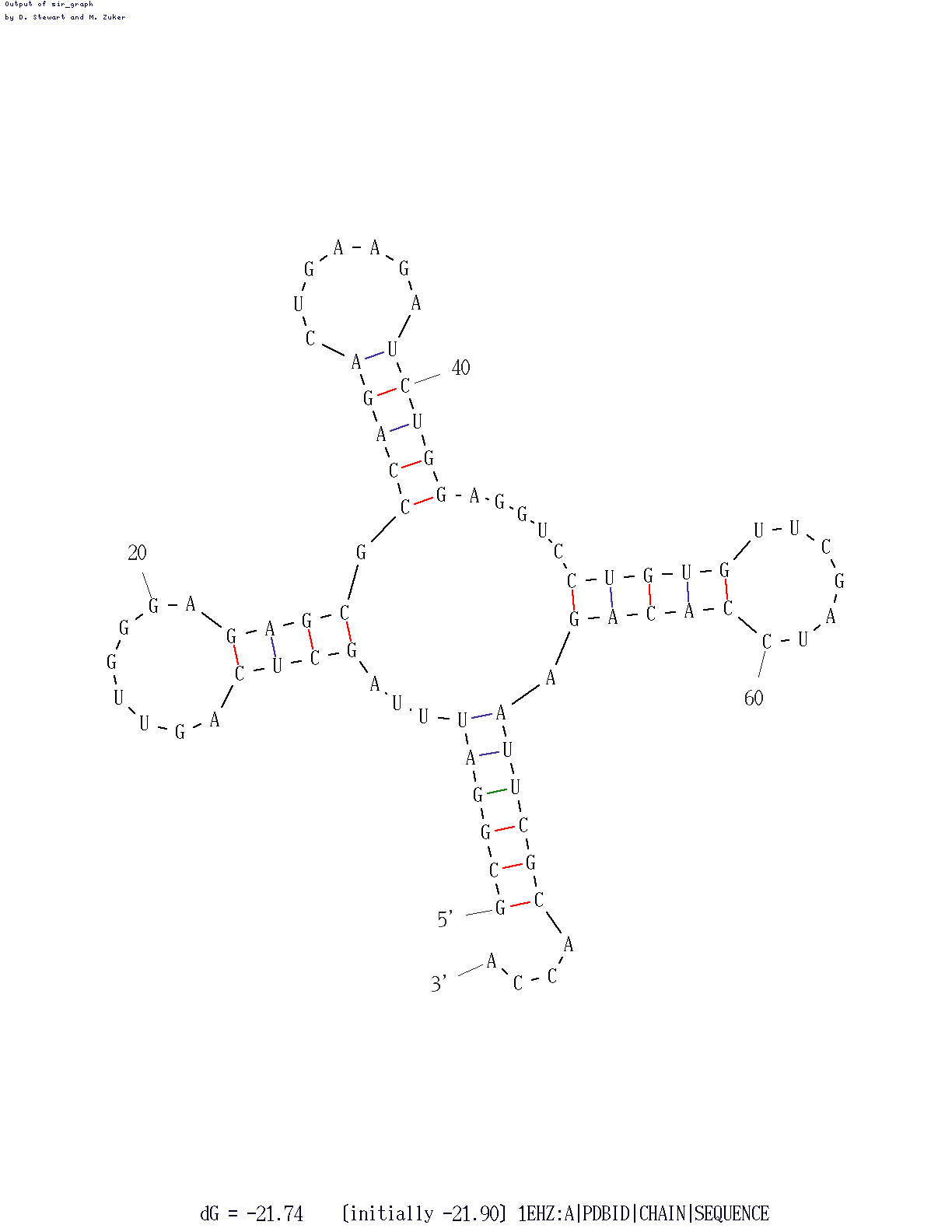
©Eliseeva Julia













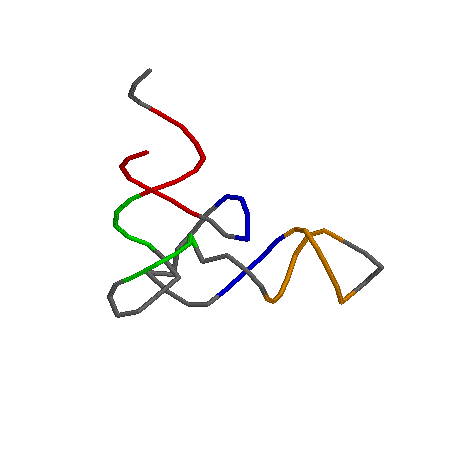
 ________________
________________
