Эволюционные домены(практикум 10)
Главная
1. Построить филогенетическое дерево по выравниванию представителей домена, полученному в практикуме 9.
Закодировала доменные архитектуры "1"(Amidohydro_3, D-aminoacyl_C) и "2"(Amidohydro_5, D-aminoacyl_C)(1,2 - просто порядок, т.к. обе архитектуры двухдоменные).
Так как у меня всё получилось бактериальное, буду использовать подтаксоны:
P - Proteobacteria, A - Actinobacteria, B - Bacteroidetes, F - Firmicutes.
Соответственно получила выравнивание:
dom2.fasta
Открыла его в Mega.
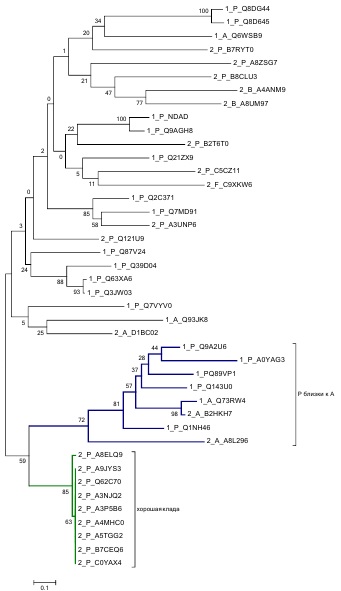
Использовала методы "Neighbor-Joining"(дерево слева) и "UPGMA"(справа).

Файлы со скобочной структурой:
Neighbor-Joining,
UPGMA.
Видимо, в эволюции происходила замена одного домена на другой, поэтому получилось такое несколько рандомное дерево.
Хотя и есть достоверные части. С помощью метода "UPGMA" дерево еще хуже(как и должно быть).
©Eliseeva Julia

