Укорененил в среднюю точку ("Midpoint root the tree").
Изображение укоренённого дерева, сделанное программой MEGA и неукорененное правильное дерево.
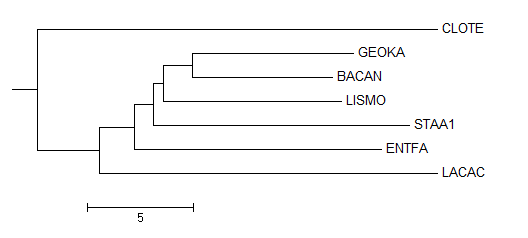
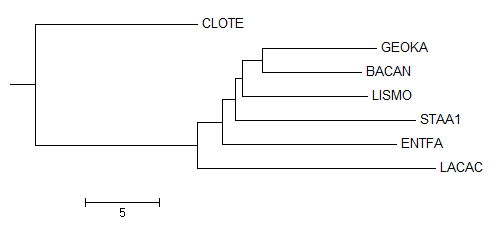
Укоренение произошло правильно. Совпадает с правильным деревом,но в тот раз укоренение было верно случайно. Так как без использования переукоренения, MEGA сама укореняет открываемое newick-дерево после Neighbour Joining в случайную точку.
2.Использование внешней группы
Деревья, построенные методом максимальной экономии ("Maximum parsimony") невозможно укоренить в среднюю точку,т.к.метод максимальной экономии (Maximum parsimony) не реконструирует длины ветвей.
Однако можно воспользоваться укоренением с помощью внешней группы.
Реконструирование методом максимальной экономии укоренённое дерево отобранных мною бактерий, используя в качестве внешней группы белок того же семейства из кишечной палочки (Escherichia coli, ECOLI).
Получила из Swiss-Prot последовательность Ecoli(if2_ecoli):
seqret sw:if2_ecoli
Записала результаты в файл со всеми бактериями(all.fasta).
Создала выравнивание отобранных белков:
воспользовалась программой muscle на kodomo( muscle -in all.fasta -out all_aligned.fasta)
Результат импортировала в программу MEGA.
Далее в меню Subtree выбрала Root и указала в качестве корня ветвь, ведущую к ECOLI.
Чтобы получить изображение укоренённого дерева без ECOLI, воспользовалась кнопкой "Show Subtree Separately" (изображение голубой лупы на фоне дерева на левой панели окна MEGA).
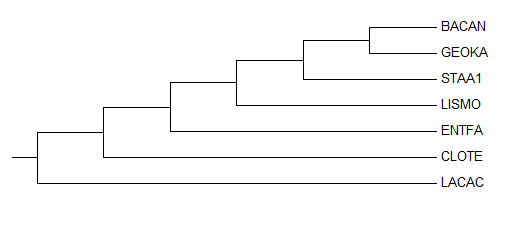

{BACAN,GEOKA,STAA1} против всех неправильно.
{BACAN,GEOKA,STAA1,LISMO} против всех правильно.
{BACAN,GEOKA,STAA1,LISMO, ENTFA} против всех правильно.
{CLOTE,LACAC} против всех неправильно.
3.Бутстрэп
Провела бутстрэп-анализ филогении своих белков, используя один из методов, доступных из прграммы MEGA.
Для этого:Analysis - Phylogeny - Construct/Test Neighbor-Joining tree.
Test of Phylogeny:Bootstrap method.
Указала число реплик, равное 100.
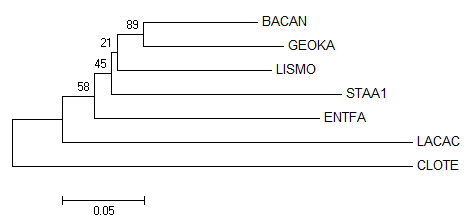
Все нетривиальные ветви совпадают с правильным деревом.
Высокая поддержка у ветвей {BACAN,GEOKA}vs{LISMO,STAA1,ENTFA,LACAC,CLOTE}(89). У остальных довольно низкая.
©Eliseeva Julia