Реконструкция деревьев по нуклеотидным последовательностям.
Анализ деревьев, содержащих паралоги.
Главная
1.Построение дерева по нуклеотидным последовательностям
1)Добыла последовательности 16S рибосомальной РНК каждой из бактерий.
В Swiss-Prot нашла ссылку на запись EMBL, описывающую полный геном.
А в ней — соответствующую "особенность" (FT), она имеет ключ (FTkey) "rRNA" и в описании, как правило: /note="16S rRNA" (иногда может быть упоминание 16S или малой рибосомной субъединицы в другой форме).
Вырезала нужный участок из записи EMBL с AC с помощью команды seqret
| Название | Мнемоника | EMBL AC | координаты | цепь |
| Bacillus anthracis | BACAN | AE016879 | 82451-83957 | прямая |
| Clostridium tetani | CLOTE | AE015927 | 8715-10223 | обратная |
| Enterococcus faecalis | ENTFA | AE016830 | 248466-249987 | прямая |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421-11973 | прямая |
| Lactococcus acidophilus | LACAC | CP000033 | 59255 - 60826 | прямая |
| Listeria monocytogenes | LISMO | AL591974 | 37466-39020 | прямая |
| Staphylococcus aureus | STAA1 | BX571856 | 559978-561532 | прямая |
2)Положила все последовательности в единый файл r16s.fasta
Воспользовалась программой muscle на kodomo:
muscle -in r16s.fasta -out r16s_aligned.fasta
r16s_aligned.fasta
3)Файл с выравниванием импортировала в программу MEGA(как выравнивание нуклеотидных последовательностей), было построено дерево методом Neighbor-Joining.
Полученное дерево
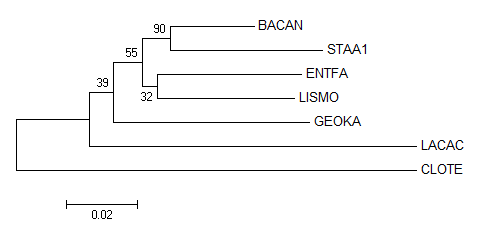
Исходное правильное дерево
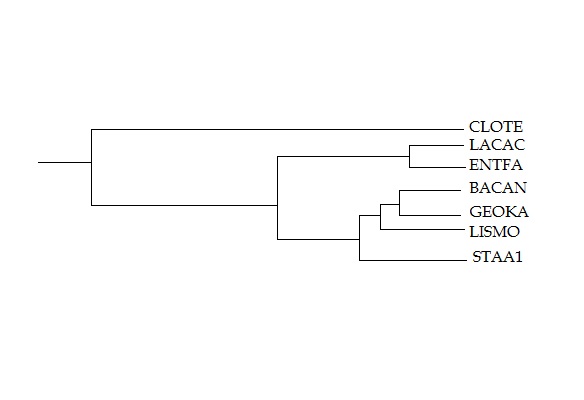
Видно,что они разные.
Построение и анализ дерева, содержащего паралоги
Нашла в своих бактериях достоверные гомологи белка CLPX_BACSU.
Чтобы найти гомологов в заданных организмах, воспользовалась файлом proteo.fasta на диске P, там лежат записи банка UNIPROT, относящиеся к бактериям, перечисленным в таблице к заданию 1.
Провела поиск программой blastp гомологов (с разумным порогом на E-value, скажем, 0,001) и отобрала по мнемонике видов только те находки, которые относятся к отобранным мною бактериям.
Сначала проиндексировала файл:
makeblastdb -in proteo.fasta -out prot -dbtype prot
Затем использовала blastp, выравнивая белки с заготовленной заранее fasta белка CLPX_BACSU
blastp -query clpx_bacsu.fasta -db prot -out prot_clpx.txt -evalue 0.001
Получила набор белков prot_clpx.txt
И создала файл, содержащий только те находки, которые относятся к отобранным мною бактериям.
clpx_homologs.fasta
Полученный файл с выравниванием импортировала в программу MEGA(использовала Neighbor-Joining).
Полученное дерево
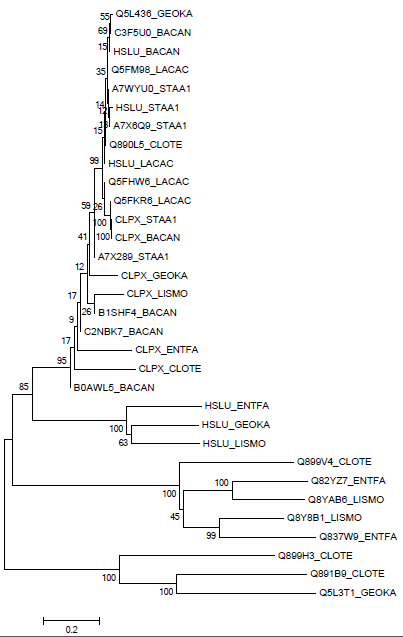
Два гомологичных белка будем называть ортологами, если они а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма будем называть паралогами.
Ортологи: HSLU_GEOKA и HSLU_LISMO, CLPX_STAA1 и CLPX_BACAN.
Некоторые паралоги:Q82YZ7_ETFA и Q837W9_ENTFA, Q899H3_CLOTE и Q891B9_CLOTE, Q5FHW6_LACAC и Q5FKR6_LACAC.
©Eliseeva Julia


