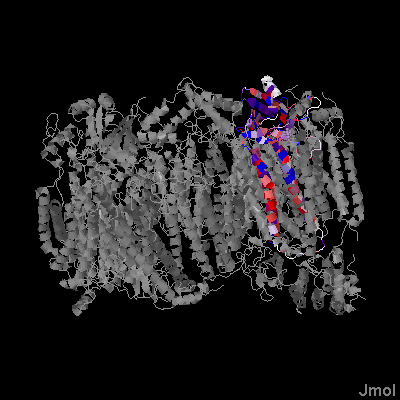
| PDB код
|
Тип
(спираль, баррель)
|
Какая мембрана
(внутренняя или внешняя, организм, органелла)
|
Толщина гидрофобной части мембраны в ангстремах
|
Медиана числа остатков в одном трансмембранном участке
|
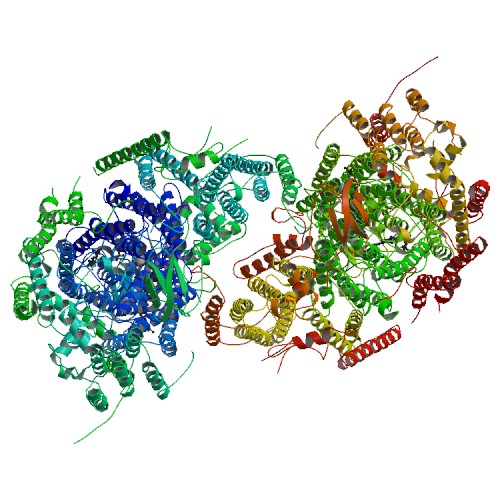
| 2GFP
|
спираль
|
Внутренняя мембрана грамм-положительной бактерии Escherichia coli
|
31.6 ± 1.5
|
21
|
| 3BEH
|
спираль
|
Внутренняя мембрана грамм-отрицательной бактерии Rhizobium loti
|
30.6 ± 0.8
|
20
|
| 1AP9
|
спираль
|
внешняя мембрана грамм-отрицательной бактерии Halobacterium salinarum
|
30.6 ± 1.2
|
19.5
|
| 3EFM
|
баррель
|
внешняя мембрана грамм-отрицательной бактерии Bordetella pertussis
|
24.7 ± 0.6
|
22
|
| 1QJP
|
баррель
|
внешняя мембрана грамм-положительной бактерии Escherichia coli
|
25.4 ± 1.5
|
9
|
| 2O4V
|
баррель
|
наружная мембрана грамотрицательной бактерии Pseudomonas aeruginosa
|
23.9 ± 1.1
|
16
|