Занятие 1.Работа с PyMol
Главная
Работа в PyMOL с записью 1LMP банка PDB.
1.
Оценила возможности Sculpting, в Wizard->Demo->Sculpting.
2.
Средствами Tcl/Tk интерфейса (Wizard->Mutagenesis) провела мутацию в белке, которая, по-моему, должна привести к потере связывания с лигандом.
Предварительно оценила зону котнакта.
select NAG, resname NAG
select NDG, resname NDG
select lig, NAG or NDG
select prot, 1lmp and !(lig)
select contact, prot within 3.5 of lig
dist hbond, prot, lig, mode=2
Мутировала ASN-103 на ARG-103.
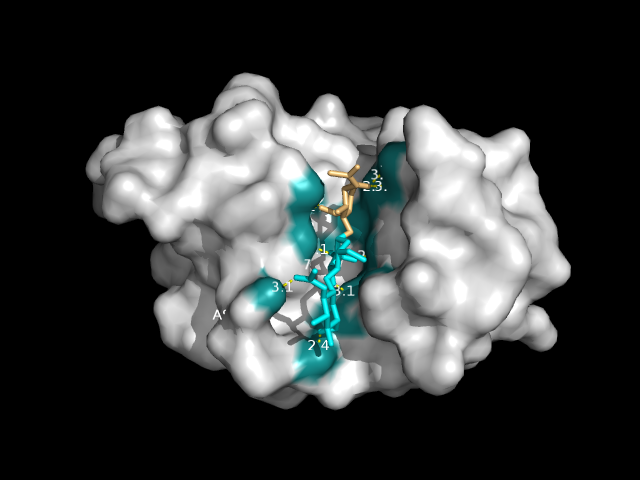
Рис.1. Изображена поверхность белка и лиганды (NAG-серый,NDG-циановый);поверхность контакта выделена бирюзовым. |
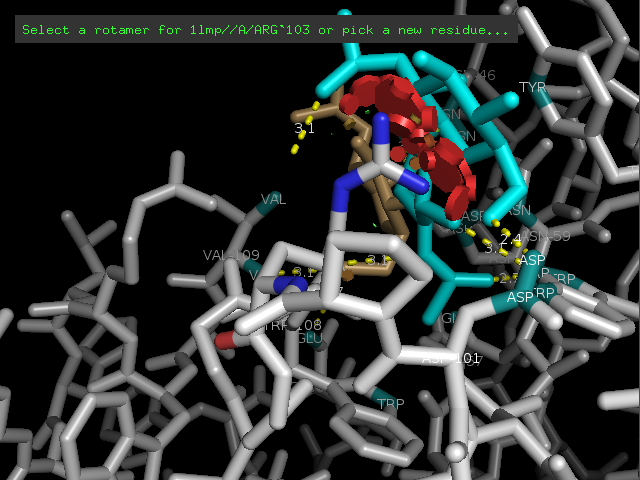
Рис.2. Мутация ASN-103 на ARG-103. |
На рис.2 видно, что мутация приводит к потере связывания с лигандом (NDG).
3.
Используя команды mset, mview, super, translate, создала анимационный ролик (mpeg) где происходит совмещение белков и показывается место мутации.
Видео.
4.
Приседенила флуорусцентную метку TAMRA к белку через сложноэфирную связь. Использовала команды fuse и torsion.
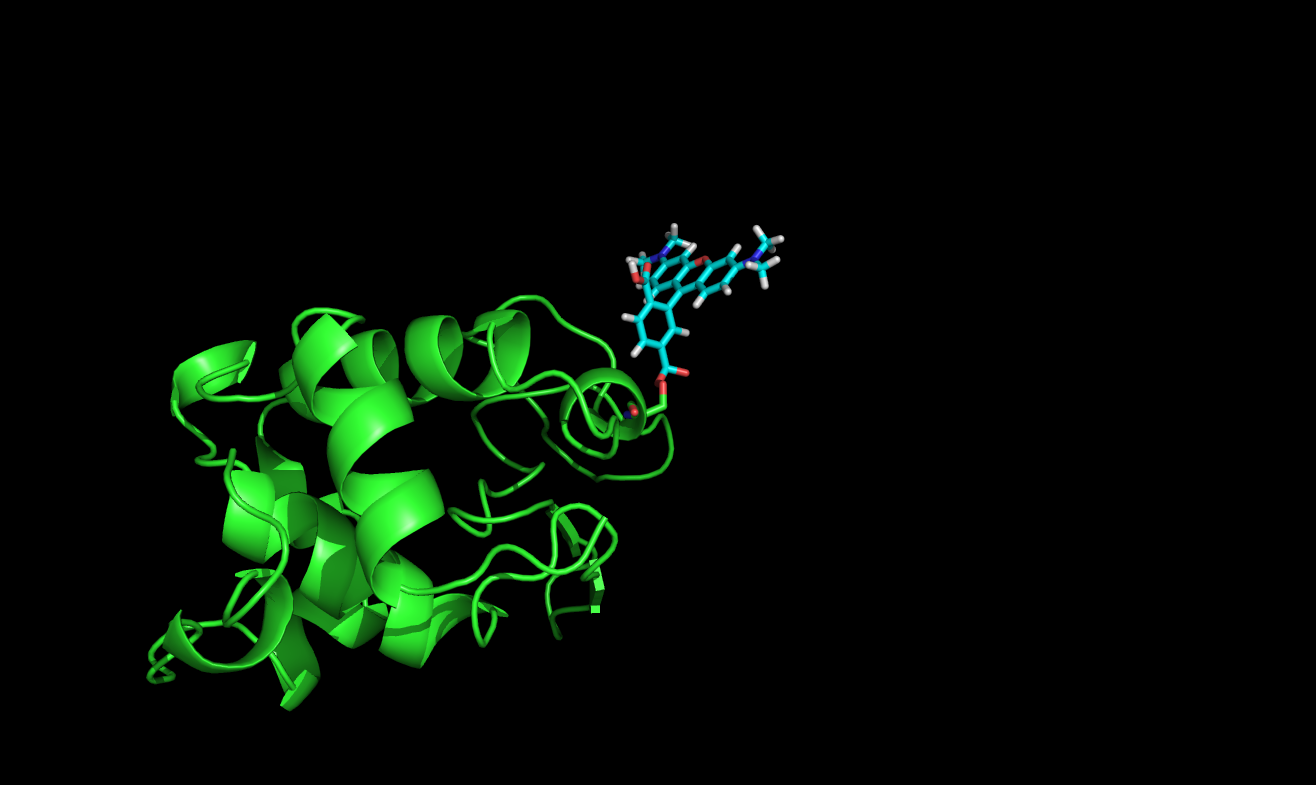
Рис.3. Белок 1LMP с флуорусцентной метку TAMRA. |
5.
Построение поли-изолейциновой альфа спирали длинной 100 аминкислот.

Рис.4.Поли-изолейциновая альфа спираль. |
Скрипт.
6.
Используя возможности babel строить третичную структуру вещества на основе SMILE нотации:
obgen my.smi > my.mol
Построила структуру кубана.

Рис.5.Структура кубана |
©Eliseeva Julia





