Занятие 7. Анализ молекулярной динамики биологических молекул в GROMACS
Главная
em.mdp - файл праметров для минимизации энергии,
Анализ результатов работы на суперкомпьютере
Визуальный анализ движения молекул
Конвертировала в pdb-формат:
trjconv -f dna_md.xtc -s dna_md.tpr -o dna_pbc_1.pdb -skip 20 -pbc mol
dna_pbc_1.pdb - полученный pdb-файл
Переход из A в B форму по визуальному анализу происходит примерно на 45 модели, t= 8800.00000.
Определение средне-квадратичного отклонения в ходе моделирования.
Отклонение в ходе всей симуляции относительно стартовой структуры:
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_1
Относительно каждой предыдущей структуры на растоянии 400 кадров:
g_rms -f dna_md.xtc -s dna_md.tpr -o rms_2 -prev 400
rms_1.xvg,
rms_2.xvg.
Привожу картинки, полученные с помощью gnuplot.
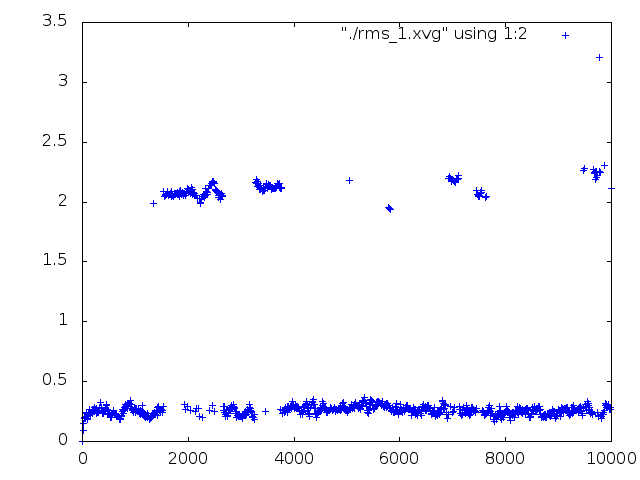
rms_1.png |
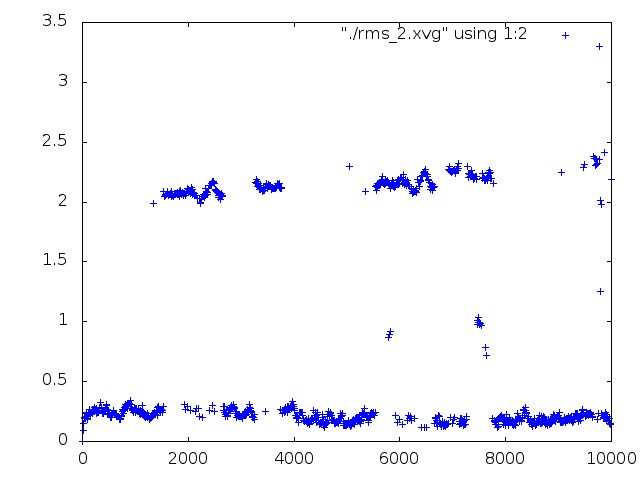
rms_2.png |
Из второго графика (rms_2.png) видим, что квадратичное отклонение, на самом деле, становится меньше.
Далее определение изменения гидрофобной и гидрофильной поверхности в ходе конформационного перехода:
g_sas -f dna_md.xtc -s dna_md.tpr -o sas_dna.xvg
sas_dna.xvg - полученный файл.
Построила зависимость изменения гидрофобной и гидрофильной поверхностей, доступных растворителю от времени.
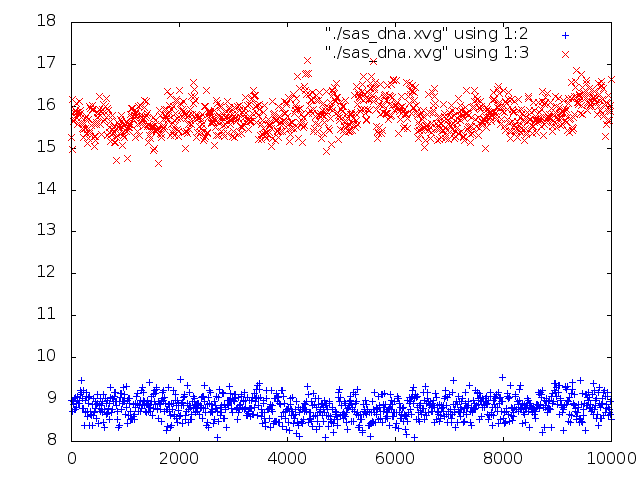
sas_dna.png |
Видим, что гидрофобность поверхности(синий цвет) не меняется, а у гидрофильности(красный цвет) в конце есть пик
Далее выполнила команду для исследования связей ДНК-ДНК:
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_dna
hbond_dna.xvg - полученный файл.
И с помощью gnuplot получила график.
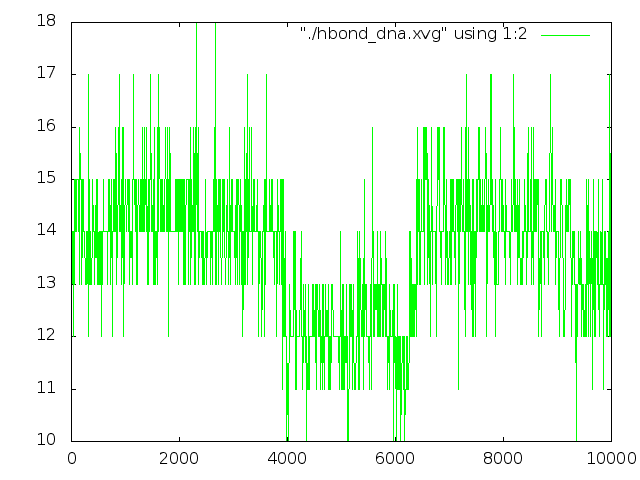
hbond_dna.png |
В результате, принципиально число водородных связей не меняется.
Для изчения количества вдородных связей ДНК-вода, воспользовалась командой:
g_hbond -f dna_md.xtc -s dna_md.tpr -num hbond_sol
hbond_sol.xvg - полученный файл.
И график:
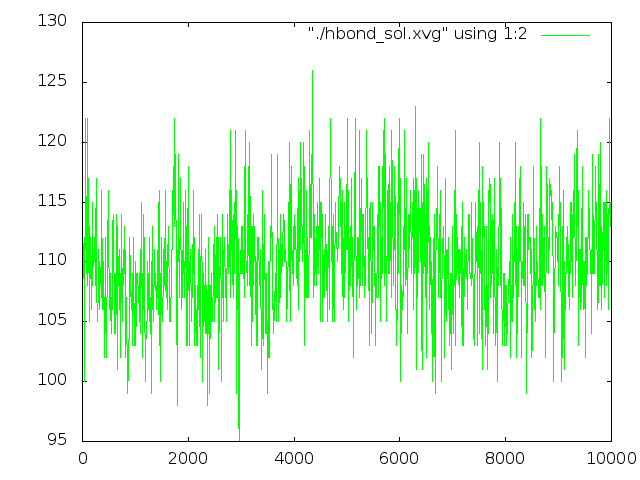
hbond_sol.png |
Видно, что в начале и в конце число водородных связей одинаково.
Следовательно, небольшие изменения в числе водородных связей не препятствуют ДНК, чтобы перейти из одной формы в другую.
©Eliseeva Julia





