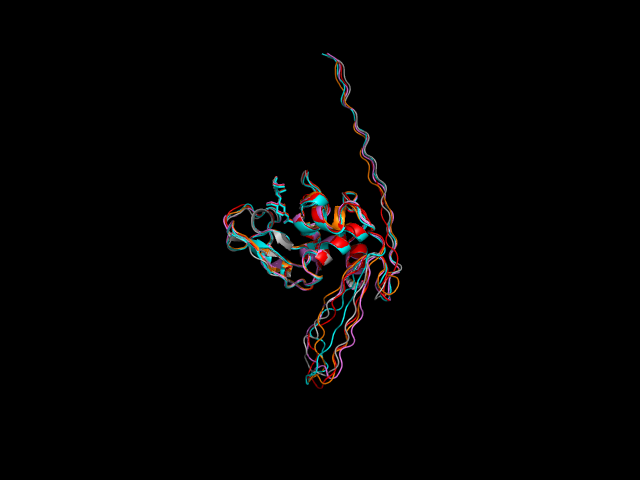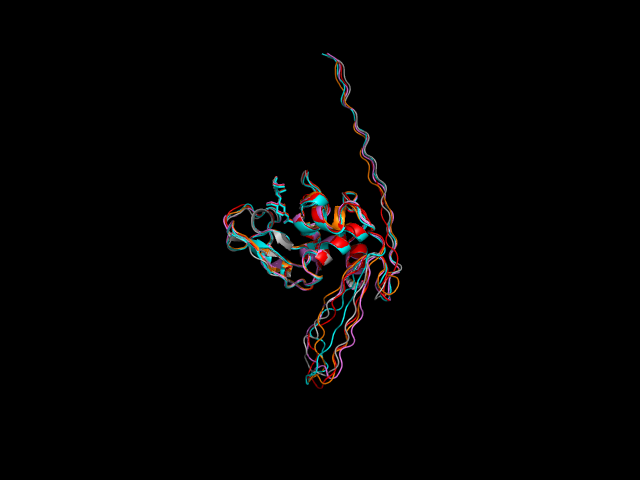Гомологичное моделирование комплекса белка с лигандом
Главная
Цель данного занятия ознакомится с возможностями гомологичного моделирования комплекса белка с лигандом.
В этом занятии мы будем пользоваться пакетом Modeller.
Из списка лизоцимов выбрала: LYS_BPPS1.
В дальнейшем требовалось построить модель комплекса этого белка с лигандом.
1.Выравнивание
Построила выравнивание последовательности из структуры ID: 1lmp и LYS_BPPS1.
Для этого использовала программу ClustalW.
Полученное выравнивание сохранила в формате PIR.
2.Модификация файла выравнивания
Переименовала последовательность в файле выравнивания (как указано в задании):
align.pir.
3.Модификация файла со структурой
Удалила всю воду из структуры (в текстовом редакторе), всем атомам лиганда присвоила один и тот же номер "остатка".
И модифицировала имена атомов каждого остатка, добавив в конец буквы A, B, C.
После модификации имен атомов изменила номера остатков на 130.
Сохранила в файле:
1lmp_now.ent.
4.Создание управляющего скрипта lys_bpps1.py
Далее, используя заготовку, дописала скрипт.
lys_bpps1.py.
5.Запуск скрипта
Запустила исполнение скрипта командой:
mod9v7 lys_bpps1.py &
6. Полученые модели
Получила 5 моделей. В приведенном ниже рисунке, изображающим их наложение, заметно, что они сходны.
Также есть неструктурированный участок (видимо, программа не разобралась).
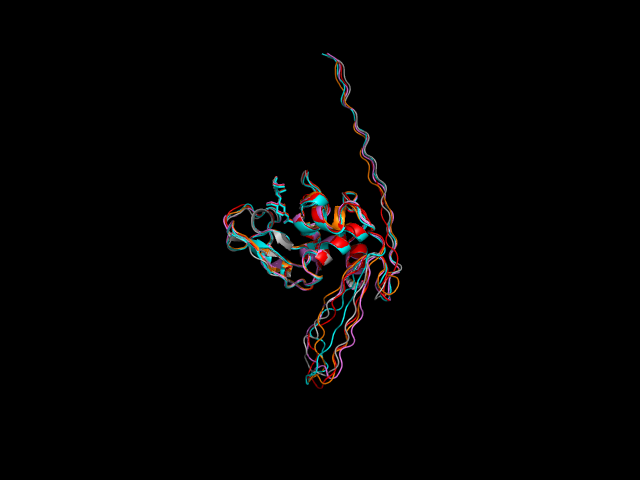
Рис.1. Наложение 5 моделей. |
7. Проверка качества моделей
Для оценки качества структуры использовала сервер WHATIF.
Искала лучшую модель. Видимо, это 4-я.
По каждому из параметров искала лучшую модель, вот что получилось:
Ramachandran Z-score : 1-я модель
RMS Z-score for bond angles: 2-я модель
Omega average and std. deviation: 4-я модель
RMS Z-score for bond lengths: 5-я модель.
©Eliseeva Julia