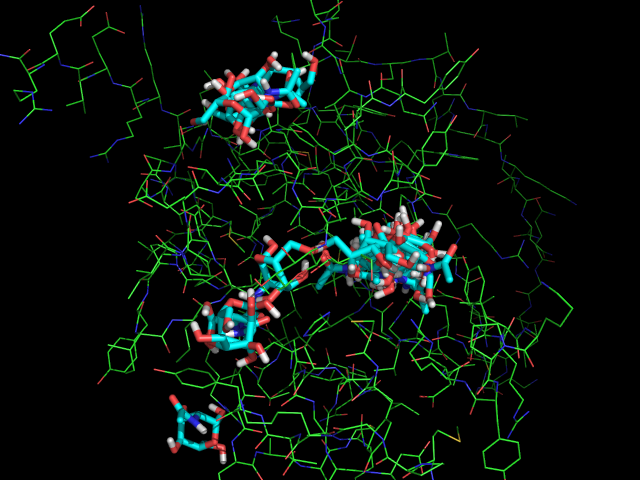
Рис.1.Все состояния лиганда.

|
|
OH
|
NH2
|
H
|
Ph
|
|
1
|
-5.1
|
-5.2
|
-4.8
|
-6.2
|
|
2
|
-5.0
|
-5.1
|
-4.6
|
-6.1
|
|
3
|
-4.9
|
-5.0
|
-4.5
|
-6.1
|
|
|
OH
|
NH2
|
Ph
|
|
1
|
-5.1
|
-5.0
|
-6.1
|
|
2
|
-4.8
|
-5.0
|
-6.0
|
|
3
|
-4.8
|
-4.9
|
-5.7
|