Занятие 1. Электронная плотность. Рассеяние на электроне.
Главная
1.Выбрала структуру белка для дальнейшей работы
Взяла структуру, с которой работала на первых курсах (1W5D).
Она удовлетворяет требованиям:
Сервис EDS знает структуру.
Имеются подходящие структурные гомологи белка. Проверка:
PDBeFold => launch => Query - PDB код, в таблице с находками посмотрела чтобы было 5 или более разных белков с RMSD от 0.8 до 2.5 ангстрем (оценка сходства) и N_align (число сопоставленных C_alpha) от половины числа аминокислотных остатков входного белка до 90%.
2.Скачала мою модель и файл структурных факторов из PDB
Скачала и загрузила через меню PDB файл (1W5D.pdb).
Файл с электронной плотностью получила на сайте EDS (http://eds.bmc.uu.se/eds/).
EDS
На странице с информацией о записи PDB в разделе Download выбрала "Maps" и заказала карту в формате по умолчанию (1w5d.omap.gz).
Скачанный файл разархивировала, прежде чем загружать в PyMOL (1w5d.omap).
Загрузила файл с электронной плотностью командой load:
PyMOL> load 1w5d.omap, 1w5d_map
3.Построила изображение электронной плотности вокруг полипептидной цепи и трех-пяти аминокислотных остатков, различных по типу
Выбрала аминокислотные остатки 241-244(svwe;серин, валин,триптофан,глутаминовая кислота).
Сначала выделила данные аминокислотные остатки с помощью команд:
PyMOL>select residues, resi 241-244
PyMOL>show sticks, residues
PyMOL>zoom residues
Удалила воду:
PyMOL>remove resn hoh
Затем оставила только выбранные аминокислотные остатки:
PyMOL>hide lines, all
Использовала 3 уровня подрезки(1, 1.5, 3) электронной плотности чтобы оценить качество модели пространственной структуры, представленной в PDB файле.
Для визуализации электронной плотности вокруг заданного множества использовала команду isomesh:
PyMOL> isomesh new_map, 1w5d_map, 1, resi 241-244, 2,5, carve=2.1
Улучшила картинку с помощью команд:
PyMOL>orient residues
PyMOL>ray
И сохранила изображение:
PyMOL>png el_plot_1
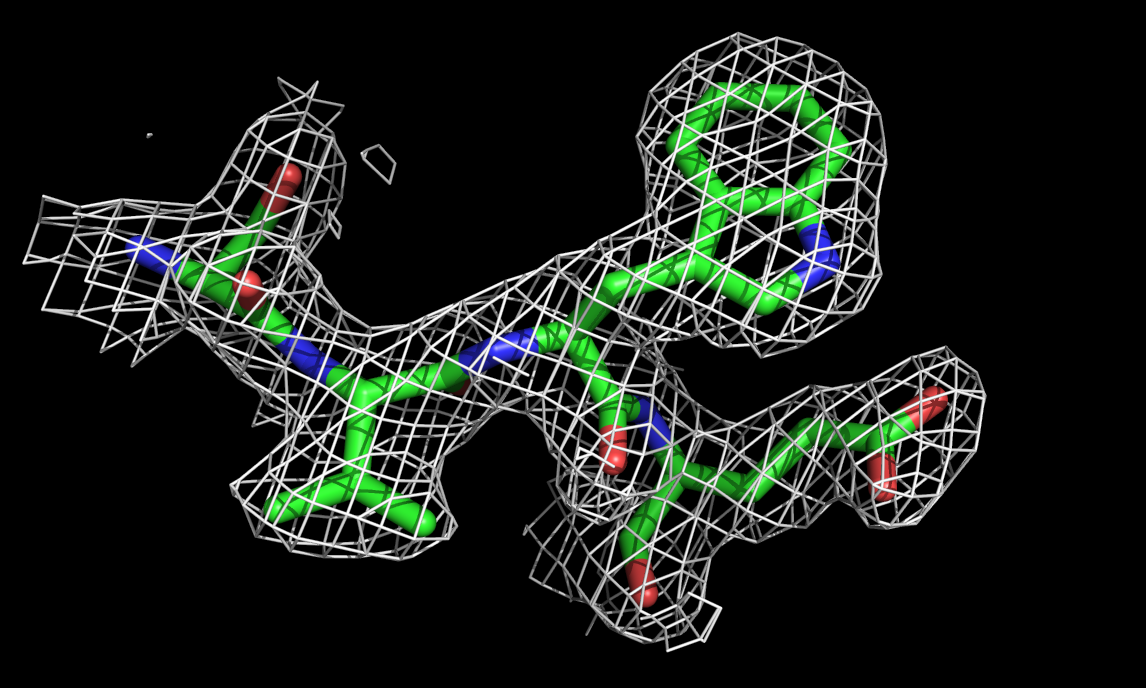
Рис.1.уровень подрезки 1 |
Это для уровня подрезки 1, аналогично сделала для 1,5 и 3 (соответственно,1 - белый цвет, 1,5 - синий, 3 - оранжевый).
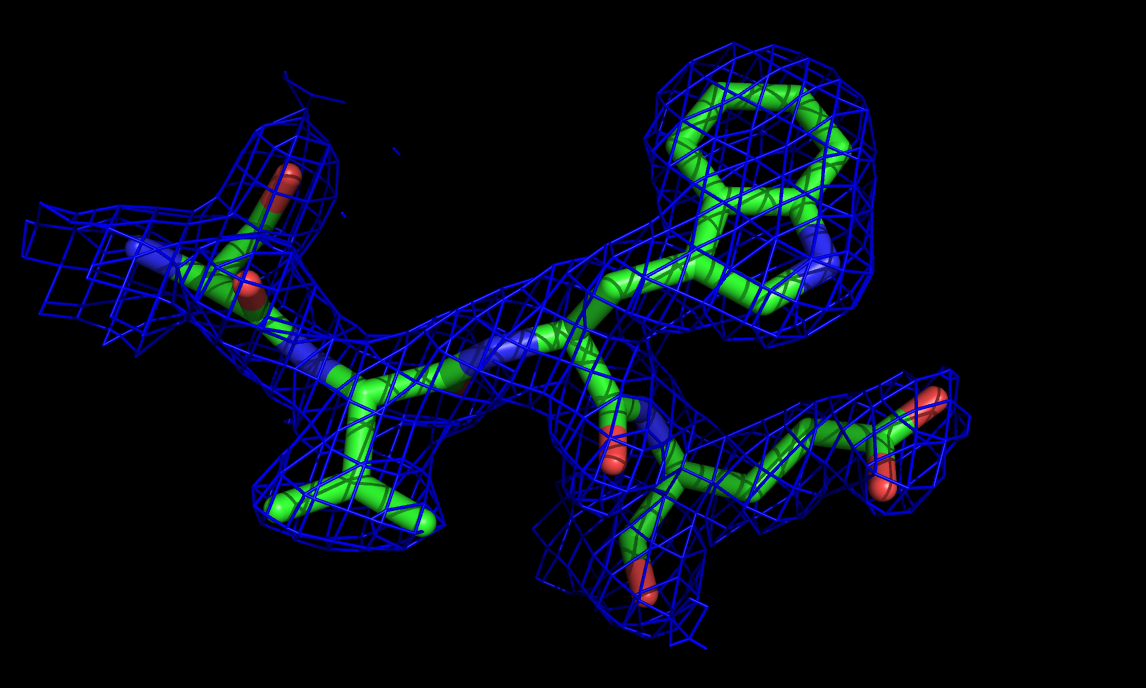
Рис.2. уровень подрезки 1,5 |
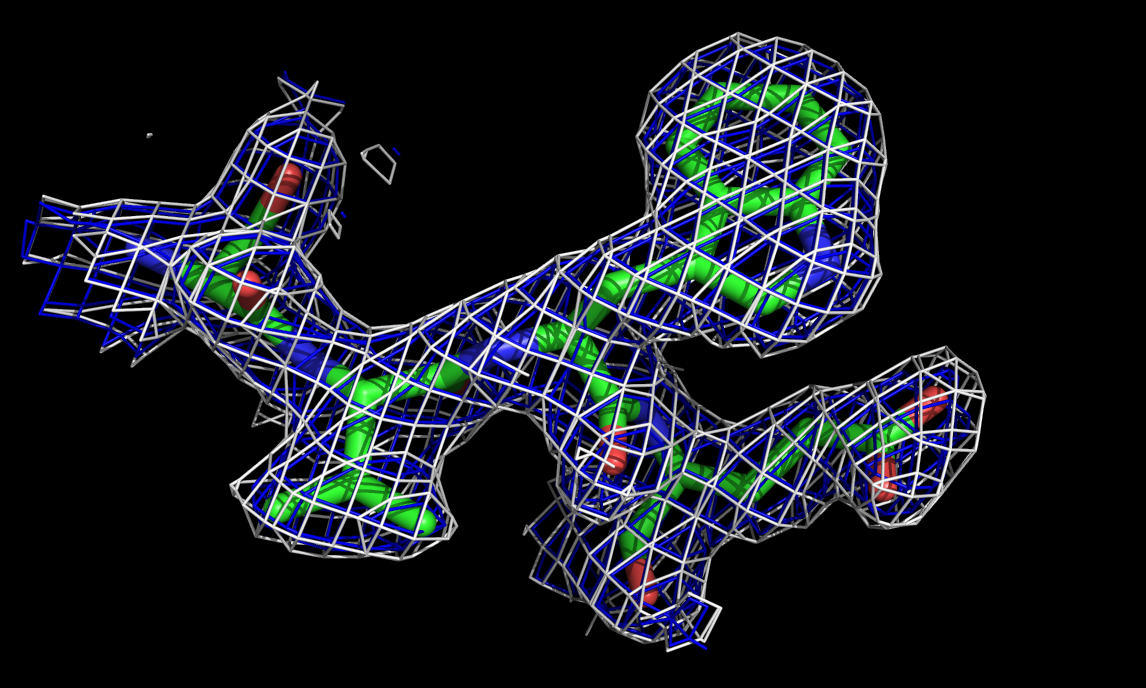
Рис.3.уровни подрезки 1 и 1,5 |
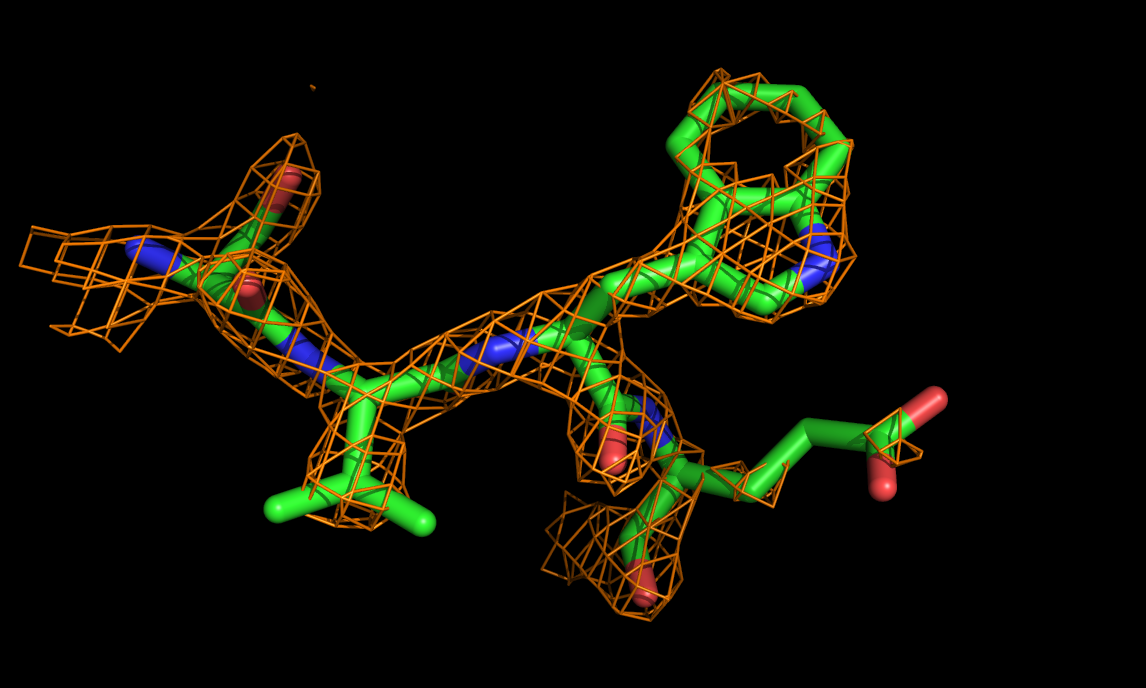
Рис.4. уровень подрезки 3 |
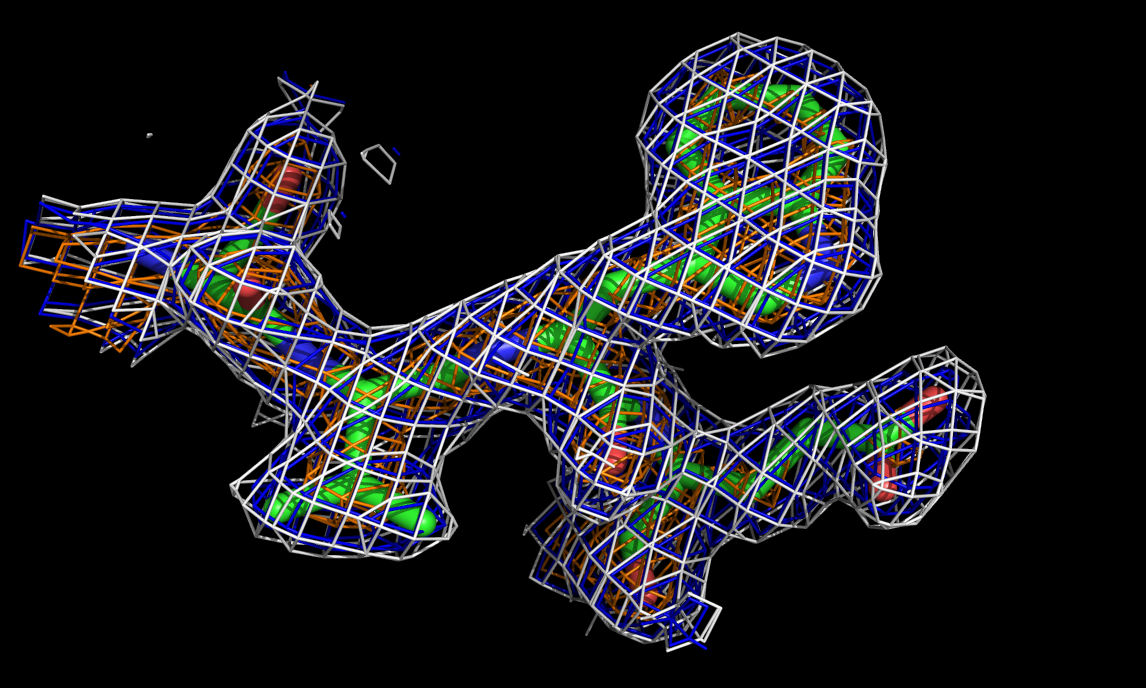
Рис.5. уровни подрезки 1, 1.5 и 3 |
Затем аналогично построила изображение электронной плотности вокруг полипептидной цепи (1 - циановый цвет, 1.5 - лимонный,3 - фиолетовый).
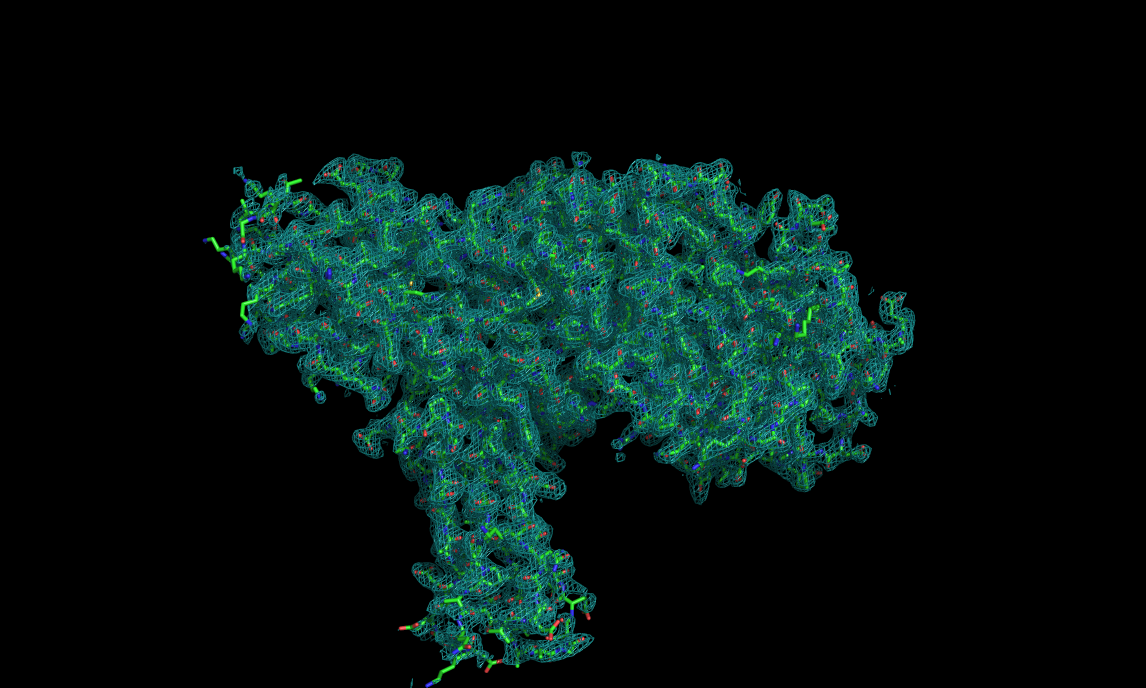
Рис.6. Для уровня подрезки 1 |
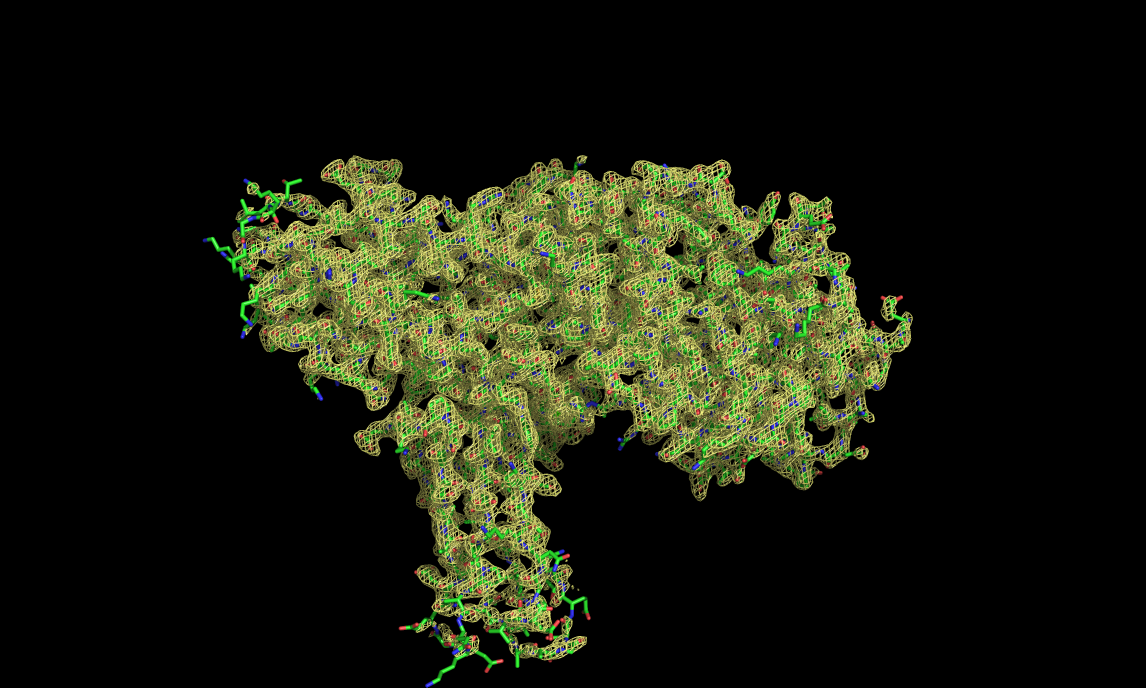
Рис.7. уровень подрезки 1,5 |
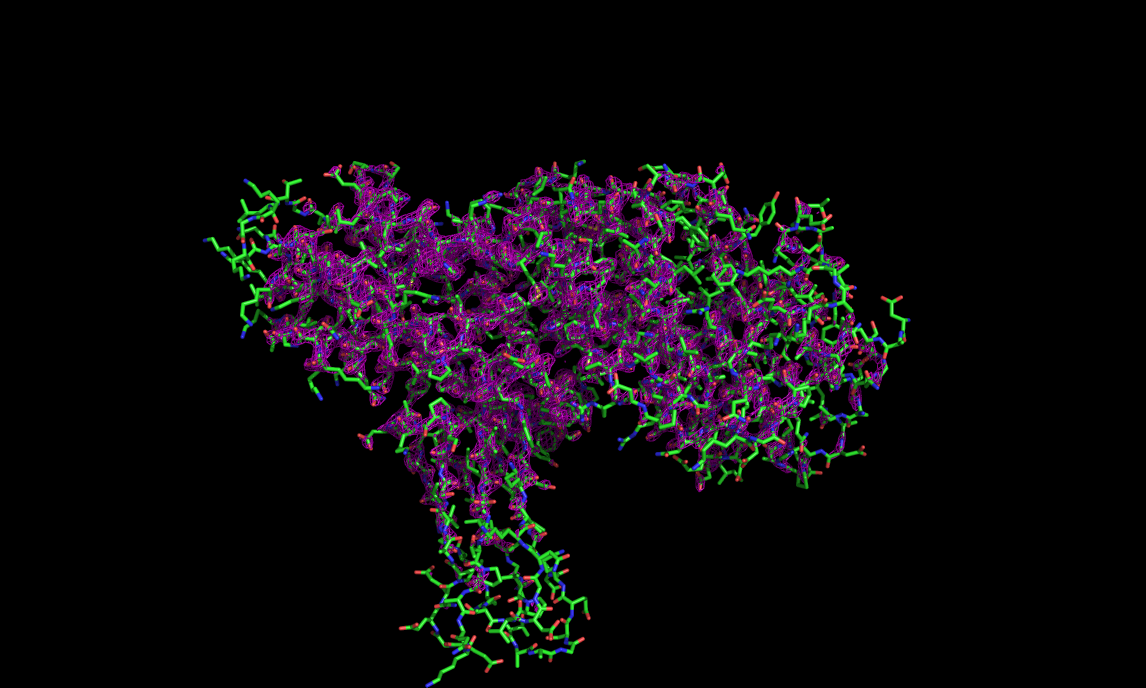
Рис.8. уровень подрезки 3 |
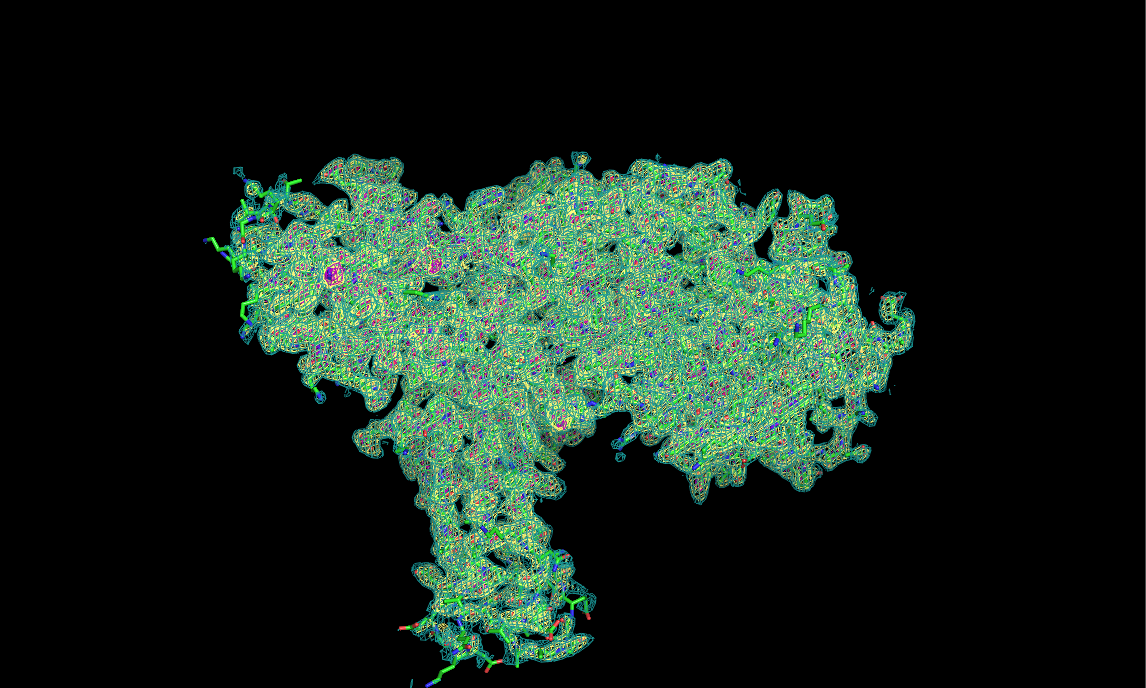
Рис.9. уровни подрезки 1, 1.5 и 3 |
Рассматривая приведенные изображения, можно заметить, что почти все атомы вписываются в электронную плотность на разных уровнях подрезки, что говорит о высоком качестве данной структуры.
Между структурными факторами и атомами модели нет соответствия, но зная координаты атомов в модели, можно, используя формулу, получить значения структурных факторов.
©Eliseeva Julia









