Занятие 3
1. Укоренение в среднюю точку
Деревья построенные алгоритмом UPGMA ультраметрические и укоренненые. По этому
укоренять их бесполезно.
Я укоренила в среднюю точку деревьево, построенное при выполнении
задания 4 предыдущего занятия методом neighbor-joining.
Для этого я использовала программу retree пакета PHYLIP.
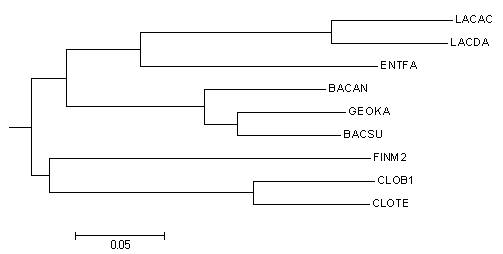
Дерево до укоренения
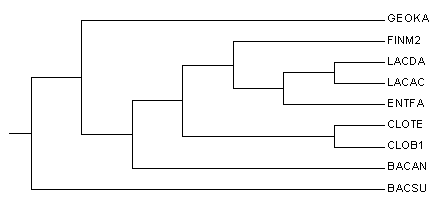
Дерево после укоренения
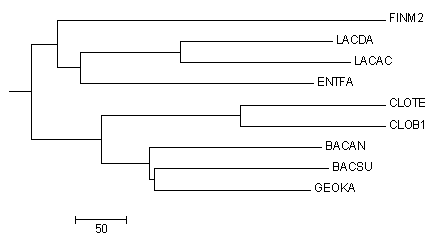
Правильное дерево
Дерео укоренилось в
ветвь {FINM2,LACDA,LACAC,ENTFA} против {CLOTE,CLOB1,BACAN,BACSU,GEOKA}
относительно правильного дерева укоренение неправильное, укорененная ветвь разделяет дерево не на теже множества, что и в правильном дереве.
2. Использование внешней группы
Деревья, построенные методом максимальной экономии
("Maximum parsimony") невозможно укоренить в среднюю точку,
при этом методе учитывается количествово мутацый, а не растояние между ветвями.
Я воспользовалась укоренением с помощью внешней группы.
Реконструиролала методом максимальной экономии укоренённое
дерево отобранных мной бактерий, используя в качестве внешней
группы белок семейства RS2 из кишечной палочки (Escherichia coli, ECOLI).
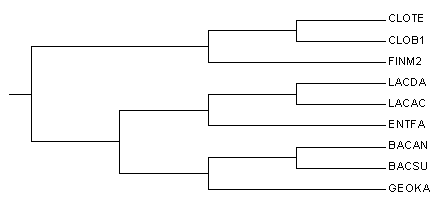
Дерево с ветвью ECOLI в качестве корня
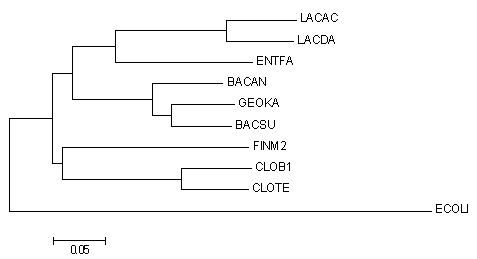
Дерево без внешней группы
Правильное дерево

{FINM2,CLOTE,CLOB1} против {LACDA,LACAC,ENTFA,BACAN,BACSU,GEOKA}
Полученное дерево очень похоже на правильное (отличие только расположение BACAN).
относительно правильного дерева укоренение правильное.
3. Бутстрэп
Я провела бутстрэп-анализ филогении моих белков, используя
один из методов, доступных из прграммы MEGA. Для этого в
окошке, которое открывается после вызова программы, в меню
"Test of Phylogeny" выбрала "Bootstrap method".
Число реплик, равное 100.
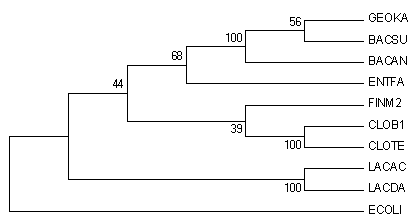
Bootstrap consensus tree
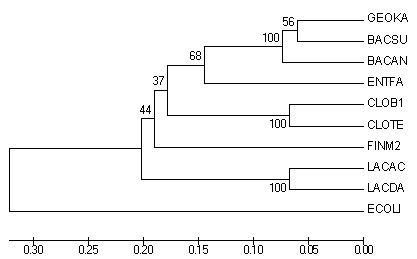
Original tree
Правильное дерево

Полученные деревья не отличаются, следовательно в данном
случае не важно строилось дерево по половине данных или
по всем.
© Julia Chudakova








