Рис.1 Структура 7PGB
1. Поиск странностей в PDB-файлах
7GPB
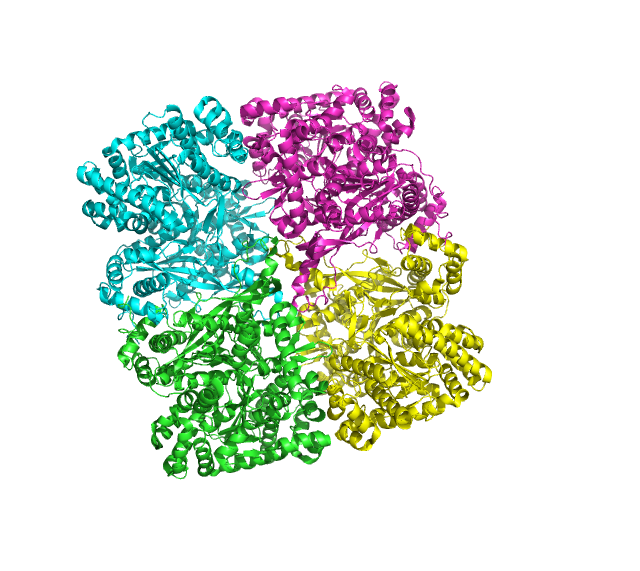
Структура 7GPB была расшифрована в 1991 году в ходе исследования: Structural mechanism for glycogen phosphorylase control by phosphorylation and AMP. Разрешение структуры - 2,9 Å. Это структура гомотетрамерного фермента гликогенфосфорилазы b, в комплексе с АМФ.

Рис.1 Структура 7PGB
В этой структуре визуально обнаруживается нарушение планарности боковых цепей: кольца некоторых триптофанов лежат не в одной плоскости, чего на самом деле быть не может. Ниже изображены триптофаны-67 цепей A и D, наиболее выраженно отклоняющиеся от нормального вида.
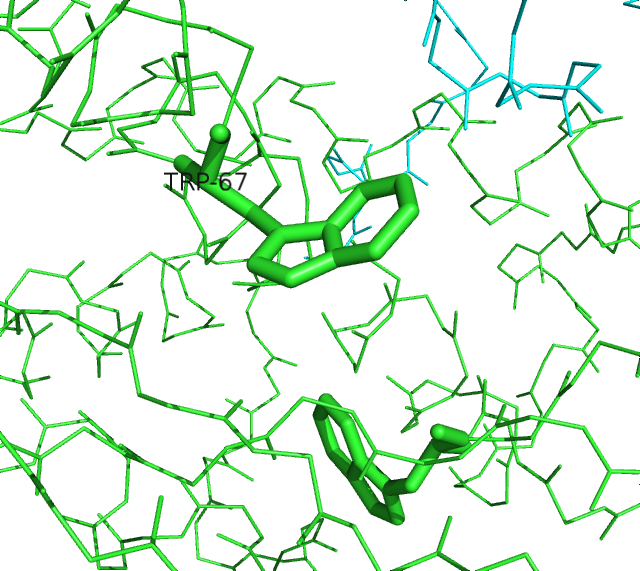 | 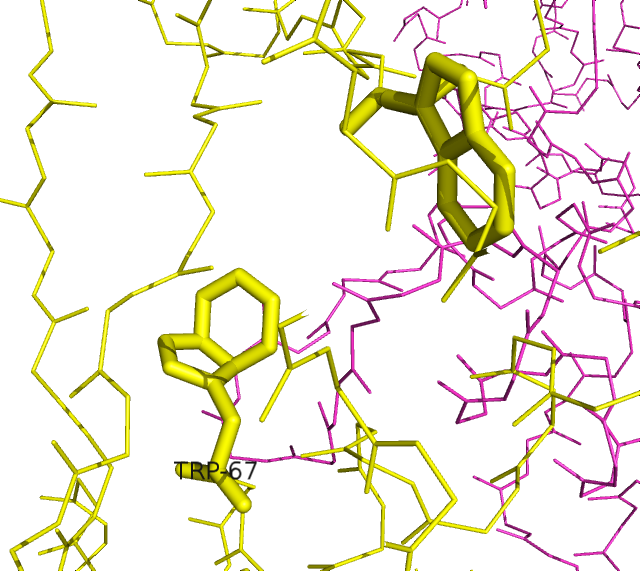 |
В PDB-файле можно найти информацию о температурных факторах (B-факторах), которые могли бы немного объяснить происходящее:
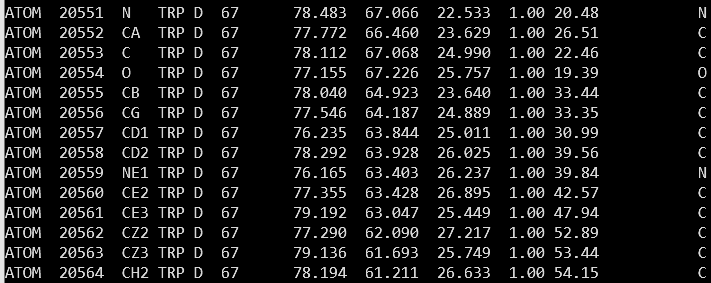 | 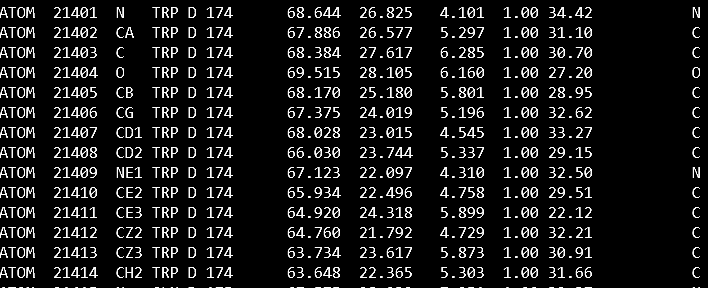 |
Действительно, для 67-го триптофана B-факторы (последняя колонка) для C-атомов боковой цепи значительно больше соответствующих B-факторов триптофана-174, хоть и B-факторы атомов главной цепи меньше для Trp-67. Для Trp-67 цепи А ситуация с B-факторами аналогичная, они немного выше обычного:
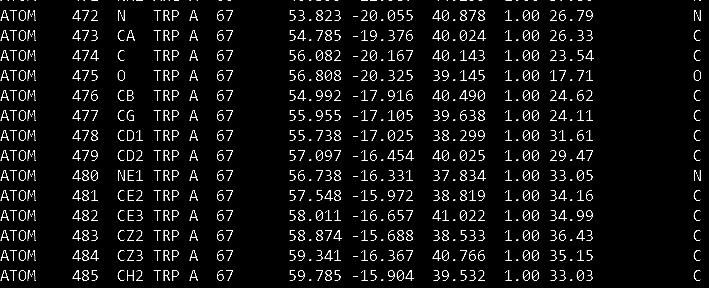
Рис.4 Данные в PDB-файле по необычному триптофану-67 цепи A.
В базе данных PDBREPORT нашла информацию по данной структуре и поискала обнаруженные для триптофанов ошибки. Между атомами NE1 и CE2 триптофана-67 цепи А детектирована необычная длина связи, а в графе "Side chain planarity problems" первые три наиболее отклоняющиеся остатка - как раз наши триптофаны (число в поледней колонке характеризует отклонение (в сигмах) подсчитанного RMS от нормального):
2527 TRP ( 67-) D 36.29 58 TRP ( 67-) A 12.99 1170 TRP ( 361-) B 10.86
5RXN
Структура расшифрована в 1984 году, с разрешением 1,2 Å. Это мономерный рубредоксин, связанный с ионом Fe.
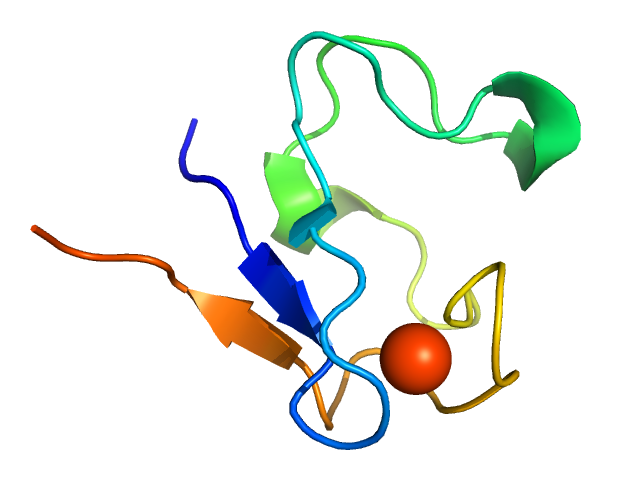
Рис.5 Структура рубредоксина в комплексе с атомом Fe, 5RXN.
В этой структуре обнаруживаются проблемы с треонином. Для белков характерен только один стереоизомер, L-треонин, хотя теоретически существует 4 формы, т.к. у треонина 2 хиральных атома. Здесь же, кроме L-треонина, можно найти также L-аллотреонин:
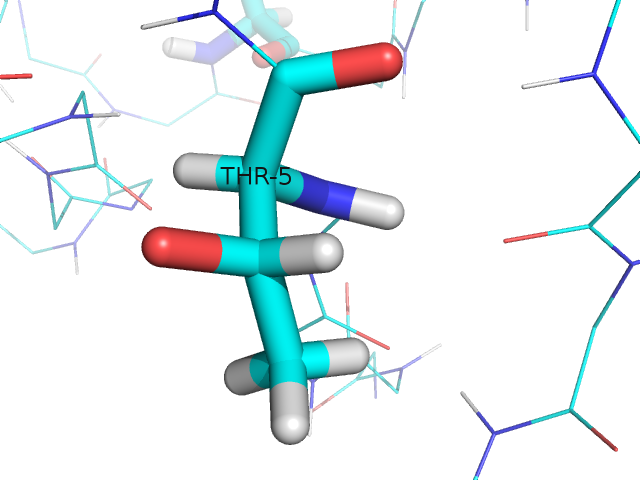 | 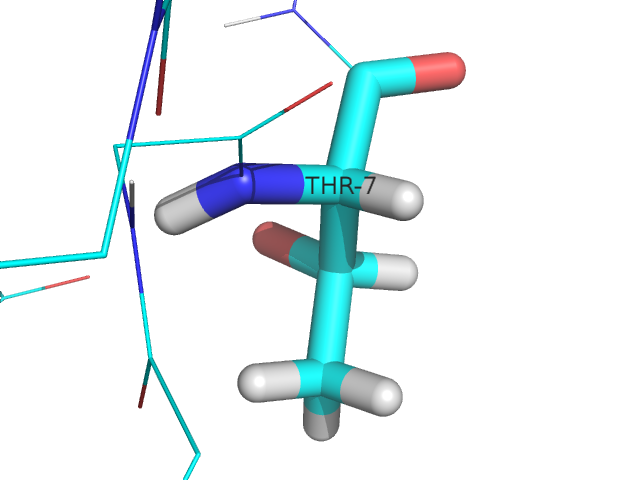 | 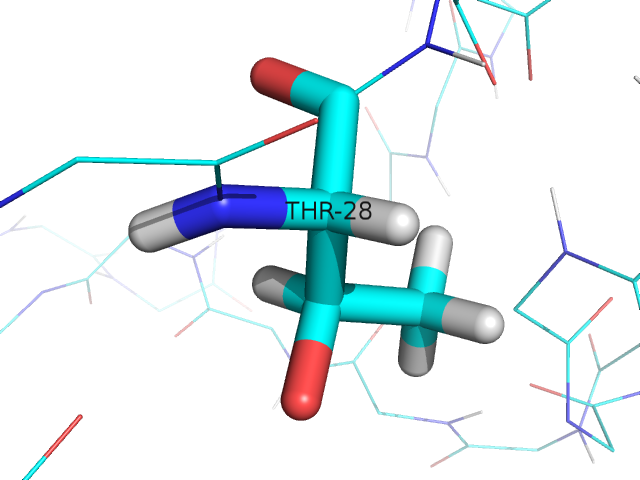 |
В PDBREPORT отмечена нетипичная длина связи между атомами CB и OG1 пятого треонина, а также его нетипичные углы (число в последне колонке означает количество сигм, на которые отклоняется значение угла от нормального):
5 THR ( 5-) A CA CB OG1 117.16 5.0 5 THR ( 5-) A CG2 CB OG1 118.47 4.6
В записи PDB информация о структурных факторах соответствует происходящему:
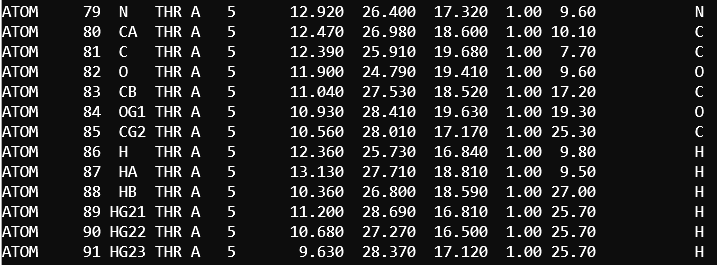 | 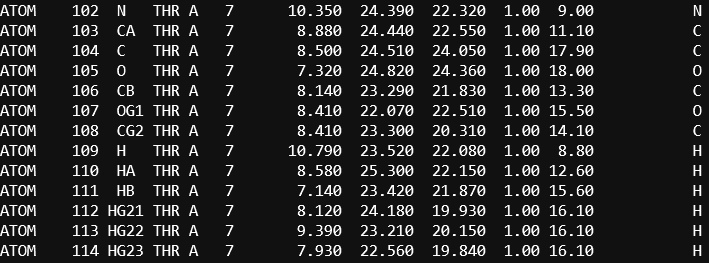 |
B-факторы для углеродов боковой цепи пятого треонина заметно больше, чем у седьмого.
Xиральностью обладают также боковые цепи изолейцина и гидроксипролина (из тех аминокислот, что встречаются в белках).
1DLP
Структура 1DLP была расшифрована в 2000 году , разрешение - 3,3 Å. Белок относится к лектинам и представляет собой гексамер.
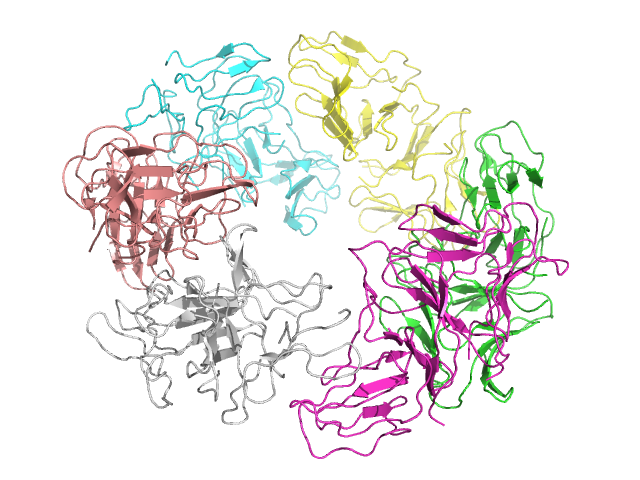
Рис.8 Структура 1DLP.
Если посмотреть на pdb-файл, сразу можно заметить значительного размера списки пропущенных остатков и атомов, а также атомов с коэффициентом заполнения 0; там же есть и предупреждение о том, что данным по этим атомам доверять нельзя:
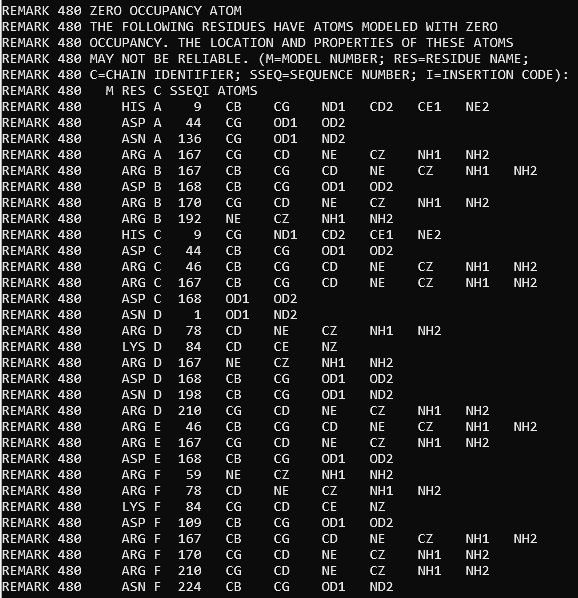
Рис.8 Данные в pdb-файле об атомах с нулевым коэффициентом заполнения.
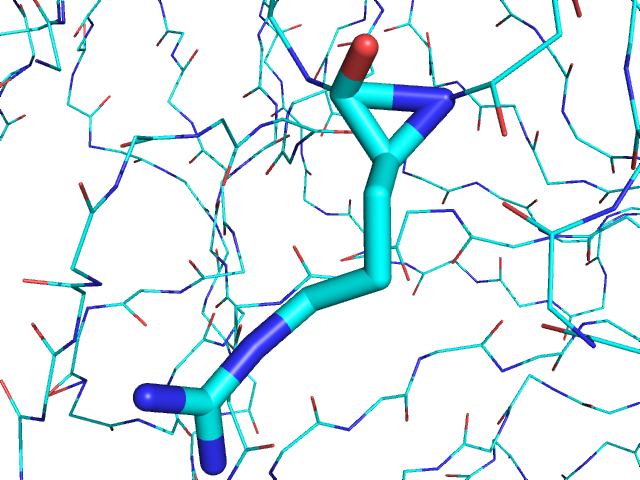
Возьмем первый же остаток из списка - девятый гистидин цепи А.
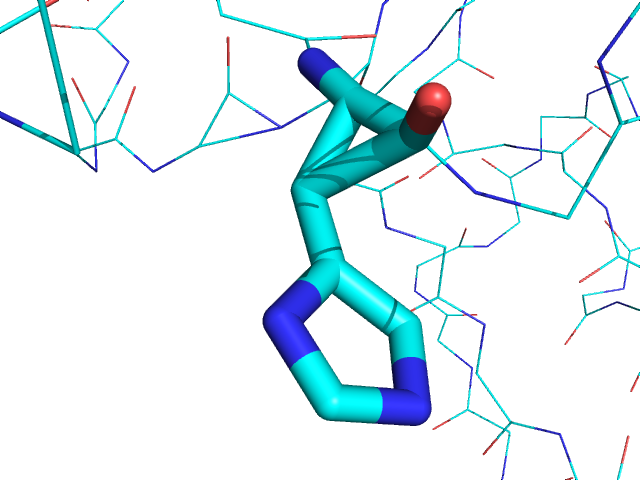
Рис.9 Структура His9A.
Видим, что строение остатка противоречит природным законам. То же самое касается остатков Asn136A и Arg167C:
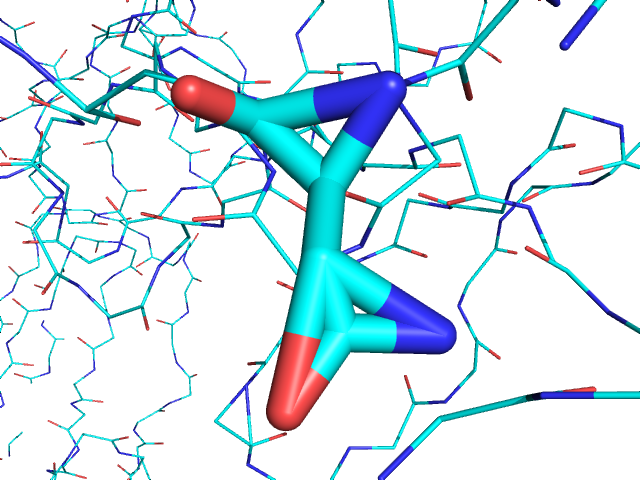 |  |
В PDBREPORT тоже есть информация об этих особенностях, а так же о многочисленных нетипичных длинах связей, углах, конформациях остова, нарушениях хиральности и т.д. Там также говорят, что нулевой коэффициент заполнения присваивается иногда в тех случаях, когда атома на самом деле не видно.
2. Сравнение коэффициентов заполнения и B-факторов в структурах атомного и среднего разрешения.
Воспользовавшись Advanced Search в PDB, выбрала две структуры: 4K8M с разрешением 0.87 Å и 4NKR с разрешением 2,41 Å.
В pdb-файле нашла данные по B-факторам и коэффициентам заполнения. Ниже представлены результаты в виде гистограмм распределения.
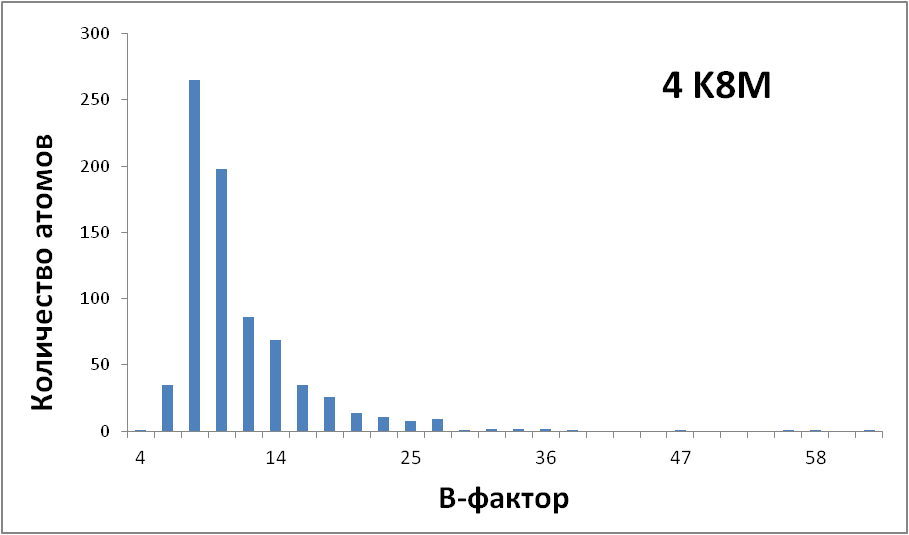 | 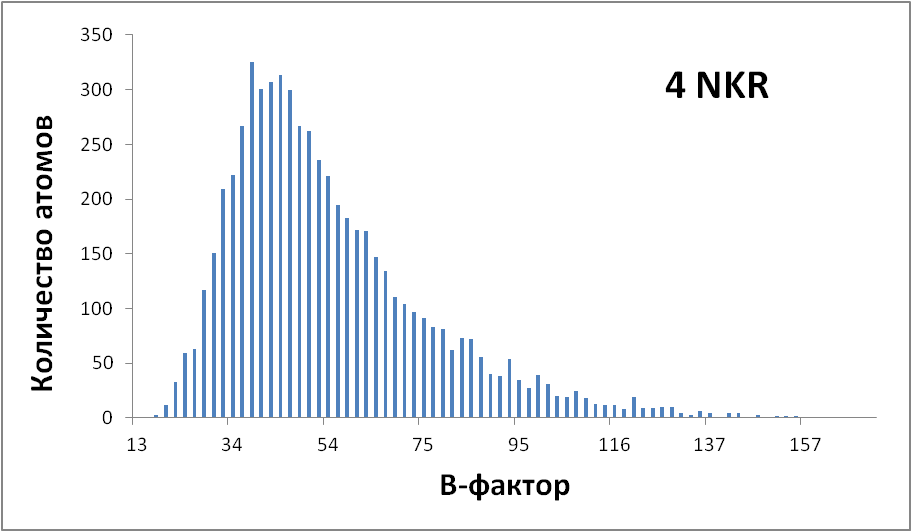 |
Видно, что диапазон значений фактора шире, а среднее значение значительно выше - у структуры с более низким разрешением, 4NKR.
Для 4NKR все коэффициенты заполнения равны 1, а для более точной структуры 4K8M распределение следующее:
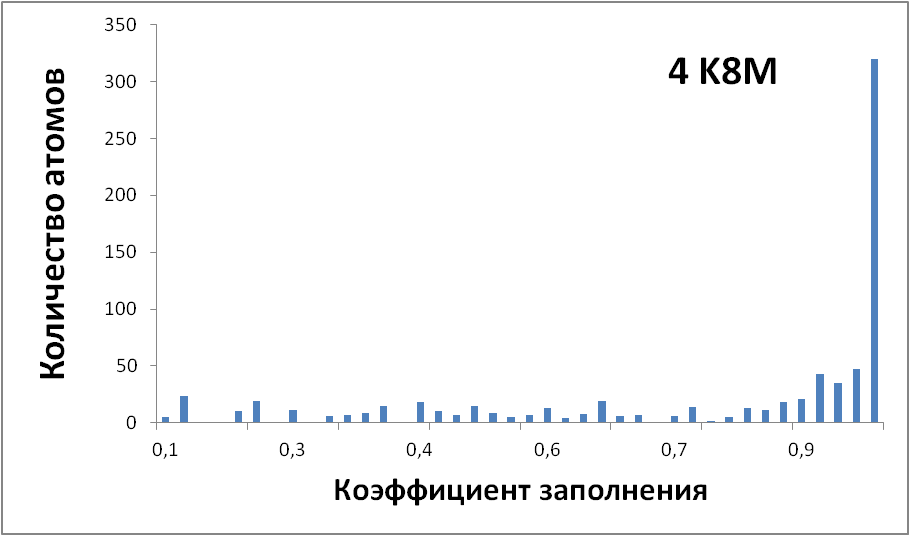
Рис.12 Гистограмма распределения факторов заполнения для 4K8M.