1. Построение совмещения структур белка и его гомологов.
Работа проводилась с тем же белком (pdb-код 1VHE). В PDBeFold найдено 4 гомолога, достаточно разных и не слишком сходных: 1xfo:A, 1ylo:A, 3isx:A, 3cpx:A.
Выравнивание последовательностей по совмещению структур: align_struct.fasta
Совмещение структур: superposed.pdb
Попарное выравнивание: align_pair.fasta
Ниже представлены фрагменты выравнивания по совмещению структур и выравнивания, построенного по алгоритму Tcoffee: align.fasta. В выравнивании по структуре следует смотреть только на большие буквы; маленькие буквы считаются не выровненными.
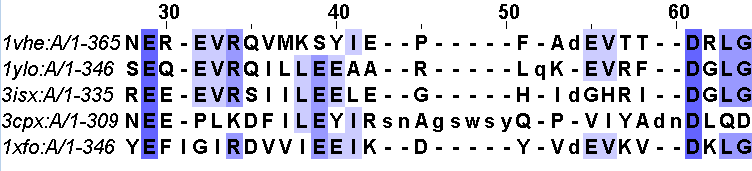
Рис.1 Фрагмент выравнивания по совмещению структур. Окраска по консервативности.
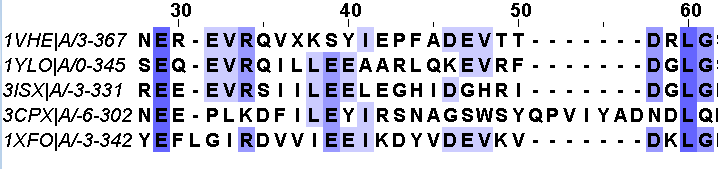
Рис.2 Фрагмент выравнивания, построенного Tcoffee.
Выравнивания на этом участке в очень похожи, несоответствие наблюдается с позиции 43. В первом случае 3CPX образует два "выпетливания", во втором - одно длинное. При этом в выравнивании Tcoffee эта последовательность выравнивается с другими не лучше, чем в структурном выравнивании, и в целом консервативность на различающемся участке слабая в обоих случаях. Очевидных преимуществ ни одно выравнивание не имеет. Посмотрим на изображение совмещения структур на этом участке.
На соответствующих изображениях ниже выделены С-α атомы, соответсвующие столбцу 43 P, R, G, S, D в выравнивании Tcoffee и столбцу 45 P, R, G, A, D в выравнивании по совмещению структур.
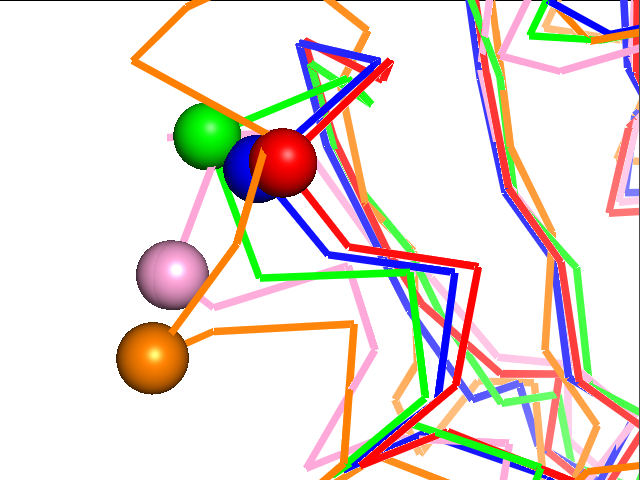 | 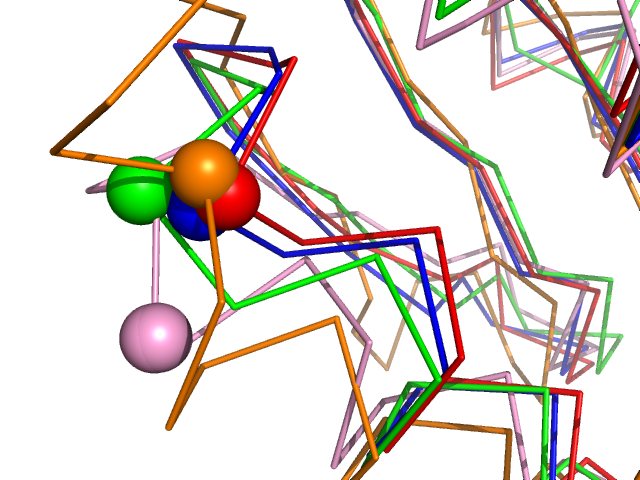 |
Рис. 3 Совмещение структур. Слева выделены выровненные Tcoffee С-α атомы, справа - соответствующие атомы, выровненные при совмещении.
На первый взгляд, по данному столбцу сложно определить, какое из выравниваний правильнее: оранжевая структура сильно отличается от других на данном участке. Но все-таки, кажется, в выравнивании по совмещению больше биологического смысла.
2. Совмещение по заданному выравниванию. Команда pair_fit
Для работы использовалась структура T-клеточного рецептора 1KGC. Ниже приведено изображение ее α- и β-цепочек.
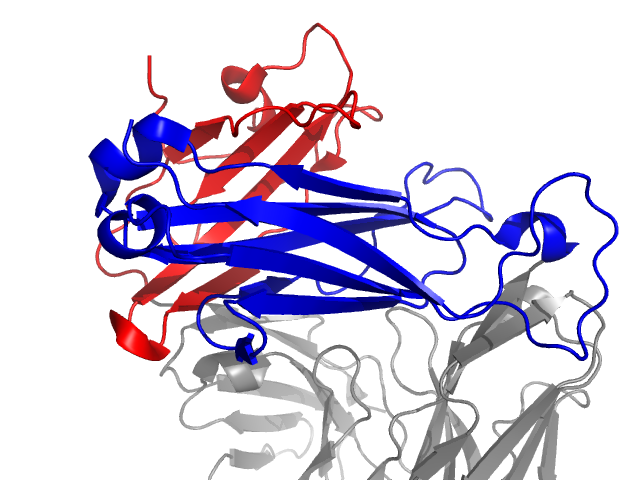
Рис. 4 Участок структуры 1KGC: α-цепь выделена красным цветом, β-цепь - синим.
Построены карты бета-листов при помощи SheeP (см. рис. 5-6).
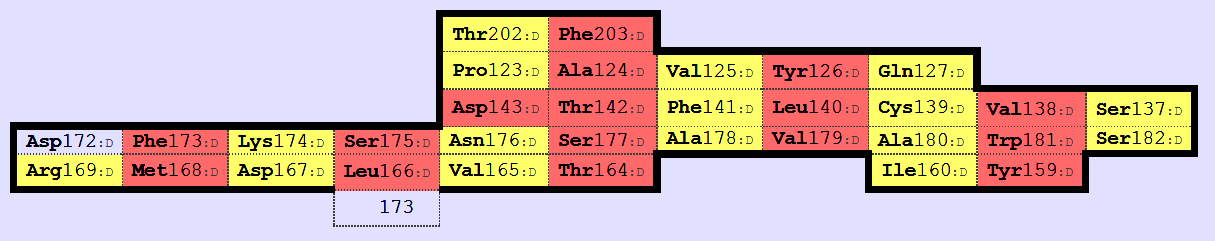
Рис. 5 Карта β-листа α-цепочки.
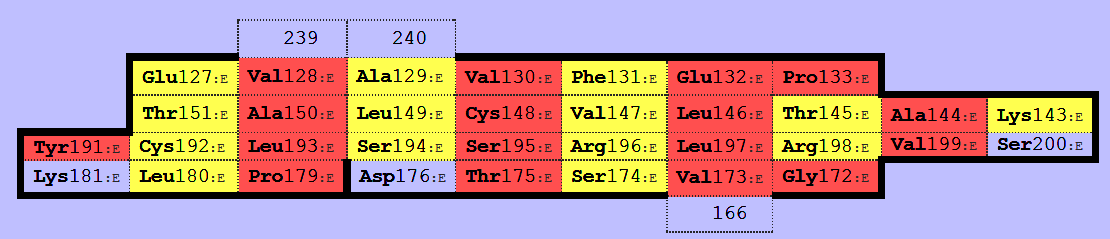
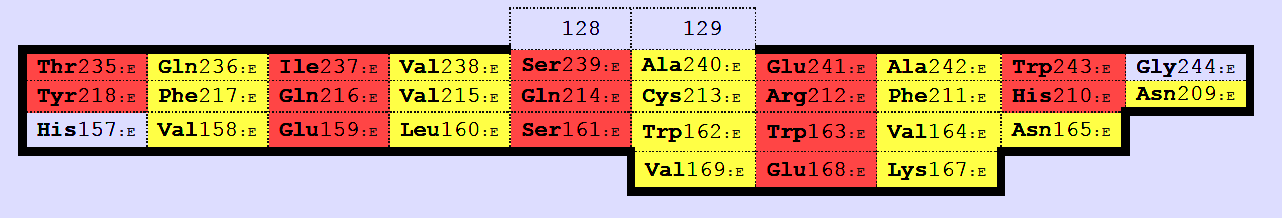
Рис. 6 Карты β-листов β-цепочки.
Первый лист в цепочке бета соответствует листу в цепочке альфа: это можно увидеть по совпадающим остаткам, соседним с их консервативными цистеинами (148 и 139, соответственно). Карты этих листов находятся в одной ориентации.
Теперь можно строить выравнивание этих листов.
Консервативные цистеины должны оказаться выровненными. Их выравнивание задает нам выравнивание всего центрального тяжа.
Для совмещения взяла по 6 остатков из каждого тяжа, так, чтобы консервативные остатки были в одинаковых позициях:
137-142 из α-цепочки, 146-151 из β-цепочки;
а также по 4 остатка из соответствующих соседних тяжей, содержащих остаток, спаренный с цистеином: 177-180 из α-цепочки и 192-195 из β-цепочки.
Для совмещения использовалась команда:
pair_fit /1kgc//d/137-142+177-180/CA, 1kgc2//e/146-151+192-195/CA
Совмещение оказалось достаточно хорошим: RMSD = 0.226 Å. Ниже приведены изображения участков совмещенных структур.
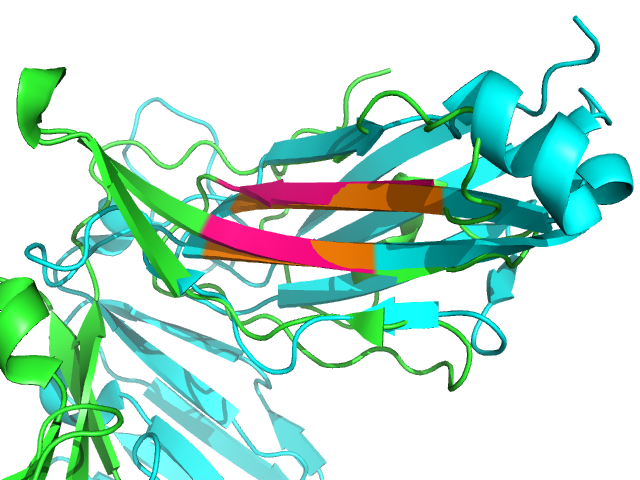 |  |
Рис. 7 Совмещение структур на участке соответствующих листов α- и β-цепочек, в виде cartoon (слева) и ribbon (справа). Розовым и оранжевым цветами выделены тяжи, по которым проводилось совмещение.
Наложение прошло хорошо, большинство тяжей совпали, наложились в том числе и маленькие. Однако петли между тяжами накладываются плохо, и топология значительно отличается, хотя две пары петель явно соответствуют друг другу, имеют очень сходное расположение в пространстве. Ссылка на сессию PyMol: superposed.pse