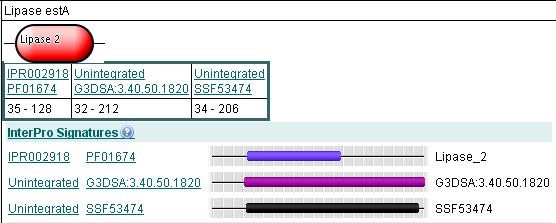
Доменная структура белка ESTA_BACSU по данным Pfam

Липазы или триацилглицерин ацилгидролазы гидролизуют эфирные связи в триацилглицерине с образованием диацилглицерина,глицерина и свободных жирных кислот.Эти ферменты катализируют следующую реакцию: Triacylglycerol + H2O = diacylglycerol + a fatty acid anion