Филогенетические деревья. Занятие 2
Задание 1 . Таксономия
Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, определила, к каким таксонам относятся отобранные мной бактерии.
| Название | Мнемоника | Таксономия |
| Bacillus subtilis | BACSU | Root; cellular organisms; Bacteria;
Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Root; cellular organisms; Bacteria;
Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | Root; cellular organisms; Bacteria;
Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Lactococcus lactis | LACLM | Root; cellular organisms; Bacteria;
Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | LISMO | Root; cellular organisms; Bacteria;
Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus epidermidis | STAES | Root; cellular organisms; Bacteria;
Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Thermoanaerobacter tengcongensis | THETN | Root; cellular organisms; Bacteria;
Firmicutes; Clostridia; Thermoanaerobacterales; Thermoanaerobacteraceae; Caldanaerobacter; Caldanaerobacter subterraneus |
На филогенетическом дереве:
Нетривиальными ветвями выделяются следующие таксоны:
класс Clostridia,порядок Clostridiales и Thermoanaerobacterales(верхняя);
класс Bacilli,порядки Lactobacillales и Bacillales (нижняя).
Задание 2
Из списка функций белков выберала одну: по белкам соответствующего семейства необходимо реконструировать филогенетическое дерево.
| Функция | Мнемоника |
| Омега-субъединица ДНК-зависимой РНК-полимеразы | RPOZ |
| Фактор элонгации трансляции Ts | EFTS |
| Рибосомный белок L2 | RL2 |
| Рибосомный белок S7 | RS7 |
| Рибосомный белок S12 | RS12 |
| Энолаза | ENO |
Выбранная функция: энолаза(ENO)
Энолаза (англ. Enolase) - фермент, участвующий в предпоследнем этапе гликолиза. Энолаза катализирует переход 2-фосфо-D-глицериновой кислоты в фосфоенолпируват. При этом от 2-фосфо-D-глицерата отщепляется одна молекула воды.
Из БД Swiss-Prot были получены последовательности белков: ENO_BACSU,ENO_CLOTE,ENO_ENTFA,ENO_LACLM,ENO_LISMO,ENO_STAES,ENO_THETN.
Задание 3,4 . Выравнивание
С помощью программы muscle было получено выравнивание полученных белков. (muscle -in eno.fasta -out eno_aligned.fasta)
Импортировала выравнивание в GeneDoc.
Цветом выделены диагностические позиции выравнивания, то есть такие, по которым можно судить о том, относится та или иная последовательность выравнивания к некоторому таксону.

Зеленый-Clostridia
Розовый-Bacilli
Задание 5. Реконструкция дерева программой fprotpars.
fprotpars -in eno_aligned.fasta -outfile eno_aligned.treefile
Программа принимает на вход выравнивание аминокислотных последовательностей.В программе fprotpars реализован метод максимальной экономии (или "бережливости", maximal parsimony).
Fprotpars выдаёт неукоренённое дерево без длин ветвей. Ответ не всегда единственен - довольно часто программа выдаёт несколько "одинаково экономных" деревьев.
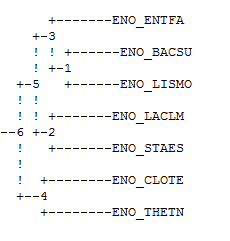
В данном случае программа выдала единственное дерево. Оно не очень совпадает с правильным.
Cохранена ветвь (CLOTE,THETN).
В правильном дереве,в отличие от построенного данной программой, есть ветви (BASCSU,LISMO,STAES),(ENTFA,LACLM).
Неукорененное также отличается тем,что содержит тривиальную ветвь ENTFA.
Правильное дерево:

Неукорененное дерево:

(((STAES,LACLM),(LISMO,((THETN,CLOTE),BACSU))),ENTFA);
Задание 6. Оценка эволюционных расстояний между последовательностями программой fprotdist.

Проверка на ультраметричность:
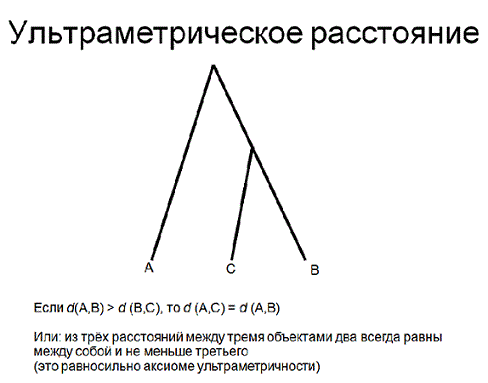
(LACLM,BACSU,LISMO):
d(LACLM,BACSU)=0,35888
d(LACLM,LISMO)=0,359046
d(BACSU,LISMO)=0,212414
d(LACLM,BACSU)=d(LACLM,LISMO), d(BACSU,LISMO)
Отклонение от ультраметроичности: 0,359046-0,358888=0,000158
Эта величина относительно расстояния:0,000158/0,212414=0,000744
Проверка на аддитивность:
Аддитивность: если есть четыре последовательности A,B,C,D, то из трёх сумм 1) d(A,B) + d(C,D) 2) d(A,C) + d(B,D) 3) d(A,D) + d(B,C)
две равны между собой и больше третьей.
Возьмем LISMO,STAES,BACSU,THETN.
1)d(LISMO,STAES)+d(BACSU,THETN)=0,342243+0,299499=0,641742
2)d(LISMO,BACSU)+d(STAES,THETN)=0,212414+0,427757=0,640171
3)d(LISM0,THETN)+d(STAES,BACSU)=0,321915+0,311973=0,633888
d(LISMO,STAES)+d(BACSU,THETN)=d(LISMO,BACSU)+d(STAES,THETN);d(LISMO,THETN)+d(STAES,BACSU)
Отклонение от аддитивности: 0,641742-0,640171=0,001571
Эта величина относительно расстояний:0,001571/0,321915=0,00488
Задание 7. Реконструкция дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining
Алгоритм Neighbor-Joining
Выдаёт неукоренённое дерево с длинами ветвей; не предполагает молекулярных часов.

Алгоритм UPGMA
fneighbor -treetype u -trout
Выдаёт укоренённое дерево с длинами ветвей. Его можно применять, если справедлива гипотеза молекулярных часов (матрица расстояний не слишком далека от ультраметрической).
(((ENO_ENTFA:0.12796,(ENO_BACSU:0.10621,ENO_LISMO:0.10621):0.02176):0.03372,
(ENO_LACLM:0.12582,ENO_STAES:0.12582):0.03587):0.03412,(ENO_CLOTE:0.14786,
ENO_THETN:0.14786):0.04794);
-У всех трех деревьев присутствует ветвь (CLOTE,THETN);
-Правильное и дерево,созданное алгоритмом UPGMA, укоренены;
-В отличие от правильного дерева, оба алгоритма строят деревья,у которых есть нетривиальная ветвь (LACLM,STAES);
-Правильное дерево и дерево UPGMA чётко отображают разделение классов Clostridia и Bacilli .
-Дерево Neighbor-Joining очень похоже на то, которое построено программой fprotpars.
-Ветвь (BACSU,LISMO) есть в правильном и в результате UPGMA деревьях.
Задание 8 . Сервис TreeTop(НИИ ФХБ им. Белозерского)

Результат такой же, как и при использовании программы fneighbor(UPGMA). Общие с правильным ветви :(CLOTE,THETN),(BACSU,LISMO).
Достаточно удобный интерфейс,выдает понятный результат(удобная матрица расстояний, дерево в разных форматах).
Главная страница
Страница четвертого семестра
© Naraykina Yulya,2011







