Алгоритмы реконструкции филогении. Занятие 3
Задание 1 . Построение дерева по нуклеотидным последовательностям
Необходимо построить филогенетическое дерево тех же бактерий, что в предыдущих заданиях, используя последовательности РНК малой субъединицы рибосомы (16S rRNA).
1)Получение последовательности 16S рибосомальной РНК каждой из бактерий:
-в записи Swiss-Prot, описывающей какой-нибудь белок, находим ссылку на полный геном бактерии;
-в записи EMBL, описывающей полный геном бактерии, ищем соответствующую "особенность" (FT), она имеет ключ (FTkey) "rRNA" и в описании: /note="16S rRNA";
-seqret embl:AL009126 16S_rRNA_bacsu.fasta -sask
Reads and writes (returns) sequences
Begin at position [start]: 30279
End at position [end]: 31832
Reverse strand [N]: N
извлекаем эти последовательности;
-кладем все последовательности в единый файл в fasta-формате, с названиями, отвечающими организмам, и выравниваем их программой muscle;
файл с выравниванием
-используя программу fdnapars, получаем дерево:
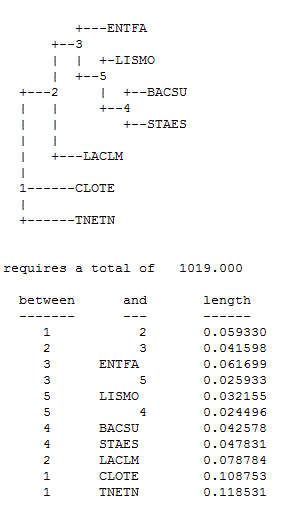
Правильное дерево:
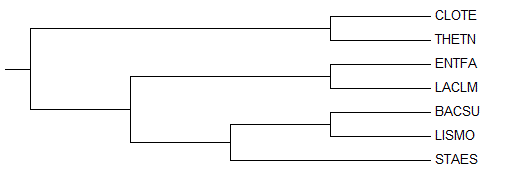
Для правильного и fdnapars деревьев характерны 2 общие ветви (LISMO,BACSU,STAES) и (CLOTE,THETN).
Построение филогенетического дерева по нуклеотидным последовательностям дает наиболее правильную реконструкцию, по сравнению с программой fprotpars.
Задание 2. Построение и анализ дерева, содержащего паралоги
Необходимо найти в своих бактериях достоверные гомологи белка CLPX_BACSU.
Чтобы найти гомологи в заданных организмах был использован файл proteo.fasta, где лежат записи банка UNIPROT, относящиеся к бактериям, перечисленным в таблице к заданию 1.
formatdb -i proteo.fasta -p T-команда для создания индексных файлов;
blastall -p blastp -d proteo.fasta -e 0.001 -i clpx_bacsu.fasta -o clpx_blastp.fasta -поиск программой BLASTP гомологов с порогом 0,001
По мнемонике видов были отобраны только те находки, которые относятся к отобранным вами бактериям:
sp|P50866|CLPX_BACSU ATP-dependent Clp protease ATP-binding subu... 770 0.0
sp|Q8Y7K9|CLPX_LISMO ATP-dependent Clp protease ATP-binding subu... 632 0.0
sp|Q8RC24|CLPX_THETN ATP-dependent Clp protease ATP-binding subu... 572 e-164
sp|Q891J8|CLPX_CLOTE ATP-dependent Clp protease ATP-binding subu... 555 e-159
sp|Q833M7|CLPX_ENTFA ATP-dependent Clp protease ATP-binding subu... 545 e-156
sp|Q8GJP6|CLPX_LACLM ATP-dependent Clp protease ATP-binding subu... 524 e-149
sp|Q834K4|HSLU_ENTFA ATP-dependent protease ATPase subunit HslU; 100 6e-22
sp|Q8Y7J8|HSLU_LISMO ATP-dependent protease ATPase subunit HslU; 96 2e-20
sp|Q8CPH0|HSLU_STAES ATP-dependent protease ATPase subunit HslU; 91 6e-19
sp|P39778|CLPY_BACSU ATP-dependent protease ATPase subunit ClpY; 90 7e-19
sp|Q8R9Y3|HSLU_THETN ATP-dependent protease ATPase subunit HslU; 90 1e-18
sp|P37571|CLPC_BACSU Negative regulator of genetic competence Cl... 49 3e-06
sp|O31673|CLPE_BACSU ATP-dependent Clp protease ATP-binding subu... 48 4e-06
sp|Q8CQ88|CLPC_STAES ATP-dependent Clp protease ATP-binding subu... 44 9e-05
seqret sw:CLPX_LISMO clpx_lismo.fasta-получены последовательности белков
Программой muscle было осуществлено выравнивание этих белков. Полученный файл был дан на вход программе fprotpars
Получившееся дерево:
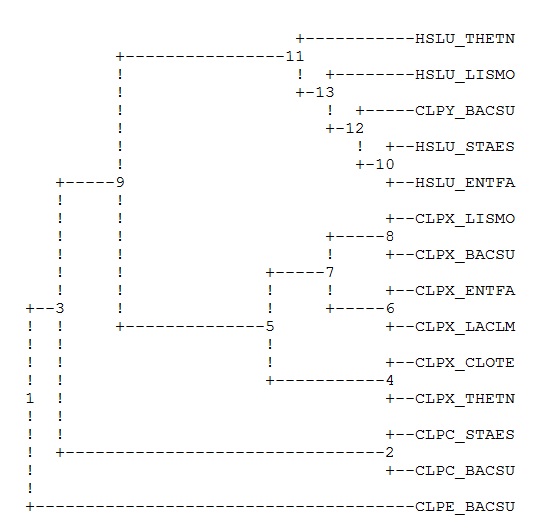
Два гомологичных белка будем называть ортологами, если они а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралагами.
Примеры пар паралогов:
- СLPX_BACSU и CLPY_BACSU;
- CLPX_BACSU и CLPC_BACSU;
- CLPX_THETN и HSLU_THETN;
- HSLU_LISMO и CLPX_LISMO
Примеры пар ортологов:
- HSLU_LISMO и HSLU_STAES
- CLPX_CLOTE и CLPX_THETN
- CLPC_BACSU и CLPC_STAES
Главная страница
Страница четвертого семестра
© Naraykina Yulya,2011


