Из представленного в задании списка выберем 8 видов:
| Название | Мнемоника |
| Rhizobium etli | RHIEC |
| Neisseria meningitidis | NEIMA |
| Erwinia carotovora | ERWCT |
| Escherichia coli | ECOLI |
| Salmonella typhimurium | SALTY |
| Yersinia pestis | YERPE |
| Vibrio fischeri | VIBFM |
| Proteus mirabilis | PROMH |
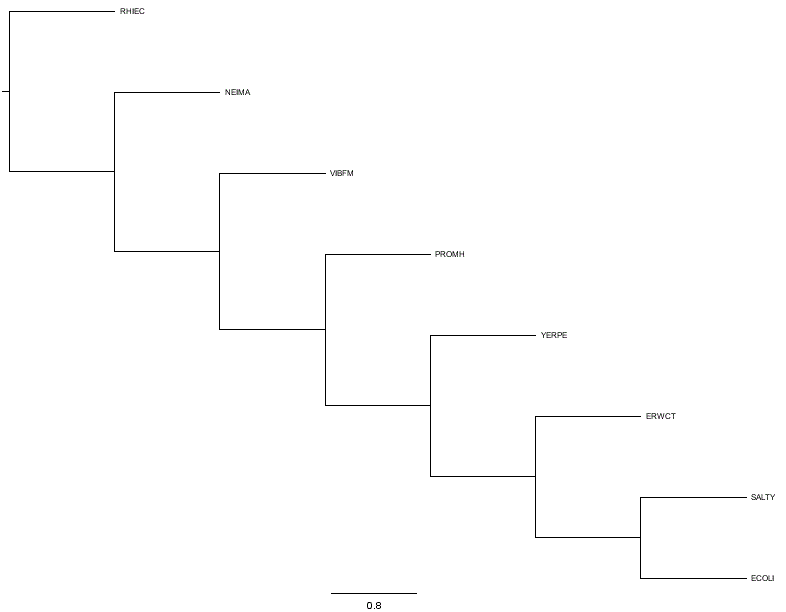
(RHIEC,(NEIMA,(VIBFM,(PROMH,(YERPE,(ERWCT,(SALTY,ECOLI)))))))

1.{RHIEC,NEIMA} и {VIBFM,PRDMH,YERPE,ERWCT,SALTY,ECOLI}
2.{RHIEC,NEIMA,VIBFM} и {PRDMH,YERPE,ERWCT,SALTY,ECOLI}
3.{RHIEC,NEIMA,VIBFM,PRDMH} и {YERPE,ERWCT,SALTY,ECOLI}
4.{RHIEC,NEIMA,VIBFM,PRDMH,YERPE} и {ERWCT,SALTY,ECOLI}
5.{RHIEC,NEIMA,VIBFM,PRDMH,YERPE,ERWCT} и {SALTY,ECOLI}
1. Rhizobium etli:
cellular organisms; Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Rhizobium2. Neisseria meningitidis:
cellular organisms; Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria3. Erwinia carotovora:
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Pectobacterium4. Escherichia coli:
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia5. Salmonella typhimurium:
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Salmonella; Salmonella enterica; Salmonella enterica subsp. enterica6. Yersinia pestis:
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia7. Vibrio fischeri:
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Aliivibrio8. Proteus mirabilis:
cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Proteus
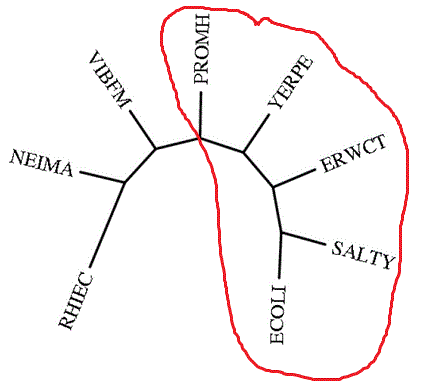
красным наиболее близкие виды.
Мною была выбрана функция Энолаза (мнемоника - ENO).
используем команду: seqret sw:ENO_XXXXX
Получим набор последовательностей белков из выбранных ранее организмов:
Rhizobium etli
Neisseria meningitidis
Erwinia carotovora
Escherichia coli
Salmonella typhimurium
Yersinia pestis
Vibrio fischeri
Proteus mirabilis
Совместные последовательности белков
Используем команду:
muscle -in eno.fasta -out eno_aligned.fasta
для анализа последовательностей программой MUSCLE.
Для работы с программой пользуемся командой :
fprotpars -sequence eno_aligned.fasta -outfile eno.fprotparsПолучили скобочную формулу данного дерева :
(((NEIMA,RHIEC),(((YERPE,ERWCT),(SALTY,ECOLI)),PROMH)),VIBFM)Так же получили одно графическое изображение данного дерева:
+--NEIMA
+--------------7
! +--RHIEC
!
! +--YERPE
+--6 +-----5
! ! ! +--ERWCT
! ! +--4
! ! ! ! +--SALTY
1 +-----2 +-----3
! ! +--ECOLI
! !
! +-----------PROMH
!
+--------------------VIBFM
Полученное дерево отличается от правильного (выданного нам). Отличие заключается в расположении бактерии ERWCT,
в полученном дереве этот вид бактерии дает кладу с YERPE.
Программа была запущена при помощи команды :
fprotdist -sequence eno_aligned.fasta -outfile rass.fprotdistБыла получена матрица расстояний :
VIBFM PROMH ECOLI SALTY ERWCT YERPE RHIEC NEIMA VIBFM 0.000000 0.152839 0.139331 0.142313 0.143594 0.124280 0.525515 0.480328 PROMH 0.152839 0.000000 0.097775 0.095445 0.106934 0.108578 0.526616 0.529689 ECOLI 0.139331 0.097775 0.000000 0.013573 0.056151 0.056078 0.479487 0.491924 SALTY 0.142313 0.095445 0.013573 0.000000 0.053870 0.061042 0.484270 0.499858 ERWCT 0.143594 0.106934 0.056151 0.053870 0.000000 0.039395 0.507924 0.500561 YERPE 0.124280 0.108578 0.056078 0.061042 0.039395 0.000000 0.497556 0.492315 RHIEC 0.525515 0.526616 0.479487 0.484270 0.507924 0.497556 0.000000 0.464549 NEIMA 0.480328 0.529689 0.491924 0.499858 0.500561 0.492315 0.464549 0.000000
Утверждение ультраметричности : "Из трех расстояний между тремя объектами два всегда равны между собой и не меньше третьего".
Рассмотрим бактерии SALTY, NEIMA, RHIEC (относятся к разным подгруппам) :
d ( SALTY, NEIMA ) = 0.499858
d ( SALTY, RHIEC ) = 0.484270
d ( NEIMA, RHIEC ) = 0.464549
Условие ультраметричности не выполняется.
Рассмотрим бактерии SALTY, ECOLI, NEIMA (есть близкие виды) :
d ( SALTY, ECOLI ) = 0.013573
d ( SALTY, NEIMA ) = 0.499858
d ( ECOLI, NEIMA ) = 0.491924
Видим, что условие ультраметричности выполняется, если считать d ( SALTY, NEIMA )= d ( ECOLI, NEIMA ) ( разница между ними 0.007934 ).
Утверждение аддитивности : если есть 4 последовательности: A, B, C, D, - то из трех сумм d ( A, B ) + d ( C, D );
d ( A, C ) + d ( B, D ); d ( A, D ) + d ( B, C ) две равны между собой и больше третьей.
Рассмотрим NEIMA, PROMH, ERWCT и ECOLI:
d ( ERWCT, NEIMA ) = 0.500561
d ( ECOLI, NEIMA ) = 0.491924
d ( PROMH, NEIMA ) = 0.529689
d ( PROMH, ERWCT ) = 0.106934
d ( PROMH, ECOLI ) = 0.095445
d ( ERWCT, ECOLI ) = 0.056151
d ( NEIMA, PROMH ) + d ( ERWCT, ECOLI ) = 0.58584
d ( NEIMA, ERWCT ) + d ( PROMH, ECOLI ) = 0.596006
d ( NEIMA, ECOLI ) + d ( PROMH, ERWCT ) = 0.598858
Видим, что две последнии суммы примерно равны между собой (разница 0.002852) и больше первой, что доказывает аддитивность.
Запускаем программы при помощи команд :
Neighbor-Joining:
fneighbor -datafile rass.fprotdist -outfile enoNJ.fneighbor -outtreefile treeNJ.tree
UPGMA:
fneighbor -datafile rass.fprotdist -outfile enoUPGMA.fneighbor -outtreefile enoUPGMA.tree -treetype u
Алгоритм UPGMA выдаёт укоренённое дерево с длинами ветвей. Его можно применять, если справедлива гипотеза молекулярных часов
(матрица расстояний не слишком далека от ультраметрической). Алгоритм Neighbor-Joining выдаёт неукоренённое дерево с длинами ветвей;
не предполагает молекулярных часов.
Результаты, полученные в результате с работой с алгоритмом Neighbor-Joining:
Получили скобочную формулу данного дерева :
((RHIEC:0.23450,NEIMA:0.23005):0.19946,(PROMH:0.06344,((ECOLI:0.00616,SALTY:0.00741):0.01834, (ERWCT:0.02122,YERPE:0.01818):0.01196):0.01036):0.02560,VIBFM:0.07119)
Так же получили одно графическое изображение данного дерева:
+-------------RHIEC
+-----------1
! +-------------NEIMA
!
! +--PROMH
! !
2-5 +ECOLI
! ! +-3
! ! ! +SALTY
! +-6
! ! +ERWCT
! +-4
! +YERPE
!
+---VIBFM
Результаты, полученные в результате с работой с алгоритмом UPGMA :
Получили скобочную формулу данного дерева :
((VIBFM:0.07024,(PROMH:0.05109,((ECOLI:0.00679,SALTY:0.00679):0.02161,(ERWCT:0.01970,YERPE:0.01970):0.00870) :0.02270):0.01914):0.18043,(RHIEC:0.23227,NEIMA:0.23227):0.01839);
Так же получили одно графическое изображение данного дерева:
+---VIBFM
!
+----------5 +--PROMH
! ! !
! +-4 +ECOLI
! ! +-1
! ! ! +SALTY
--7 +-3
! ! +ERWCT
! +-2
! +YERPE
!
! +-------------RHIEC
+-6
+-------------NEIMA
Деревья, полученные с помощью алгоритмов UPGMA и Neighbor - Joining, совпадают с деревом, полученным с помощью программы fprotpars,
но все они содержат отличия от истинного дерева. Они обьединяют в одну кладу YERPE и ERWCT.