Откроем домашнюю страничку БД PDBsum (содержит краткие схематические отображения информации о структуре).
Найдем документ с идентификатором PDB - 2ZW3. Затем подведем курсор к выравниванию последовательности в структуре,
нажмем на картинку
и получим окно с выравниванием последовательности из UniProt и последовательности из PDB.
Сохраним выравнивание в файл-fasta, затем импортируем выравнивание в Genedoc и сохраним как файл.

Нумерация в двух БД совпадает, однако последовательность из PDB оказалась короче, чем из UnipProt (можно предположить, что это N- и С- концевые домены, а также гэп в середине последовательности).
По идентификаторам CXB6_HUMAN и CXB2_HUMAN получим последовательности для выравнивания из UniProt с помощью программы seqret.
Последовательности были выравнены с помощью needle. Получили файл.

Откроем домашнюю страничку БД ОРМ,
по PDB ID найдем описание ТМ-сегментов в белке-прототипе.
Импортируем созданное в упр.1.2 выравнивание в Genedoc (needle 6.fasta 2.fasta -auto -aformat msf pp.msf). Под строчкой с выровненной
последовательностью белка-прототипа поместим строчку с разметкой трансмембранных сегментов. Сохраните все в файле.

фиолетовый цвет (символ H) - трансмембранные участки
зеленый цвет (символ +) - цитоплазматические участки
желтый цвет (символ -) - остальные
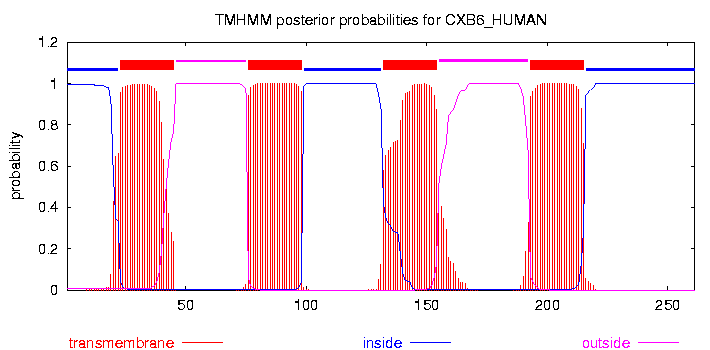
Добавим к последовательностям в файле с прошлого задания еще одну искусственную последовательность с разметкой трансмембранных спиралей в соответствии с результатами предсказания. Эту последовательность назовем "TMHMM". Получили файл.
